Corso di Genetica per Scienze per l’Ambiente e la Natura · DNA RNA PROTEINE Non tutti i geni...
Transcript of Corso di Genetica per Scienze per l’Ambiente e la Natura · DNA RNA PROTEINE Non tutti i geni...
Espressione genica: trascrizione
Corso di Geneticaper Scienze per l’Ambiente e la
Natura
Alberto Pallavicini
Schema generale
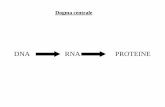
Nel 1956 Crick postulò il Dogma centrale:
DNA RNA PROTEINE
Non tutti i geni codificano per le proteine (non vengono tradotti).
• mRNA=RNA messaggero
• tRNA=RNA transfer
• rRNA=RNA ribosomale
• snRNA=piccolo RNA nucleare
La sintesi dell’RNA
Ad ogni gene sono associati delle sequenze chiamate elementi regolatori che sono coinvolti nelle regolazione dell’espressione genica.
La trascrizione è catalizzata da un enzima RNA polimerasi. Per cominciare necessità dello srotolamento della doppia elica. Nei procarioti è mediato direttamente dalla polimerasi mentre negli eucarioti è opera di u n complesso multiproteico.
La sintesi dell’RNA
La sintesi avviene in direzione 5’-3’.
L’elica che viene letta si chiama elica stampo e quella sintetizzata o quella complementare viene chiamata elica senso.
I precursori dell’RNA sono i ribonucleosidi trifosfato ATP, GTP, CTP, UTP.
La sintesi è molto simile a quella del DNA.
La sintesi dell’RNA
Le RNA polimerasi non richiedono l’utilizzo di un innesco a RNA, ma non possiedono attività di correttore di bozze.
Utilizza UTP anziché la timina, pertanto se nell’elica stampo di trova un nucleotide A verrà inserito nella catena di RNA un nucleotide U.
Inizio della trascrizione: i promotori
La trascrizione avviene in tre fasi: inizio, allungamento e terminazione.
Particolari segnali vengono utilizzati per indicare i punti di inizio e di termine.
Generalmente un gene procariote può essere diviso in tre regioni:
•Una sequenza a monte del punto di inizio della trascrizione, chiamata promotore, riconosciuta dalla polimerasi.
•La regione codificante.
•Il terminatore a valle della regione codificante.
Inizio della trascrizione: i promotori
Con l’analisi comparativa delle sequenze a monte di regioni codificanti e con lo studio degli effetti provocatio dalle alterazioni di tali sequenze sono state identificate i E. coli 2 regioni importanti per l’inizio della trascrizione.
Queste sequenze sono localizzate a –35 e –10 bp a monte di +1.
Inizio della trascrizione: i promotori
La sequenza consenso per la regione a –35 è: 5’-TTGACA-3’
La regione a –10 (Pribnow box) è: 5’-TATAAT-3’
Affinchè la trascrizione abbia inizio un complesso multipeptidico (oloenzima). L’oloenzima è composto dal core della RNA polimerasi (i peptidi 2α-β-β’) legato ad un altro fattore (σ). Questo fattore è indispensabile per il riconoscimento delle sequenze –35 e –10.
In E. coli ci sono numerosi fattori σ che giocano un fattore importante per la regolazione dell’espressione genica.
Allungamento e terminazione
Dopo che sono stati polimerizzati i primi nucleotidi il fattore σ si dissocia e può essere utilizzato in altre reazioni di inizio della trascrizione.
Il nucleo enzimatico procede nella sintesi srotolando il DNA. La tensione prodotta serve per fare riformare la doppia elica dietro la RNA polimerasi.
La trascrizione procede alla velocità di 30-35 nucleotidi al secondo.
La terminazione è segnalata da delle sequenze di terminazione.
Trascrizione negli eucarioti
Negli eucarioti (al contrario dei procarioti) tre diverse polimerasi trascrivono i 4 tipi diversi di RNA.
La RNA polimerasi I localizzata esclusivamente nel nucleolo catalizza la sintesi degli rRNA.
La RNA polimerasi II che si trova nel nucleoplasma sintetizza gli mRNA.
La RNA polimerasi III anch’essa nupleoplasmatica sintetizza gli tRNA, 5S rRNA, snRNA.
Sono state isolate le RNA polimerasi in base alla loro diversa sensibilità alla tossina α-amanitina.
La trascrizione da parte della RNA polimerasi II
Descriveremo ora gli eventi per la sintesi di mRNA codificante le proteine.
Il prodotto della RNA polimerasi II e una molecola chiamata mRNA precursore (pre-mRNA).
I promotori per i geni che codificano per le proteine sono stati analizzati in 2 modi:
•esaminare gli effetti delle mutazioni che alterano l’espressione.
•confrontare tra loro le sequenze e verificare se ci sono elementi comuni.
Si sono così identificati gli elementi promotori basali e elementi promotori prossimali.
La trascrizione da parte della RNA polimerasi II
Promotori Basali negli eucarioti:
TATA box localizzato a –25, la sequenza completa TATAAAA. Si denatura più facilmente.
CAAT box localizzato a –75, GC box localizzati a –90.
Hanno tutti un’attività generica nell’inizio della trascrizione.
L’accurato inzio della trascrizione e la sua modulazione richiede l’assemblaggio della RNA polimerasi II con alcuni fattori base della trascrizione (TF).
Il complesso che si forma è sufficiente per un basso livello trascrizionale, per un maggiore livello sono necessari altri fattori chiamati attivatori che si legano agli enhancer.
Oltre a queste regioni attivatrici abbiamo anche regioni silenziatrici (silencer) che si legano a fattori trascrizionali repressori.
Entrambi funzionano sia a valle che a monte del gene.
Gli RNA messaggeri eucarioti
La molecola di mRNA è generalmente composta in tre parti:
Sequenza leader o 5’ UTR
Sequenza codificante
La sequenza di coda o 3’ UTR
Le regioni 5’ e 3’ UTR possono contenere sequenze regolatrici.
La maturazione dell’mRNA
Gli mRNA eucariotici sono generalmente modificati sia all’estremità 5’ che all’estremità 3’.
Nel 1977 Roberts, Sharp e Berger hanno visto che nei geni di certi virus animali erano presenti sequenze non codificanti.
Negli eucarioti la maggior parte dei geni che codificano per proteine contengono introni, intramezzate alle altre sequenze presenti nell’mRNA: gli esoni.
Introne = intervening sequences
Esone = expressed sequences
Modifiche al 5’ e al 3’
Capping del 5’
Dopo la sintesi di circa 20-30 nucleotidi l’estremità 5’ viene modificata con l’aggiunta di un cappuccio (cap).
Generalmente si tratta di una 7-metil-guanosina.
Modifiche al 5’ e al 3’
Poliadenilazione del 3’
Non si è ancora trovata una sequenza di terminazione della trascrizione.
Esistono dei siti chiamati siti di poliadenilazione
posizionati a 10-30 bp a valle della sequenza consenso AAUAAA
Esiste una specifica poliA-polimerasi
Transcription termination at protein-coding genes is triggered by cleavage and polyadenylation specificity factor (CPSF), cleavage stimulatory factor (CstF), cleavage factor I (CFI) and CFII,
Gli introni
I pre-mRNA contengono spesso un certo numero di introni.
Essi devono essere excisi.
Si era già notato che nel nucleo esistevano un gran numero di molecole di RNA di diverse lunghezze chiamate hnRNA.
La prima dimostrazione avvenne solo nel 1978 con Leder.
Studiando la β-globina umana hanno visto che hnRNA corrispondente era coolineare con il gene mentre l’mRNA non lo era. Conclusero che fosse presente in introne di 800 bp.
Oggi si sa che alcuni geni presentano anche decine di introni di varie dimensioni, alcuni di poche bp fino a molte migliaia.
RNA editing
Fenomeno scoperto a metà degli anni 80 nel protozooo Tripanosoma.
Nelle piante superiori ci sono cambiamenti tra C e U per produrre codone d’inizio da un codono ACG
Nei mammiferi nell’apolipoproteinaB Formazione di stop codon tessuto specifica
Il codice genetico
I tre tipi principali di RNA operano assieme per sintetizzare le proteine durante il processo della traduzione.
La sequenza di aminoacidi sintetizzata è specificata dalla sequenza di nucleotidi nella molecola di mRNA.
Le regole che determinano questa codifica sono racchiuse nel codice genetico.
Gli aminoacidi sono 20 e i nucleotidi 4.....come facciamo?
I polipeptidi sono polimeri
Se considerate nei loro livelli fondamentali le strutture dei polipeptidi e degli acidi nucleici sono uguali. Polimeri entrambi.
Gli aminoacidi:
Gruppo R:
polare o non-polare
positivo, negativo o senza carica
Il legame peptidico
La struttura polimerica di un polipeptide viene ottenuta legando assieme una serie di AA con legami peptidici.
Condensazione tra un gruppo carbossilico e il gruppo aminico di due AA.
La struttura della proteine
La sequenza aminoacidica è la chiave della struttura e della funzione della proteina.
Ciò può venire facilmente compreso considerando la struttura secondaria nella quale sono noti alcuni aminoacidi che a causa delle loro caratteristiche chimico-fisiche , interagiscono e si legano ad altri aa.
Oppure formano strutture ad α-elica o a foglietto-β.
La prova avviene dalla denaturazione –rinaturazione.
Anche la funzione dipende strettamente dalla struttura primaria.
Es. proteine che interagiscono con il DNA.
Il codice genetico
Durante gli anni 50 i pochi biologi molecolari, riuniti attorno alla figura di Crick, cercarono di capire come l’informazione si trasmettesse tra il DNA e le proteine.
Si cominciò con il semplificare il problema usando come dogma la colinearità tra gene e proteina.
Ciascuna parola del codice è costituita da una tripletta di nucleotidi.
1 nucleotide = 4 parole del codice
2 nucleotidi = 16 parole del codice
3 nucleotidi = 64 parole del codice
La decifrazione del codice
Verso la fine degli anni 50 due progressi tecnologici permisero la decifrazione del codice:
La sintesi di molecole di RNA artificiali con la polinucleotide fosforilasi (Ochoa, 1955)
La sintesi di RNA in un sistema privo di cellule (Niremberg e Matthaei).
Analisi con omopolimeri:
Nel 1961 si scopri che l’omopolimero poli(U) codifica per la fenilalanina. In seguito analizzarono il poli(A) e poli(C).
La decifrazione del codice
Eteropolimeri a caso:
sapendo la percentuale di C rispetto alle A avevamo la probabilità che ci fosse un codone rispetto all’altro.
Comunque non tutti i codoni riuscirono ad essere identificati.
Eteropolimeri ordinati:
Khorana riusci a sintetizzare polimeri partendo da dinucleotidi per cui AC darà i due tipi di codoni ACA e CAC.
ma anche da trinucleotidi UGU darà UGU,GUU,UUG.
Aspetti del codice genetico
Il codice è degenerato:
Tutti gli aminoacidi, tranne metionina e triptofano, hanno più di un codone. I codoni con il maggior numero di siononimi sono spesso raggruppati in famiglie (GGA;GGU,GGG,GGC codificano per la glicina).
Il codice contiene i codoni per la punteggiatura:
Tre codoni UAA, UGA, UAG non codificano per un aminoacido ma se presenti nel mezzo di un eteropolimero causano l’interruzione della sintesi proteica. Sono i codoni di terminazione.
Ugualmente il codono AUG è sempre presente all’inizione di un gene e segnala l’inizio della traduzione (attenzione che codifica anche per la metionina, ed è l’unico).
Aspetti del codice genetico
Il codice genetico non è universale:
Quando nell 1966 venne completata la decifrazione del codice genetico si dette per scontato che fosse universale.
Risultava difficile pensare ad alterazione del codice compatibili con la vita.
Nel 1979 Sanger scoprì che i geni dei mitocondri umani usano un codice genetico lievemente diverso. Differenze sono state trovate anche per geni nucleari in protozoi Tetrahymena e Paramecium.
Il caso del codone UGA e la selenocisteina.
Esiste un segnale nel 3’UTR che fa in modo che il codone UGA venga riconosciuto da un tRNA con la selenocisteina.
Aspetti del codice genetico
L’anticodone vacilla:
dovrebbero esserci circa 61 diversi tRNA.
Ipotesi del vacillamentoIpotesi del vacillamento
La base 5’ dell’anticodone non è sottoposta a restrizioni dal punto di vista tridimensionale come le altre due basi
Il ruolo del tRNA nella traduzione
Ciascuna cellula contiene un certo numero di tRNA.
Ogni tRNA viene distinto dalla sua specificità per uno dei 20 AA.
Una molecola di tRNA forma un legame covalente con il suo AA e può legarsi solo al codone per quell’AA.
Più tRNA per un AA (isoaccettori).
Aminoacilazione del tRNA
Il caricamento del tRNA, il legame si forma tra il gruppo carbossilico dell’aa e il gruppo 3’OH del terminale nucleotidico del tRNA. Questo meccanismo è controllato dalle aminoacil-tRNA sintetasi.
Inizio della traduzione
Il primissimo evento nella traduzione è il legame della subunità 30S ad una molecola di mRNA.
Il sito di legame del ribosoma assicura che il punto d’inizio della traduzione avvenga in posizione esatta.
Il corretto sito di legame viene indicato come sito di legame al ribosoma. In E.coli ha come sequenza consenso:
5’-AGGAGGU-3’
Questa sequenza, nota come sequenza di Shine-Dalgarno si appaia all’RNA 16S.
Una volta legato all’mRNA la subunita 30S si muove fino a che incontra un codone AUG.
Inizio della traduzione
Formazione del complesso di inizio.
Il processo di traduzione inizia, quando un tRNA aminoacetilato si associa mediante accoppiamento di basi con un codone di inizio.
Questo tRNA iniziatore è caricato con metionina.
Nei batteri essa viene modificata con un gruppo formico (fmet).Tale sostituzione blocca l’aminogruppo direzionando in questo modo la polimerizzazione.
La molecola di mRNA, la subunità 30S e il tRNAfmet costituiscono il complesso di inizio.
Inizio della traduzione
Fattori di inizio
L’area principale della ricerca sulla traduzione, per la quale manca ancora una comprensione totale, è il ruolo svolto da alcuni fattori proteici non ribosomali.
Ad esempio l’inizio in E.coli richiede tre proteine chiamate fattori di inizio. IF1 e IF3 sembrano importanti per la dissociazione tra le subunita 30S e 50S.
IF2 partecipa al legame del tRNA iniziatore caricato.
L’elongazione della catena polipeptidica
Una volta che il complesso di inizio si sia formato, la subunità grande del ribosoma si può legare.
Richiesta dell’idrolisi di una molecola di GTP.
Il risultato è la produzione di due siti distinti e separati nei quali le molecole di tRNA si possono legare.
Il sito peptidico (P) è inizialmente occupato dal tRNAfmet.
Il sito aminoacilico (A) è posto in corrispondenza del secondo codone ed è inizialmente vuoto.
L’elongazione inizia quando la corretta molecola di tRNA entra nel sito A. Servono due fattori di elongazione EF-Tu e EF-Ts.
L’elongazione della catena polipeptidica
Formazione del legame peptidico e traslocazione.
Ora i due siti del ribosoma sono occupati da molecole di tRNA aminoacetilate e i due AA sono a diretto contatto.
Il passaggio successivo è la formazione del legame peptidico.
La reazione è catalizzata dall’enzima peptidil transferasi. Questo è un enzima complesso e misterioso forse la stessa rRNA 23S ne fa parte (ribozimi?). Agisce assieme ad un secondo enzima ribosomale la tRNA deacilasi, che spezza il legame fmet-tRNA.
A questo punto avviene una traslocazione. Il ribosoma scivola lungo l’mRNA per una distanza di tre nucleotidi il tRNA-aa entra nel sito P scalzando il tRNAfmet scarico.
L’elongazione della catena polipeptidicaCiascun mRNA può essere tradotto da vari ribosomi nello stesso tempo.
Dopo molti cicli di elongazione, l’inizio della molecola di mRNA non è piu associata con il ribosoma e un secondo ciclo di traduzione può cominciare.
Il risultato finale è un polisoma, un mRNA che viene tradotto da vari ribosomi nello stesso tempo.
Sono anche visualizzabili al microscopio elettronico.
Terminazione della catena
La terminazione avviene quando un codone di terminazione (UAA,UAG o UGA) entra nel sito A.
Non vi sono molecole di tRNA con anticodoni capaci di appaiarsi con questi codoni di terminazione; al contrario, uno dei due fattori di rilascio ( RF1 o RF2) entrano nel sito A e tagliano via il polipeptide completato dalla molecola di tRNA terminale.
Il ribosoma rilascia il polipeptide e l’mRNA e successivamente si dissocia nelle subunita 30S e 50S.
Il polipeptide si ripiega nella sua struttura terziaria ed inizia la sua vita funzionale all’interno dellla cellula.
La traduzione negli eucarioti
La traduzione negli eucarioti è sostanzialmente uguale a quella di E. coli.
La maggior differenza consiste nel legame della subunità piccola (40S) all’mRNA.
Dopo che il 40S ha contattato il CAP la subunità minore si sposta lungo il 5’ UTR fino a quando non inccontra l’AUG di inizio.
L’AUG di inizio non necessariamente è il primo che viene incontrato e quello che serve ad iniziare la traduzione è caratterizzato dall’essere immerso in un intorno di sequenze che lo caratterizzano. La sequenza con sensu di inzio è GCCA/GCCAUGG
dove A/G tre basi a monte del AUG e la G a valle dell’AUG
influenzano l’efficienza di inizio fino a 10 volte.






































































![Ragionamento Qualitativo in Genomicaprocesso, descrivendo sinteticamente anche le fasi della regolazione genica citate pocanzi. 1.1.1 DNA, RNA e Proteine Nel 1931 P.A. Levene[2] ha](https://static.fdocumenti.com/doc/165x107/5e4b083cd369322615510a24/ragionamento-qualitativo-in-genomica-processo-descrivendo-sinteticamente-anche.jpg)