· Web viewFigura 1 I batteri si difendono dalle infezioni virali grazie agli enzimi di...
Transcript of · Web viewFigura 1 I batteri si difendono dalle infezioni virali grazie agli enzimi di...

DNA ricombinante e sue applicazioni
1. La tecnica del DNA ricombinante è alla base delle moderne biotecnologie
Possiamo definire biotecnologie tutte quelle teniche che, basandosi sull’utilizzo di organismi viventi o di loro derivati, siano volte alla produzione di sostanze specifiche.
Per esempio, gli enzimi utilizzati dalle cellule per realizzare le reazioni chimiche necessarie a compiere determinate funzioni possono essere sfruttati in laboratorio per altri scopi. Allo stesso modo, le conoscenze chimiche sulla struttura degli acidi nucleici, sulle proprietà di determinati enzimi e sulle regole di complementarietà delle basi sono il punto di partenza per manipolare il DNA.
2. Come si fa a inserire nuovi geni nelle cellule?
I biologi molecolari hanno trovato in natura gli strumenti con cui riempire la loro «cassetta degli attrezzi», ma la straordinaria varietà della vita offre alle biotecnologie molto più di questo. Spesso la funzione che manca a una specie che per noi è di particolare interesse, si può trovare in altre specie. Si tratta soltanto di scoprire il modo di inserire nel genoma della specie in questione tale gene estraneo; anche in questo caso in natura troviamo gli organismi adatti: i virus.
3. Le genoteche e il DNA sintetico
Le principali fonti da cui provengono i frammenti di DNA utilizzati nelle procedure di clonazione sono tre: i frammenti casuali di cromosomi conservati in biblioteche geniche (o genoteche), il DNA complementare ottenuto per trascrizione inversa a partire da RNA e il DNA generato per sintesi artificiale o per mutazione.
4. Il sequenziamento del genoma
egli anni Settanta del secolo scorso si è sviluppato un altro filone di ricerca molto importante, finalizzato a sequenziare il DNA. Sequenziare vuol dire determinare l’ordine dei nucleotidi nella molecola del DNA. A volte il sequenziamento viene confuso con la «decifrazione» del DNA, ma in realtà esso è solo il primo passo in questa direzione, per quanto importante. Dopo avere sequenziato una molecola di DNA, infatti, i biologi molecolari devono studiarla per capire cosa significhino le sequenze identificate.
5. Le nuove frontiere delle biotecnologie
Le moderne biotecnologie trovano una vasta gamma di applicazioni in agricoltura e in zootecnia, in medicina e nella produzione di alimenti. E ancora molti altri ambiti di ricerca vengono esplorati ogni giorno.
La tecnica del DNA ricombinante è alla base delle moderne biotecnologie
Possiamo definire biotecnologie tutte quelle teniche che, basandosi sull’utilizzo di organismi viventi o di loro derivati, siano volte alla produzione di sostanze specifiche.
Per esempio, gli enzimi utilizzati dalle cellule per realizzare le reazioni chimiche necessarie a compiere determinate funzioni possono essere sfruttati in laboratorio per altri scopi. Allo stesso modo, le conoscenze chimiche sulla struttura degli acidi nucleici, sulle proprietà di determinati enzimi e sulle regole di complementarietà delle basi sono il punto di partenza per manipolare il DNA.

La tecnologia del DNA ricombinante richiede l’uso di enzimi specifici
Si definisce tecnologia del DNA ricombinante l’insieme delle tecniche di laboratorio che consentono di isolare e tagliare brevi sequenze di DNA per trasferirle e inserirle nel genoma di altre cellule, in modo da modificarne uno o più geni. Questa tecnologia permette interventi mirati, che modificano in modo specifico solo i geni dei caratteri su cui si vuole agire. Inoltre, le metodologie odierne consentono di trasferire DNA non solo tra individui della stessa specie, ma anche tra specie diverse, spesso molto differenti l’una dall’altra. Si possono, per esempio, trasferire geni da un batterio a una pianta o introdurre in un batterio un gene proveniente da una cellula eucariotica.
La tecnologia del DNA ricombinante è molto complessa dal punto di vista operativo, ma dal punto di vista concettuale si basa su criteri abbastanza semplici:
identificare il gene; tagliarlo e isolarlo dalla molecola del DNA; unire il gene a un vettore a sua volta costituito da DNA; trasferirlo all’interno di una cellula ricevente.
Gli scopi di questa operazione possono essere diversi: determinare un miglioramento genetico nell’individuo ricevente (per esempio, una maggiore resistenza agli attacchi dei parassiti), oppure utilizzare l’organismo ricevente per clonare il gene introdotto e servirsi della cellula ospite come una «fabbrica» per la produzione di molecole utili.
Il primo passaggio consiste sempre nel tagliare il DNA. Talvolta questa operazione serve solo per studiare meglio le caratteristiche dei frammenti ottenuti, ma il più delle volte è la premessa per una ricombinazione: si uniscono cioè molecole di DNA di provenienza diversa. Per tagliare e ricucire i geni sono necessari enzimi specifici (enzimi di restrizione e ligasi). La loro scoperta è stata la chiave che ha aperto la porta allo sviluppo di tutte le tecniche di manipolazione del DNA.
2
Gli enzimi di restrizione tagliano il DNA in corrispondenza di determinate sequenze
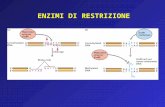
Alla fine degli anni Sessanta del Novecento si scoprì che alcuni batteri si difendono dall’attacco dei virus producendo particolari enzimi, denominati enzimi di restrizione, che tagliano le molecole di DNA estraneo riducendole in frammenti più piccoli, non infettanti ( figura 1▶ ).

Figura 1 I batteri si difendono dalle infezioni virali grazie agli enzimi di restrizione I batteri producono enzimi di restrizione che degradano il DNA dei fagi, tagliandolo in frammenti. La metilazione protegge il DNA batterico dall’azione dei propri enzimi di restrizione.
In un processo chiamato digestione da restrizione questi enzimi spezzano l’ossatura del DNA rompendo i legami tra il gruppo ossidrile all’estremità 3' di un nucleotide e il gruppo fosfato all’estremità 5' del nucleotide successivo.
Esistono numerosi enzimi di restrizione, ciascuno dei quali taglia il DNA a livello di una specifica sequenza di basi (normalmente 4-6 basi), definita sequenza di riconoscimento o sito di restrizione.
Per esempio, l’enzima *EcoRI taglia il DNA soltanto quando incontra la seguente sequenza di basi appaiate nella doppia elica del DNA:
Se osservi con attenzione questa sequenza, e la leggi in direzione 5' → 3', noterai che è la stessa su entrambi i filamenti: è un palindromo, come la frase «ai lati d’Italia».
La maggior parte degli altri enzimi di restrizione lavora riconoscendo sequenze palindrome e operando un taglio «obliquo» nel filamento di DNA. Come vedremo più avanti, questi tagli «sfalsati» sono fondamentali per ottenere DNA ricombinante.
Gli enzimi di restrizione agiscono solo sul DNA estraneo e non tagliano mai il DNA della cellula batterica che li ha prodotti. Il batterio infatti protegge il proprio DNA con un meccanismo chiamato metilazione: alcuni enzimi aggiungono un gruppo metile (–CH3) in tutti i punti del DNA batterico che potrebbero essere
riconosciuti dagli enzimi di restrizione.
Gli enzimi di restrizione possono venire isolati ed estratti dalle cellule che li producono per essere utilizzati in laboratorio come se fossero delle «forbici biochimiche». Infatti il DNA di qualsiasi organismo, se incubato in provetta con un enzima di restrizione, verrà tagliato in tutti i punti in cui è presente il relativo sito di restrizione (cioè una sequenza di basi specifica).

Oggi disponiamo di centinaia di enzimi di restrizione, purificati a partire da vari microrganismi. Perciò uno stesso campione di DNA può venire tagliato da più enzimi che riconoscono siti di restrizione diversi. In questo modo i biologi molecolari possono scegliere con precisione chirurgica dove tagliare un campione di DNA optando tra molti siti diversi. I tratti di DNA che si ottengono sono chiamati frammenti di restrizione.
Poiché le sequenze di riconoscimento non si presentano a intervalli regolari, i frammenti di restrizione hanno lunghezze diverse, ed è proprio grazie a questa variabilità che li possiamo separare. Tale operazione può servire sia a stabilire il numero dei singoli frammenti, con le rispettive dimensioni molecolari, sia a individuare e purificare un frammento di particolare interesse. Come vedremo, un sistema adatto a separare o purificare i frammenti di DNA è l’elettroforesi su gel.
*EcoRI deriva da una precisa nomenclatura degli enzimi di restrizione. E sta per il genere batterico (Escherichia), co per la specie (coli), R per il ceppo di laboratorio (in questo caso si tratta del ceppo RY13), I indica che fu il primo identificato in quel ceppo batterico.
Come si separano i frammenti di DNA
L’elettroforesi su gel separa i frammenti di DNA dopo il taglio con gli enzimi di restrizione utilizzando un gel di agarosio ( figura 2A▶ ), un polisaccaride che si ricava dalle alghe.
Il gel è posto in uno stampo di forma rettangolare; a una delle estremità del gel si trovano delle piccole cavità verticali chiamate pozzetti, allineate a formare una fila. Ogni campione, composto da una miscela di frammenti di DNA e colorato con una sostanza blu, viene deposto (o «caricato») in un pozzetto; quindi si applica al gel un campo elettrico, con il polo negativo posizionato vicino ai pozzetti e il polo positivo all’estremità opposta.
A pH neutro, il DNA è carico negativamente grazie alla presenza dei gruppi fosfato; poiché le cariche opposte si attraggono, i frammenti di DNA migrano verso il polo positivo del campo elettrico. Il gel funziona da «setaccio molecolare»: le molecole piccole, infatti migrano attraverso l’agarosio più velocemente di quelle grandi. Dopo un certo intervallo di tempo si interrompe la corrente elettrica e si esamina la distanza percorsa dai frammenti; per visualizzare il DNA si usa un colorante che diventa fluorescente quando viene esposto alla luce ultravioletta ( figura▶ 2B).
Figura 2 La «corsa» dei frammenti di DNA (A) Lo schema illustra la tecnica dell’elettroforesi su gel; (B) il risultato come appare alla luce ultravioletta.

Come puoi vedere nella figura, i frammenti si presentano come bande e possono essere esaminati o rimossi singolarmente.
L’elettroforesi su gel ci fornisce principalmente due tipi di informazione:
La dimensione dei singoli frammenti: per determinare le dimensioni dei frammenti si pone, in un pozzetto di fianco al campione, un marcatore costituito da DNA di dimensioni note, che serve come standard di riferimento.
La presenza di determinate sequenze di DNA: una sequenza nota può essere messa in evidenza all’interno del campione mediante l’uso di una sonda di DNA marcata con una sostanza radioattiva. Il campione di DNA viene denaturato (cioè despiralizzato e separato in filamenti singoli) quando è ancora nel gel e immobilizzato. In seguito, il campione viene esposto a una sonda di DNA a singolo filamento contenente una sequenza complementare a quella cercata. Se nel campione di DNA è presente la sequenza che ci interessa, la sonda si unirà ad essa. A ibridazione avvenuta, una macchia di radioattività indicherà il punto in cui la sonda ha intercettato la sequenza di DNA desiderata, mentre le sonde che non si sono legate rimarranno nella soluzione. È quindi possibile prelevare la porzione di gel corrispondente alla zona che contiene il frammento cercato (per dimensione o sequenza) e poi estrarre dal gel il frammento di DNA allo stato puro.
Un enzima in azione
L’enzima EcoRI è formato da due subunità contenenti due siti attivi identici, che tagliano contemporaneamente i due filamenti tra le basi G e A. In un tipico genoma procariotico, la sequenza di riconoscimento per EcoRI si trova in media una volta ogni 40000 coppie di basi (ossia una volta ogni quattro geni). Perciò questo enzima può tagliare un grosso pezzo di DNA in frammenti più piccoli che contengono, in media, soltanto pochi geni. L’espressione «in media» non significa che l’enzima tagli tutti i frammenti a intervalli regolari. Nelle 40000 coppie di basi del fago T7, per esempio, la sequenza di riconoscimento per EcoRI non compare neppure una volta, un fatto di vitale importanza per il virus, dato che il suo ospite è proprio E. coli.
Se impiegato in laboratorio per tagliare piccoli genomi (come quelli virali che possiedono solo decine di migliaia di coppie di basi), EcoRI produrrà pochi frammenti; invece, con i grandi cromosomi eucariotici costituiti da decine di milioni di coppie di basi, si otterranno molti frammenti.
I frammenti di restrizione forniscono un’impronta genetica
Oggi gli enzimi di restrizione si utilizzano per studiare sia il genoma dei batteri sia quello delle cellule eucariotiche. Nel caso dell’uomo, frammentando e analizzando il DNA di un enorme numero di cellule è stato possibile verificare che ciascun individuo possiede una propria «impronta genetica». Il DNA frammentato, infatti, ha caratteristiche diverse da persona a persona: esistono però delle somiglianze tra consanguinei, in particolare tra genitori e figli. Questa caratteristica viene definita polimorfismo della lunghezza dei frammenti di restrizione (RFLP, che si legge «rif-lip»)
Per ottenere la cosiddetta impronta genetica (genetic fingerprint o DNA profiling), si utilizza una procedura basata su due metodi: la digestione con enzimi di restrizione, che riduce il DNA in frammenti, e l’elettroforesi su gel, che li separa in base alla dimensione.
Tale tecnica di tipizzazione del DNA è utile per identificare le persone, stabilire rapporti di parentela e cercare geni responsabili di malattie ereditarie. Per identificare una persona o stabilire parentele sono

molto adatti i geni polimorfi, che presentano vari alleli e pertanto hanno un’alta probabilità di essere diversi nei singoli individui. I polimorfismi più usati sono principalmente due.
1. I polimorfismi da singolo nucleotide (SNP) sono variazioni che riguardano un’unica base nucleotidica. Per esempio, se in un determinato punto del genoma un genitore è omozigote per la base A e l’altro genitore lo è per la base G, la progenie sarà eterozigote (un cromosoma conterrà A e l’altro conterrà G).
2. Le ripetizioni brevi in tandem (STR) sono tratti di DNA contenenti sequenze di 2-10 basi che si ripetono più volte, una di seguito all’altra, secondo uno schema preciso. Anche questi schemi di ripetizione sono ereditari: per esempio, un soggetto può ereditare dalla madre un cromosoma 15 con una breve sequenza ripetuta sei volte e dal padre un altro cromosoma 15 con la stessa sequenza ripetuta due volte. Se una STR si trova tra due sequenze di riconoscimento per un enzima di restrizione, il DNA dell’individuo eterozigote tagliato da tale enzima darà origine a due frammenti di lunghezza diversa: uno più grande (materno) e uno più piccolo (paterno). La differenza è osservabile con l’elettroforesi su gel ( figura 3▶ ). Esaminando molte STR diverse si possono riconoscere combinazioni che caratterizzano univocamente quell’individuo.
Tuttavia, non è sempre possibile raggiungere dei risultati sicuri. La determinazione dell’impronta genetica richiede almeno 1 µg di DNA, corrispondente a quello contenuto in circa 100 000 cellule umane: una quantità non sempre disponibile. Fortunatamente, è possibile amplificare il DNA che ci interessa e ottenere così la quantità necessaria per effettuare la digestione da restrizione e l’elettroforesi. La tecnica che si utilizza per questo scopo è chiamata PCR, che analizzaremo ora.
*Impronta genetica è la traduzione quasi letterale di genetic (o DNA ) fingerprint, cioè «impronta digitale». In realtà, però, questa impronta è identificabile con una precisione di molto superiore a quella consentita dall’impronta dei polpastrelli.
Figura 3 La tipizzazione del DNA con ripetizioni brevi in tandem Il numero di STR ereditate da ogni genitore può essere usato per ottenere l’impronta genetica di un individuo. Gli alleli sono identificati in base alle dimensioni grazie all’elettroforesi su gel.
La reazione a catena della polimerasi (PCR)
Da quando è stata ottenuta la duplicazione del DNA in laboratorio, è possibile fare copie multiple di una sequenza di DNA. La reazione a catena della polimerasi (o PCR, dall’inglese Polymerase Chain Reaction) è una tecnica che automatizza questo processo copiando molte volte in provetta un tratto di DNA.
La PCR ( figura 4▶ ), è un processo ciclico fatto di tappe che si ripetono molte volte:

Figura 4 La reazione a catena della polimerasi Le tappe di questo processo ciclico vengono ripetute molte volte al fine di produrre copie multiple di un determinato frammento di DNA:
i filamenti di DNA a doppia elica, sottoposti a riscaldamento, si separano in filamenti singoli (denaturazione);
si aggiunge alla soluzione un breve primer sintetizzato artificialmente, insieme ai quattro desossiribonucleosidi trifosfati e alla DNA polimerasi;
la DNA polimerasi catalizza la produzione di nuovi filamenti complementari.
In pochi minuti, un singolo ciclo può raddoppiare la quantità di DNA presente in soluzione e lo riporta allo stato di doppio filamento. La ripetizione del ciclo per molte volte produce una crescita esponenziale del numero di copie della sequenza di DNA; per questo il processo prende il nome di amplificazione della sequenza.
La tecnica della PCR richiede la conoscenza delle sequenze di basi all’estremità 3' di ciascun filamento della sequenza bersaglio, in modo da costruire in laboratorio il primer complementare, di solito della lunghezza di 15-20 basi. Data la peculiarità di questa sequenza, nel genoma di un organismo di solito ci sarà una sola regione del DNA a cui potranno legarsi due primer di questa lunghezza. Questa specificità, a fronte dell’incredibile diversità del DNA bersaglio, è la chiave della potenza della PCR.
All’inizio esisteva un problema con la PCR: i requisiti termici. Per denaturare il DNA, infatti, bisogna scaldarlo a più di 90 °C, una temperatura che distrugge la DNA polimerasi. Se però fosse necessario aggiungere nuova polimerasi dopo ogni ciclo di denaturazione, la PCR sarebbe impraticabile.
Il problema è stato risolto dalla natura: in alcuni geyser, come anche in altre sorgenti termali, vive un batterio chiamato Thermus aquaticus. Portando avanti una ricerca di base, Thomas Brock e i suoi collaboratori scoprirono che questo organismo poteva sopravvivere a temperature fino a 95 °C grazie a un apparato metabolico termoresistente, completo di una DNA polimerasi (chiamata Taq polimerasi o polimerasi termostabile) che non si denatura ad alte temperature. In questo modo è possibile compiere più cicli di PCR.
Dopo aver letto l’articolo di Brock, il biochimico statunitense Kary Mullis pensò di usare la Taq polimerasi per la PCR; l’idea funzionò, tanto da fruttargli il premio Nobel nel 1983. Da allora, la PCR ha avuto grande importanza nella ricerca genetica e trova tuttora straordinarie applicazioni.

Le DNA ligasi ricombinano i frammenti di DNA
Tutte le cellule contengono enzimi chiamati DNA ligasi che durante il processo di duplicazione del DNA uniscono i frammenti di Okazaki (vedi capitolo B2▶ ). Quando i biologi riuscirono a isolare tali enzimi, si resero conto che potevano essere utili per saldare insieme due sequenze qualsiasi di DNA. Nel 1973, Stanley Cohen e Herbert Boyer tagliarono con enzimi di restrizione e poi unirono con una DNA ligasi due plasmidi di E. coli, ciascuno contenente un gene per la resistenza agli antibiotici. Il plasmide risultante, una volta reinserito in cellule di E. coli, le rese resistenti a entrambi gli antibiotici ( figura 5▶ ). Era appena iniziata l’era del DNA ricombinante.Figura 5 Il DNA ricombinante Cohen e Boyer realizzarono il primo esperimento in cui due diverse sequenze di DNA si combinavano in laboratorio per ottenere una nuova molecola di DNA funzionale.
Come si ottiene la ricombinazione? Sappiamo che molti enzimi di restrizione effettuano tagli sfalsati a livello di una sequenza di riconoscimento palindroma ( figura 6▶ ). In questo caso, i frammenti di restrizione terminano con due estremità a filamento singolo. Tali estremità contengono una sequenza di basi capace di legarsi a un’altra per complementarietà, perciò sono definite estremità coesive.Figura 6 Tagliare e cucire il DNA Alcuni enzimi di restrizione (è mostrato l’esempio relativo a EcoRI) praticano tagli sfalsati nelle molecole di DNA. EcoRI può essere usato per tagliare il DNA proveniente da due fonti diverse (blu e giallo). I tagli lasciano delle estremità coesive che possono ibridarsi con frammenti complementari. Le estremità coesive di DNA diversi possono legarsi le une alle altre, dando origine a DNA ricombinante.
Dal momento che contengono estremità coesive complementari, i frammenti di restrizione possono unirsi tra loro in seguito alla formazione di legami a idrogeno tra le basi, indipendentemente dalla loro origine: ciò avviene grazie alle DNA ligasi, che stabilizzano i legami tra frammenti adiacenti unendo l’estremità 5' di uno di essi con l’estremità 3' di quello vicino.
Alcune DNA ligasi sono in grado di unire (con meccanismi un po’ diversi) anche frammenti di restrizione con estremità «piatte».
Le ligasi sono molto sensibili alla temperatura (che ha molta influenza sui legami a idrogeno) e richiedono ATP per funzionare. Utilizzando questi enzimi, i biologi molecolari riescono a produrre DNA ricombinante «tagliando e cucendo» molecole di DNA provenienti dalle fonti più diverse. Per poter unire molecole di provenienza diversa è però necessario che le estremità coesive dei due frammenti siano complementari; ciò è possibile solo se entrambi sono stati tagliati dal medesimo enzima di restrizione.

Come si fa a inserire nuovi geni nelle cellule?I biologi molecolari hanno trovato in natura gli strumenti con cui riempire la loro «cassetta degli attrezzi», ma la straordinaria varietà della vita offre alle biotecnologie molto più di questo. Spesso la funzione che manca a una specie che per noi è di particolare interesse, si può trovare in altre specie. Si tratta soltanto di scoprire il modo di inserire nel genoma della specie in questione tale gene estraneo; anche in questo caso in natura troviamo gli organismi adatti: i virus.
Nel 1997 il ricercatore Ian Wilmut ottenne l’attenzione di tutta la comunità scientifica pubblicando un articolo sulla rivista Nature in cui annunciava di avere clonato una pecora a partire da una cellula adulta e non embrionale: si trattava di Dolly ( figura 7▶ ). Curiosamente l’opinione pubblica si convinse che il successo fosse la clonazione in sé, mentre per i ricercatori l’aspetto importante era un altro, cioè l’essere partiti dal nucleo di una cellula adulta e non embrionale.
Figura 7 Il famoso caso di Dolly Il primo mammifero clonato fu una pecora chiamata Dolly (a sinistra nella foto), che una volta cresciuta si è riprodotta dando alla luce un agnello «normale» (a destra).
L’esperimento era molto simile a quelli di Gurdon. L’unico accorgimento particolare di Wilmut fu lasciare per alcuni giorni in carenza di nutrimento le cellule candidate a fornire il nucleo per il trapianto, per bloccarne il ciclo cellulare. Wilmut inoltre usò come «madre in affitto» una pecora di razza diversa da quella che aveva fornito il nucleo cellulare, in modo da avere una prova della reale origine di Dolly. È da notare, tuttavia, che per Wilmut come per Gurdon la percentuale dei successi fu molto bassa: uno solo su 277 tentativi.
La clonazione permette di ottenere molte copie di un certo gene
Uno degli scopi della tecnologia del DNA ricombinante è la clonazione (cioè la produzione in molte copie) di un particolare gene allo scopo di poterlo analizzare o per poter ottenere grosse quantità del suo prodotto proteico.
In quest’ultimo caso, il DNA ricombinante deve essere inserito, o trasfettato, in cellule ospiti che in seguito a tale modifica sono chiamate transgeniche. Perché il tentativo riesca è importante la scelta della cellula ospite, che può essere procariotica oppure eucariotica.
Una volta scelta la specie ospite, il DNA ricombinante viene messo a contatto con una popolazione di cellule ospiti e, in condizioni opportune, penetra in alcune di esse. Dal momento che tutte le cellule ospiti (non solo quelle in cui è penetrato DNA ricombinante) si moltiplicano, il ricercatore deve poter stabilire quali contengono effettivamente la sequenza da clonare. Un metodo comune per riconoscere le cellule

contenenti DNA ricombinante è quello di contrassegnare la sequenza inserita con geni reporter, cioè geni di cui è facile osservare il fenotipo e che quindi servono da marcatori genetici per la sequenza di interesse.
L’inserimento di geni può avvenire in cellule procariotiche oppure eucariotiche
I primi successi ottenuti con la tecnologia del DNA ricombinante furono raggiunti usando come ospiti i batteri. Infatti, come abbiamo visto nei capitoli precedenti, le cellule batteriche sono facili da coltivare e maneggiare in laboratorio. Inoltre, la biologia molecolare dei procarioti, soprattutto di alcuni batteri ben studiati come E. coli, è ampiamente nota ed esistono numerosi marcatori genetici utilizzabili per riconoscere le cellule che ospitano il DNA ricombinante. In più, i batteri contengono plasmidi, piccoli cromosomi circolari facilmente manipolabili per trasportare il DNA ricombinante dentro la cellula.
Tuttavia, i batteri non sono gli organismi ideali per lo studio e l’espressione dei geni eucariotici. Basta pensare a quanto sono diversi i genomi e i processi di trascrizione e traduzione nei procarioti e negli eucarioti ( tabella 1▶ ), oppure al fatto che tali funzioni sono spesso regolate da segnali contenuti nel DNA stesso; i batteri, per esempio, sono privi del macchinario necessario per l’asportazione degli introni dall’RNA trascritto da un gene eucariotico. Ricorda poi che molte proteine eucariotiche, dopo il processo di traduzione, vengono chimicamente modificate, e che spesso queste modifiche sono indispensabili al corretto funzionamento della proteina.
Quando si vuole ottenere l’espressione di un nuovo gene in un eucariote, ossia quando si vuole ottenere un organismo transgenico, è meglio scegliere un ospite eucariotico, come una drosofila, un topo, una pianta o un lievito.
Gli ospiti eucariotici più usati negli studi sul DNA ricombinante sono i lieviti come Saccharomyces cerevisiae. I vantaggi di usare i lieviti comprendono la rapidità della divisione cellulare (tutto il ciclo si completa in poche ore), la facilità di coltivazione in laboratorio e le dimensioni relativamente ridotte del genoma (circa 12 milioni di coppie di basi e 6000 geni). I lieviti inoltre possiedono molte delle caratteristiche fondamentali comuni a tutti gli eucarioti, salvo quelle implicate nella condizione pluricellulare.
A volte anche i lieviti sono inadatti allo scopo: è il caso della produzione di alcune proteine umane che, dopo il processo di traduzione, devono essere chimicamente modificate per poter funzionare. Poiché i lieviti non possono apportare le modifiche necessarie, il gene viene trasferito in un genoma animale o vegetale. Quale che sia l’ospite scelto, per trasportare il DNA al suo interno è necessario un vettore.
I vettori introducono nuovo DNA nelle cellule ospiti
Quando si inserisce nuovo DNA all’interno di una cellula, la sfida non consiste tanto nel farlo entrare nella cellula ospite, quanto nel far sì che il DNA estraneo si duplichi quando la cellula si divide: infatti la DNA polimerasi, l’enzima che catalizza la duplicazione del DNA, non si lega a qualsiasi sequenza di DNA. Per potersi duplicare, il nuovo DNA deve entrare a far parte di un replicone, cioè di un segmento di DNA che contenga un’origine della duplicazione.
In linea di massima, un frammento di DNA appena inserito si può integrare in un replicone in due modi.
Dopo essere penetrato nella cellula, il frammento si può inserire in un cromosoma ospite in prossimità di un sito di origine della duplicazione. Un inserimento di questo tipo, pur essendo un evento casuale, è un metodo utilizzato spesso per integrare un nuovo gene in una cellula ospite.

Il frammento di DNA può entrare nella cellula ospite come parte integrante di una sequenza di DNA trasportatrice (un vettore) che possiede già un’origine della duplicazione adatta.
Per essere efficace, un vettore deve avere quattro caratteristiche fondamentali:
1. essere capace di duplicarsi in modo indipendente nella cellula ospite; 2. possedere una sequenza di riconoscimento per un enzima di restrizione che lo possa tagliare e
combinare con il nuovo DNA; 3. contenere un gene reporter che ne segnali la presenza nella cellula ospite; 4. avere dimensioni minori dei cromosomi dell’ospite.
I vettori che rispondono a questi requisiti sono diversi e comprendono plasmidi, virus e cromosomi artificiali.
I plasmidi ( figura 8A▶ ) hanno piccole dimensioni (un plasmide di E. coli è costituito da 2000-6000 coppie di basi, mentre il cromosoma principale ne contiene oltre 4,6 milioni) e spesso contengono un’unica sequenza di riconoscimento per un dato enzima di restrizione. Per questo un plasmide tagliato con un enzima di restrizione si trasforma in una molecola lineare provvista di estremità coesive, che si possono appaiare con le estremità coesive di un altro frammento di DNA. Altre due caratteristiche rendono i plasmidi ottimi vettori: come abbiamo già visto, molti plasmidi contengono geni che conferiscono resistenza agli antibiotici, che possono funzionare da geni reporter; inoltre, i plasmidi possiedono un’origine della duplicazione (ori) e possono duplicarsi indipendentemente dal cromosoma della cellula ospite. I plasmidi tuttavia sono ottimi vettori solo per geni di piccole dimensioni; essi infatti sono troppo piccoli per accogliere l’inserzione di frammenti molto grandi di DNA. Perciò non possono essere usati come vettori per la maggior parte dei geni eucariotici.
Figura 8 Vettori per il trasporto di DNA all’interno di cellule (A) Un plasmide contenente geni reporter per la resistenza a specifici antibiotici può essere incorporato in una cellula di E. coli. (B) Una molecola di DNA sintetizzata in laboratorio diventa un cromosoma artificiale che può trasportare in cellule di lievito il frammento di DNA inserito.
I virus vengono frequentemente usati come vettori per i geni eucariotici e procariotici. Dal momento che per un virus infettare le cellule è un fatto naturale, i virus presentano un grande vantaggio rispetto ai plasmidi: non richiedono particolari artifici per essere indotti a penetrare nelle cellule ospiti.
I cromosomi artificiali di lievito (Yeast Artificial Chromosome, YAC; figura 8B▶ ) sono cromosomi «minimi» creati in laboratorio per risolvere un problema che spesso si riscontra con i plasmidi: il DNA eucariotico utilizza un’origine della duplicazione differente da quella del DNA procariotico. Un

cromosoma minimo è una molecola di DNA che contiene non soltanto l’origine della duplicazione del lievito, ma anche le sequenze del centromero e del telomero, il che la rende un vero e proprio cromosoma eucariotico. Contiene, inoltre, siti di restrizione e geni reporter (legati a particolari esigenze nutritive del lievito) sintetizzati artificialmente. Pur comprendendo soltanto 10000 coppie di basi circa, i cromosomi artificiali possono accogliere l’inserimento di segmenti lunghi da 50000 fino a 1,5 milioni di coppie di basi. All’interno di cellule di lievito, questi cromosomi artificiali effettuano normalmente la duplicazione del DNA e l’espressione genica tipiche degli eucarioti.
Le genoteche e il DNA sintetico
Le principali fonti da cui provengono i frammenti di DNA utilizzati nelle procedure di clonazione sono tre: i frammenti casuali di cromosomi conservati in biblioteche geniche (o genoteche), il DNA complementare ottenuto per trascrizione inversa a partire da RNA e il DNA generato per sintesi artificiale o per mutazione.
Le genoteche contengono raccolte di frammenti di DNA
Una genoteca è l’insieme dei frammenti clonati corrispondenti al genoma di una cellula. Anche se ogni cellula possiede un solo genoma, le genoteche che si possono ottenere frammentando il suo DNA contengono un numero diverso di frammenti a seconda delle tecniche adottate per ottenerli. Consideriamo ora come esempio una cellula umana.
Le 23 coppie di cromosomi umani possono essere considerate una «biblioteca» che contiene l’intero genoma della nostra specie. Ogni cromosoma, un «volume» della biblioteca, contiene in media 80 milioni di coppie di basi di DNA che codificano per un migliaio di geni. Una molecola così enorme non è molto utile per studiare l’organizzazione del genoma o per isolare un particolare gene. Per questo, mediante enzimi di restrizione, i biologi frammentano i cromosomi umani in pezzi più piccoli.
Questi frammenti di DNA costituiscono ancora una genoteca ( figura 9▶ ), ma l’informazione è ora contenuta in un numero maggiore di «volumi» più piccoli, ciascuno inseribile in un vettore che potrà essere trasfettato in una cellula ospite. Quando come ospiti si utilizzano i batteri, la proliferazione di una sola cellula dà origine a una colonia di cellule ricombinanti, ciascuna contenente molte copie dello stesso frammento di DNA umano. Quando i vettori usati sono plasmidi, per costituire una biblioteca del genoma umano servono circa 200000 frammenti distinti. Se invece si usa il fago λ, un virus capace di trasportare una quantità di DNA circa quattro volte superiore rispetto a un plasmide, il numero di volumi può scendere a circa 50000.
Figura 9 La costruzione di una genoteca Il DNA di un cromosoma umano viene isolato e tagliato in piccoli frammenti mediante l’utilizzo di enzimi di restrizione. I frammenti vengono successivamente inseriti in vettori (quelli illustrati nella figura sono plasmidi) e incorporati da cellule batteriche ospiti, ciascuna delle quali riceve un singolo frammento del cromosoma umano.

L’informazione contenuta nelle colture batteriche e nelle diverse colonie che ne derivano costituisce una biblioteca genica.
Le biblioteche di cDNA vengono costruite a partire da trascritti di mRNA
Una biblioteca di DNA di dimensioni molto più ridotte, contenente soltanto i geni trascritti da un particolare tessuto, può essere ottenuta dal DNA complementare o cDNA. La prima tappa della produzione di cDNA è l’estrazione di mRNA da un tessuto. Questo mRNA funziona da stampo per l’enzima trascrittasi inversa, che sintetizza DNA a partire da RNA. Si forma così un filamento di cDNA complementare all’mRNA di partenza.
L’insieme dei cDNA ottenuti da un particolare tessuto in un determinato momento del ciclo biologico di un organismo viene definito biblioteca di cDNA ( figura 10▶ ). Gli mRNA non durano a lungo nel citoplasma e spesso sono presenti in quantità ridotte; perciò una biblioteca di cDNA è un modo per fissare nel tempo lo schema di trascrizione della cellula e darci un’immagine istantanea dell’insieme degli mRNA trascritti da un certo tipo di cellule in un dato momento (trascrittoma).
Figura 10 La sintesi di DNA complementare A partire da DNA complementare (cDNA) è possibile ottenere biblioteche geniche comprendenti soltanto i geni che vengono trascritti in un particolare tessuto e in un particolare periodo del ciclo biologico di un organismo. La sintesi di cDNA risulta particolarmente utile per l’identificazione di mRNA presente soltanto in poche copie, e costituisce spesso il punto di partenza per clonare un gene.
Le biblioteche di DNA complementare si sono rivelate di enorme utilità per confrontare l’espressione genica nei diversi tessuti a vari stadi di sviluppo. L’uso di queste biblioteche ha dimostrato, per esempio, che ben un terzo di tutti i geni di un animale si esprime soltanto durante lo sviluppo prenatale. Il DNA complementare è anche un ottimo punto di partenza per la clonazione dei geni eucariotici, soprattutto per i geni espressi a livelli bassi e solo in pochi tipi di cellule.
Due procedimenti per produrre DNA sintetico
Un frammento di DNA può essere anche prodotto artificialmente utilizzando due diverse procedure.
Sintesi chimica: quando si conosce la sequenza amminoacidica di una proteina, applicando il codice genetico possiamo ricostruire la sequenza del DNA che codifica ciascuno dei suoi amminoacidi: siamo così in grado di montare insieme i relativi tratti di DNA.
La determinazione della sequenza di basi del gene che codifica una data proteina è soltanto il primo passo per progettare un gene sintetico. È infatti necessario aggiungere altre sequenze, come le sequenze fiancheggiatrici per l’inizio, la terminazione e la regolazione della trascrizione, nonché i codoni di inizio e di stop della traduzione. Naturalmente, se si vuole che il gene sintetico venga trascritto, queste sequenze non codificanti devono essere quelle effettivamente riconosciute dalla cellula ospite.

Mutazioni indotte: le mutazioni spontanee sono state importanti per dimostrare i rapporti di causa-effetto in campo biologico, ma la tecnologia del DNA ricombinante ci permette di rispondere a domande del tipo «Che cosa succederebbe se…?» senza bisogno di scoprire mutazioni già esistenti in natura.
Dal momento che gli scienziati possono produrre DNA sintetico con qualsiasi sequenza desiderata, è anche possibile manipolarlo in modo tale da creare specifiche mutazioni indotte, le cui conseguenze si manifestano nel momento in cui tale DNA mutante si esprime in una cellula ospite.
Con queste tecniche di mutagenesi sono stati chiariti molti rapporti di causa-effetto. Per esempio, si riteneva che la sequenza segnale presente all’inizio di una proteina destinata alla secrezione fosse indispensabile per il suo passaggio attraverso la membrana del reticolo endoplasmatico.
Per dimostrare tale ipotesi è stato sintetizzato il gene per una proteina di questo tipo, ma senza i codoni della sequenza segnale. Come previsto, la proteina espressa da questo gene in cellule di lievito non attraversava la membrana del reticolo endoplasmatico, cosa che invece poteva fare un’altra proteina destinata a rimanere nel citoplasma, se al gene che la codifica si aggiungevano i codoni della sequenza segnale appropriata.
Il sequenziamento del genoma
Negli anni Settanta del secolo scorso si è sviluppato un altro filone di ricerca molto importante, finalizzato a sequenziare il DNA. Sequenziare vuol dire determinare l’ordine dei nucleotidi nella molecola del DNA. A volte il sequenziamento viene confuso con la «decifrazione» del DNA, ma in realtà esso è solo il primo passo in questa direzione, per quanto importante. Dopo avere sequenziato una molecola di DNA, infatti, i biologi molecolari devono studiarla per capire cosa significhino le sequenze identificate.
Da una sequenza genomica è possibile ricavare principalmente tre tipologie di informazioni.
1. Le finestre di lettura (ORF, Open Reading Frames), ossia i tratti codificanti dei geni. Per i geni che codificano proteine, queste regioni sono riconoscibili in base ai codoni di inizio e di stop della traduzione.
2. Le sequenze amminoacidiche delle proteine, deducibili dalle corrispondenti sequenze di DNA delle finestre di lettura, applicando le regole del codice genetico ( capitolo B3▶ ).
3. Le sequenze regolatrici, come ad esempio i promotori e i terminatori della trascrizione.
Come si determina la sequenza nucleotidica del DNA
Il sequenziamento del DNA è una tecnica importante che permette di stabilire la sequenza delle basi presenti in una molecola di DNA e che si basa sull’utilizzo di nucleosidi modificati artificialmente. Come abbiamo già visto, i desossiribonucleosidi trifosfati (dNTP) che costituiscono il normale substrato per la duplicazione del DNA contengono lo zucchero desossiribosio.
Se sostituiamo questo zucchero con il 2,3-didesossiribosio, i didesossiribonucleosidi trifosfati (ddNTP) risultanti sono aggiunti dalla DNA polimerasi a una catena in allungamento come se fossero nucelotidi normali; essendo però privi del gruppo ossidrile in posizione 3', essi non consentono l’aggiunta del nucleotide

successivo ( figura 12▶ ). Pertanto la sintesi si arresta nella posizione in cui all’estremità in crescita del filamento di DNA è stato incorporato il nucleoside modificato.
Figura 12 Confronto fra dNTP e ddNTP Le due formule chimiche evidenziano la differenza tra i nucleotidi.
Per stabilire la sequenza del DNA ( figura 13▶ ) si denatura un frammento di lunghezza inferiore alle 700 coppie di basi, in seguito il filamento singolo risultante viene messo in una provetta insieme a:
Figura 13 Le tappe del sequenziamento In questo schema vengono descritte le tappe fondamentali per sequenziare il DNA.
una DNA polimerasi per sintetizzare il filamento complementare;
alcuni brevi primer sintetizzati artificialmente;
i quattro desossiribonucleosidi trifosfati (dATP, dGTP, dCTP e dTTP);
piccole quantità dei quattro didesossiribonucleosidi trifosfati, ciascuno legato a un’«etichetta» fluorescente che emette una luce di colore diverso.
La duplicazione del DNA va avanti e ben presto nella provetta troviamo una miscela contenente i filamenti di DNA che hanno fatto da stampo e i nuovi filamenti complementari più brevi. I nuovi filamenti, che terminano ciascuno con un didesossiribonucleoside fluorescente, sono di lunghezza variabile. Infatti, ogni volta che sullo stampo compare, poniamo, una T nel filamento complementare in formazione la DNA polimerasi potrà aggiungere un dATP o un ddATP; in quest’ultimo caso però la sintesi si interrompe.
Si lascia che la duplicazione del DNA vada avanti per un certo lasso di tempo e poi si denaturano i nuovi frammenti di DNA staccandoli dai loro stampi. Quindi i frammenti vengono sottoposti a elettroforesi; mentre migrano attraverso il gel, i frammenti sono attraversati da un raggio laser che eccita i marcatori fluorescenti. La luce emessa viene intercettata e l’informazione così ottenuta (quale colore della fluorescenza ovvero quale didesossiribonucleoside si trova all’estremità del frammento) viene inserita in un computer. Il computer elabora l’informazione e produce la sequenza del frammento di DNA.
Il metodo che abbiamo descritto è il più usato attualmente e si chiama Sanger, dal nome del suo creatore, il chimico britannico che per tale scoperta (avvenuta nel 1975) vinse il premio Nobel nel 1980.

Il Progetto Genoma Umano
I primi genomi a essere sequenziati e studiati sono stati quelli di alcuni virus e batteri che, per le loro dimensioni contenute, erano più facili da trattare per i ricercatori. Fin da subito, tuttavia, fu chiaro che l’obiettivo più interessante era giungere a sequenziare il genoma umano; questa impresa prometteva infatti le più importanti ricadute pratiche, soprattutto nel campo della medicina.
Il Progetto Genoma Umano (o HGP dall’inglese Human Genome Project) è stato uno dei più grandi progetti di ricerca in campo biologico dell’intero Novecento. Avviato all’inizio degli anni Novanta su iniziativa di James Watson, uno dei due padri della doppia elica, il progetto è stato completato ufficialmente nel 2003, anche se una prima bozza era stata pubblicata con grande clamore nel 2001.
Il progetto ha visto la collaborazione di un ente pubblico statunitense, i National Institutes of Health (NIH), e di un’azienda privata, la Celera Corporation fondata e diretta dal biochimico Craig Venter. A questi due gruppi si sono aggiunte collaborazioni di altri enti in USA, Canada, Gran Bretagna e Nuova Zelanda.
L’obiettivo del progetto sarebbe apparso inimmaginabile solo pochi anni prima; la sequenza completa del corredo cromosomico aploide di un essere umano, infatti, comprende oltre 3 miliardi di nucleotidi. Poiché ogni genoma è unico per alcuni suoi tratti, il Progetto ha previsto la comparazione di diversi campioni con un’elaborazione statistica dei dati.
I NIH hanno usato un approccio tradizionale, frammentando il genoma con diversi enzimi di restrizione, così da ottenere frammenti di piccole dimensioni, che poi sono stati collegati tra loro per ricavare frammenti di dimensioni via via maggiori. La Celera ha fatto invece ricorso a una tecnica chiamata shotgun sequencing che, sfruttando potenti computer, è in grado di trovare i collegamenti tra più frammenti, accelerando molto le operazioni, anche se con minore precisione. Attualmente l’intera sequenza del genoma umano è scaricabile da internet, dove è conservata presso siti specifici, consultabili dai ricercatori.
L’esame delle sequenze ha rivelato alcuni dati di estremo interesse. Il più sorprendente è che il nostro genoma contiene dai 28000 ai 30000 geni, contro i circa 100000 che i genetisti ritenevano plausibili in precedenza. I geni sono stati individuati attraverso un algoritmo informatico di ricerca degli ORF.
A fronte di questo scarso numero di geni, risulta che circa il 95% del DNA umano è costituito da sequenze che non vengono mai tradotte in polipeptidi. Per questo tipo di DNA è stato coniato il nome poco lusinghiero di junk DNA («DNA spazzatura»). I ricercatori, tuttavia, stanno iniziando solo ora a chiarire la vera natura di questa componente, che appare molto eterogenea e comprende anche tratti di DNA che svolgono diverse funzioni, alcune delle quali potrebbero rivelarsi importanti; vediamone qualcuna.
I telomeri e i centromeri sono indispensabili per la corretta conservazione dei cromosomi. Gli introni, attraverso il meccanismo di splicing e in particolare attraverso lo splicing alternativo,
garantiscono al DNA eucariotico una versatilità sconosciuta al genoma dei batteri. Alcune sequenze sono coinvolte nella regolazione dell’espressione genica, direttamente o attraverso
speciali RNA, come nel caso del meccanismo chiamato interferenza dell’RNA. Più dubbio è il giudizio sugli pseudogeni, antichi geni che hanno perso funzionalità in seguito a
mutazioni. Molti biologi ritengono che la loro presenza possa favorire, per mutazione, la comparsa di nuovi geni in una specie. Vi sono inoltre rari casi di pseudogeni che hanno riacquistato la funzione perduta attraverso una successiva mutazione. Il numero degli pseudogeni presenti nel nostro

genoma non è noto con precisione, ma si valuta che sia attorno a diverse migliaia, pari forse al 10% del DNA totale.
Un caso a sé è quello dei trasposoni, elementi in grado di moltiplicarsi e spostarsi nel DNA che da soli costituiscono fino al 50% del genoma umano. A tutt’oggi nessuna funzione utile è stata scoperta per questi elementi, che, anzi, possono essere coinvolti nell’insorgenza di tumori.
L’ultima componente del junk DNA è costituita dalle sequenze ripetute. Molti studiosi ritengono che tali sequenze siano importanti per dare compattezza al DNA, e recenti ricerche hanno anche fatto emergere un loro ruolo nel processo di regolazione della trascrizione.
A pochi anni di distanza, è già difficile dare l’idea della grandiosità dell’HGP, dato che i vertiginosi progressi tecnologici hanno fatto sì che un nuovo genoma possa essere sequenziato in pochi mesi. Resta il fatto che questo campo di ricerche (denominato genomica) è stato aperto proprio dalle esperienze fatte grazie all’HGP.
La genomica
La possibilità di studiare i genomi segna l’inizio di quella branca della biologia molecolare che definiamo genomica e che studia la struttura del genoma, le informazioni in esso contenute, il modo in cui le sue diverse parti interagiscono e la sua evoluzione. Si può parlare di genomica funzionale e di genomica comparata.
1. La genomica funzionale consiste nell’assegnazione di ruoli funzionali ai prodotti dei geni individuati con il sequenziamento ( figura 14▶ ). Sequenziando un intero genoma si trovano, infatti, molte sequenze geniche sconosciute e non associabili ad alcuna funzione. Tale funzione può spesso essere ricavata da ricerche successive, comparando, per esempio, la struttura del gene scoperto a quella di altri geni. Il sequenziamento può quindi condurre all’aumento delle conoscenze sulle funzioni delle cellule.
Figura 14 L’organizzazione funzionale
del genoma L’intera sequenza del DNA del batterio Haemophilus influenzae è costituita da 1830137 paia di basi.

2. Nella genomica comparata si confrontano le sequenze genomiche di organismi diversi per individuare i geni presenti in un organismo e assenti in un altro, al fine di mettere in relazione questi risultati con la rispettiva fisiologia. La genomica comparata fornisce anche un solido sostegno alla teoria dell'evoluzione.
In questi anni, lo studio della genomica si è rivelato di grande importanza per le sue applicazioni in campo medico-sanitario, ma anche, per esempio, alimentare o ecologico. Un caso particolarmente affascinante è quello della produzione di un organismo artificiale, dotato di un genoma costruito interamente in laboratorio. Questa impresa, alla quale si è dedicato Craig Venter dopo avere completato il Progetto Genoma Umano, non è solo suggestiva, ma interessante come primo passo verso la produzione di specie batteriche costruite secondo specifiche necessità. L’organismo, chiamato Mycoplasma laboratorium, costituirebbe la base per la produzione di tutti i futuri batteri artificiali, poiché è stato dotato del genoma minimo indispensabile per una cellula.
Le nuove frontiere delle biotecnologie
Le moderne biotecnologie trovano una vasta gamma di applicazioni in agricoltura e in zootecnia, in medicina e nella produzione di alimenti. E ancora molti altri ambiti di ricerca vengono esplorati ogni giorno.
Le cellule come fabbriche di farmaci
Se un gene eucariotico viene inserito in un plasmide e poi trasfettato in E. coli, il relativo prodotto genico sarà espresso in quantità minima o nulla dalla cellula procariotica, a meno che non si aggiungano altre sequenze chiave. Infatti, perché il gene si esprima e il suo prodotto venga sintetizzato dalla cellula batterica non possono mancare il promotore che si lega specificamente con l’RNA polimerasi batterica, il terminatore della trascrizione e una particolare sequenza dell’mRNA che gli permette di legarsi al ribosoma.
Per risolvere questo tipo di problema, i biologi costruiscono vettori di espressione che possiedono tutte le caratteristiche dei vettori tipici con in più le sequenze necessarie affinché un gene estraneo (detto anche transgene) si esprima nella cellula ospite ( figura 15▶ ).
Figura 15 Un vettore di espressione permette a un gene
estraneo (transgene) di essere espresso in una cellula ospite Un gene eucariotico trasformato può non essere espresso in E. coli perché manca delle sequenze necessarie al batterio per l’inizio e la terminazione della trascrizione e per il legame ai ribosomi. I vettori di espressione contengono queste sequenze addizionali, permettendo la sintesi di una proteina eucariotica da parte di una cellula procariotica.
Una volta inserite queste sequenze nella sede appropriata del vettore, un gene trasfettato da tale vettore si esprimerà praticamente in ogni tipo di cellula ospite.
Grazie alle biotecnologie si sintetizzano molti prodotti utili in campo medico, tra i quali vaccini, ormoni proteici e

farmaci per trattare l’infarto e l’ictus. Un altro modo per ottenere elevate quantità di prodotti utili alla medicina è il cosiddetto pharming, la produzione di farmaci proteici prodotti da animali transgenici. Un’applicazione prevede, per esempio, lo sfruttamento di vacche transgeniche nel cui latte si accumulano specifici farmaci antitumorali.
Le biotecnologie modificano l’agricoltura
La moderna tecnologia del DNA ricombinante consente di intervenire in agricoltura agendo direttamente su specifici geni, invece di ricorrere alle tradizionali tecniche di incrocio e selezione. Questo approccio consente tempi molto più rapidi e risultati più controllabili; alcune delle applicazioni più diffuse delle biotecnologie riguardano l’agricoltura.
La produzione di specie resistenti ai parassiti e alle infezioni virali, che possono essere coltivate riducendo drasticamente l’uso di insetticidi e di erbicidi. Il primo esempio in questo senso è stato l’uso di una tossina letale per gli insetti, prodotta dal batterio Bacillus thuringiensis.
La manipolazione per ottenere cereali con caratteristiche nutrizionali potenziate o in grado di produrre farmaci utili.
Un discusso ma interessante risultato in questo campo è stato la produzione del cosiddetto golden rice (ovvero il riso dorato; figura 16▶ ), un riso geneticamente modificato per arricchirlo di β-carotene, il precursore della vitamina A. Tale vitamina è assente nel riso e la sua carenza può causare gravi malattie e anche la morte. Nel golden rice è stato inserito un gene del narciso che consente la sintesi del β-carotene. Questo cereale è attualmente al centro di un dibattito tra ricercatori, intellettuali e ambientalisti sull’opportunità del suo utilizzo per l’alimentazione umana.
Le biotecnologie sono sicure per l’uomo e per l’ambiente?
L’uso delle biotecnologie, soprattutto applicate all’agricoltura, ha suscitato fin da subito perplessità e opposizioni, le cui ragioni sono economico-politiche, filosofiche, ma anche più propriamente scientifiche. Tra queste ultime, si possono segnalare:
la possibilità di immissione di allergeni nella catena alimentare; la possibilità di aumento della resistenza di patogeni naturali a determinati antibiotici o altre
sostanze; le conseguenze su specie diverse da quelle desiderate; la diminuzione della biodiversità.
Riguardo agli allergeni, chi si oppone agli OGM sostiene che inserire geni estranei in un organismo potrebbe causare reazioni allergiche legate alla specie da cui provengono i geni estranei inseriti nel genoma ospite.
Riguardo all’induzione di effetti indesiderati di resistenza, sappiamo che alcune piante GM permettono di ridurre l'uso dei diserbanti grazie all’inserimento nel loro genoma di un gene che conferisce resistenza ai pesticidi; se tale gene si trasmettesse da una pianta coltivata a un’erbaccia che cresce nelle vicinanze, si genererebbe un’infestante capace di prosperare anche nelle aree trattate con diserbanti. Il problema non è soltanto teorico, dato che il «trasferimento laterale» di geni è un meccanismo ben noto.
È stato fatto presente un altro problema sempre legato alla coltivazione degli OGM: sappiamo che alcune piante GM sono resistenti ai parassiti grazie a un gene ricavato dal batterio Bacillus thuringensis, che

consente di produrre una tossina letale per gli insetti ma innocua per gli esseri umani. Tali tossine, tuttavia, potrebbero causare gravi danni anche alle popolazioni di insetti utili, come gli impollinatori.
In merito alla biodiversità, la questione è che l’uso di coltivazioni standardizzate, con poche specie ultraselezionate e uguali in tutto il mondo (cosa che peraltro già si verifica con le sementi selezionate) è una condizione di per sé instabile, che espone a rischi potenzialmente gravi.
Per placare tali dubbi ci sono diverse possibilità. La scelta più radicale è la messa al bando degli OGM, mentre un approccio meno drastico potrebbe prevedere:
l’utilizzo sotto stretto controllo, con aree delimitate per la coltivazione di OGM: una serie di precise norme sull’etichettatura; l’incentivazione di biotecnologie sicure e rispettose dell’ambiente.
A proposito di quest’ultimo punto, l’Unione Europea sovvenziona da anni programmi di ricerca che studiano la possibilità di un utilizzo «dolce» delle biotecnologie.