Scuola di Specializzazione in Fisica Sanitaria Università...
Transcript of Scuola di Specializzazione in Fisica Sanitaria Università...
Scuola di Specializzazionein Fisica SanitariaUniversità degli Studi di Milano
Elementi di teoria dell‘analisi compartimentale
Augusto Giussani
Helmholtz Zentrum MünchenInstitute of Radiation Protection
Milano, 17/03/2008
Helmholtz Zentrum MünchenInstitute of Radiation ProtectionWorking Group Internal Dosimetry and BiokineticsIngolstädter Landstr. 185764 Neuherberg
Augusto Giussani
Helmholtz Zentrum MünchenInstitute of Radiation Protection
Milano, 17/03/2008
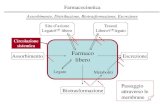
Rateo di dose equivalente all’organo bersaglio T:
qSj = Attività del radionuclide j nell’organo sorgente S
SEE(T S;t) = specific effective energy:
Dose equivalente impegnata (fino all’età di 70 anni)*:
* (nell’ipotesi che tutte le grandezze siano indipendenti dal tempo)
Dose efficace impegnata:
∑∑ ←=S j
jj,S0T )t;ST(SEE)t(qc)t,t(H&
∑←
=i T
iiii,R
)t(M)t;ST(AFEYw
( ) ( )∑∑∫ ←==S j
jj,S
70
t0T0T STSEEUcdt)t,t(HtH
0
&
∑=
+=12
1TremremTT HwHwE ∑ ⎥⎦
⎤⎢⎣
⎡ +=
T
FT
MT
T 2HHwE
∑∑ ←=S j
jj,S0T )t;ST(SEE)t(qc)t,t(H&
( ) ∑ ←⋅=h
hkh0
k )rr(SA
rD τ ICRP Publication 53
Radiation dose from radiopharmaceuticals (1988)
( ) ( ) ( ) ( ) n,,2,1i0QRQRQRRtQn
ij1j
ii0
n
ij1j
ijijij0ii KK& ==−−+= ∑ ∑≠=
≠=
Statostazionario
Un sistema viene descritto come una seriedi unità (compartmenti) all‘interno delle quali le quantità di interesse (variabili di stato) si comportano in maniera omogeneaed uniforme.
La descrizione matematica di un modellocompartimentale è data da un sistema di equazioni differenziali:
Modello compartimentale
ICRP Publication 69: Age dependent doses to members of the public from intake of radionuclides: part 3. Ingestion dose coefficients. Annals of the ICRP, Vol. 25/1, 1995.
CASO 1
186186ReRe--HEDP HEDP nellanella terapiaterapia palliativapalliativa del del doloredolore da da metastasimetastasi osseeossee
In collaborazione con Ospedali Riuniti di Bergamo.
Somministrato a pazienti che non rispondono alla terapia standard di palliazione del dolore.
Protocollo individualizzato.
Possibili modalità alternative di somministrazione (frazionamento, infusione).
7 pazienti (età 58-72) con pregresso tumore alla prostata e metastasiossee dolenti.
Misure di attività in sangue intero, plasma, siero e urine.
Determinazione di attività in alcuni tessuti (osso metastatico, osso sano, rene, vescica) con una gamma-camera ELSCINT Helix (+ immagini S-CT per calibrazione dell‘attività in funzione della profondità).
A.Giussani et al., Workshop on Internal Dosimetry of Radionuclides, Oxford, Sept. 2002
Post-administration time (h)0 10 20 30 40 50
Act
ivity
conce
ntr
atio
n (
MBq m
l-1)
0.00
0.05
0.10
0.15
0.20
0.25
0.30
Whole bloodBlood plasma
Post-administration time (h)0 10 20 30 40 50 60 70 80 90 100
Perc
enta
ge
cum
ula
ted e
xcre
tion
0%
20%
40%
60%
80%
CASO 1: 186Re-HEDPCASO 1: CASO 1: 186186ReRe--HEDPHEDP
CurveCurve di di attivitattivitàà in in sanguesangue eded urineurine
Post-administration time (h)
0.0 0.5 1.0 1.5 2.0 2.5
Act
ivity
conce
ntr
atio
n in p
lasm
a (M
Bq m
l-1)
0.01
0.1
Bound fractionUnbound fraction
A.Giussani et al., Workshop on Internal Dosimetry of Radionuclides, Oxford, Sept. 2002
CASO 1: 186Re-HEDPCASO 1: CASO 1: 186186ReRe--HEDPHEDP
Frazione slegata
q11Other organs
ModelloModello propostoproposto per la per la biocineticabiocinetica di di 186186Re Re tenendotenendo in in considerazioneconsiderazione la la dissociazionedissociazionedelladella molecolamolecola
CASO 1: 186Re-HEDPCASO 1: CASO 1: 186186ReRe--HEDPHEDP
A.Giussani et al., Workshop on Internal Dosimetry of Radionuclides, Oxford, Sept. 2002
CASO 2
TerapiaTerapia con con radioiodioradioiodio del del nodulonodulo autonomoautonomo
In collaborazione con Ospedale Maggiore Policlinico di Milano.Una parte della tiroide (nodulo) è iperattiva, e lavora in maniera autonoma
rispetto al resto della ghiandola.21 pazienti (6 M e 15 F, età 63 a [45-85]) con un singolo nodulo autonomo, con
TSH soppresso e valori di FT3 e FT4 normali o elevati.Studio dosimetrico effettuato con 123I. Uptake e clearance nel nodulo e nel tessuto
extranodulare determinato da imaggine del collo prese a 2, 4, 24, 48, 72 e 120 ore. Gamma camera Prism 2000XP (Philips) data di un collimatore a foriparalleli Low-Energy-High-Resolution.
Trattamento terapeutico personalizzato con 131I. Cinetica dello 131I studiata a 2, 4, 24, 48, 96 ore e, per la maggior parte dei pazienti, ad intervalli successivi finoad un massimo di 46 giorni. Stessa gamma camera dotata di un collimatoreHigh-Energy-General-Purpose.
Escrezione nelle urine monitorata in 3 pazienti durante gli studi dosimetrici e in 6 pazienti dopo la terapia. L’attività nei campioni di urine è stata determinatausando rivelatori NaI calibrati (geometria a pozzetto).
ModelloModello di di partenzapartenza
CASO 2: 131ICASO 2: CASO 2: 131131II
A.Giussani et al., EANM Annual Congress, Amsterdam, Aug. 2003
A.Giussani et al., Workshop on InternalDosimetry of Radionuclides, Montpellier, Oct. 2006
Time after administration (h)
0 200 400 600 800 1000
Nodule
upta
ke
0%
4%
8%
12%
16%
20%UptakeUptake dellodello iodioiodio nelnel nodulonodulo((tempitempi brevi)brevi)
CASO 2: 131ICASO 2: CASO 2: 131131II
A.Giussani et al., EANM Annual Congress, Amsterdam, Aug. 2003
A.Giussani et al., Workshop on InternalDosimetry of Radionuclides, Montpellier, Oct. 2006
Time after administration (h)
0 200 400 600 800 1000
Nodule
upta
ke
0%
4%
8%
12%
16%
20%UptakeUptake dellodello iodioiodio nelnel nodulonodulo((tempitempi brevi).brevi).
⎯⎯ modellomodello di di partenzapartenza
CASO 2: 131ICASO 2: CASO 2: 131131II
A.Giussani et al., EANM Annual Congress, Amsterdam, Aug. 2003
A.Giussani et al., Workshop on InternalDosimetry of Radionuclides, Montpellier, Oct. 2006
Time after administration (h)
0 200 400 600 800 1000
Nodule
upta
ke
0%
4%
8%
12%
16%
20%UptakeUptake dellodello iodioiodio nelnel nodulonodulo((tuttitutti i i datidati).).
⎯⎯ modellomodello di di partenzapartenza ((tempitempibrevi)brevi)
A.Giussani et al., EANM Annual Congress, Amsterdam, Aug. 2003
A.Giussani et al., Workshop on InternalDosimetry of Radionuclides, Montpellier, Oct. 2006
CASO 2: 131ICASO 2: CASO 2: 131131II
Time after administration (h)
0 200 400 600 800 1000
Nodule
upta
ke
0%
4%
8%
12%
16%
20%UptakeUptake dellodello iodioiodio nelnel nodulonodulo..
⎯⎯ modellomodello di di partenzapartenza ((tempitempibrevi)brevi)
⎯⎯ modellomodello di di partenzapartenza ((tuttitutti i i datidati))
A.Giussani et al., EANM Annual Congress, Amsterdam, Aug. 2003
A.Giussani et al., Workshop on InternalDosimetry of Radionuclides, Montpellier, Oct. 2006
CASO 2: 131ICASO 2: CASO 2: 131131II
ModelloModello modificatomodificato
A.Giussani et al., EANM Annual Congress, Amsterdam, Aug. 2003
A.Giussani et al., Workshop on InternalDosimetry of Radionuclides, Montpellier, Oct. 2006
CASO 2: 131ICASO 2: CASO 2: 131131II
Time after administration (h)
0 200 400 600 800 1000
Nodule
upta
ke
0%
4%
8%
12%
16%
20%UptakeUptake dellodello iodioiodio nelnel nodulonodulo..
⎯⎯ modellomodello di di partenzapartenza ((tempitempibrevi)brevi)
⎯⎯ modellomodello di di partenzapartenza ((tuttitutti i i datidati))
⎯⎯ modellomodello modificatomodificato..
A.Giussani et al., EANM Annual Congress, Amsterdam, Aug. 2003
A.Giussani et al., Workshop on InternalDosimetry of Radionuclides, Montpellier, Oct. 2006
CASO 2: 131ICASO 2: CASO 2: 131131II
Patient
7 9 24 35 36 38 sinis 38 dest 39 40
Del
ta [%
]
0
5
10
15
20
100 Simple170 Simple270-430 Simple
A.Giussani et al., EANM Annual Congress, Amsterdam, Aug. 2003
A.Giussani et al., Workshop on InternalDosimetry of Radionuclides, Montpellier, Oct. 2006
CASO 2: 131ICASO 2: CASO 2: 131131II
SISTEMA IN STATO STAZIONARIO COSTANTE E SENZA INPUT DALL’ESTERNO
Ipotesi: ho un solo compartimento accessibile, tutto il resto lo vedo come un unico insieme
Pool non Pool non accessibileaccessibile
Rate of appearance: Ra(t) = R10(t)+E1(t)Rate of disappearance:
( ) ( ) ( ) ( ) n,,2,1i0QRQRQRRtQn
ij1j
ii0
n
ij1j
ijijij0ii KK& ==−−+= ∑ ∑≠=
≠=
( ) ( ) ( )∑ ∑≠= =
−=n
1j0j
n
2jj11j tRtRtRd
SISTEMA IN STATO STAZIONARIO COSTANTE E SENZA INPUT DALL’ESTERNO
Ra=RdSe si vuole conoscere Ra, bisogna trovare il collegamento tra Ra e la variabile accessibile alla misura (es. concentrazione della sostanza). Questo collegamento è dato dalla relazione:
h(t) è il “kernel” (nocciolo) del sistema: dà la risposta del sistema ad uno stimolo (che è Ra). Nel caso stazionario, Ra e C sono costanti, e la relazione precedente può essere espressa come
( ) ( )∫ ττ⋅τ=t
0
d)(Ra,thtC
( ) ττ=
∫∞
0dh
CRa
DETERMINAZIONE DELLA FUNZIONE h(t) UTILIZZO DEL TRACCIANTE
Condizioni che deve soddisfare il tracciante:1) Comportarsi come la sostanza da tracciare (tracee)
2) Quantità trascurabile rispetto al tracee
3) Discriminabile dal tracee per mezzo di opportune tecniche analitiche di misura (traccianti radioattivi, traccianti stabili)
Quindi: si somministra un tracciante, se ne determina la concentrazione nel compartimento accessibile (funzione risposta), si ricava h. SOMMINISTRAZIONE IN BOLO
INFUSIONE A TASSO COSTANTE
“PRIMED INFUSION”
SOMMINISTRAZIONE IN BOLO
( ) ∑=
α−=k
1i
ti
ieAdtc
0 50 100 150 2000
5
10
15
20
25
∑= α
= k
1i i
iACRa
d/)t(c)t(h =
0 50 100 150 2000
5
10
15
20
25
“PRIMED INFUSION”
( ) ( ) ∑∑∑=
α−
=
α−
=
α−⎟⎟⎠
⎞⎜⎜⎝
⎛⎟⎟⎠
⎞⎜⎜⎝
⎛α
−⋅+α
=−α
+=k
1i i
ii
t
i
ik
1i
t
i
ik
1i
ti
ArAdeAre1AreAdtc iii
0ArAdk
kk =
α−⋅
kdr
α=
cCrRa =
Equazioni della cinetica del tracciante
Siccome nello stato stazionario Rijs e Qjs sono costanti, allora l’equazione che regola la cinetica del tracciante è un’equazionelineare, QUALE CHE SIA LA DINAMICA DEL TRACEE.
( ) ( )is
ijisji Q
tqRtr =n,,2,1irrrq 0i
n
ij0j
ji
n
ij1i
iji KK& =+−= ∑∑≠=
≠=
n,,2,1irqQR
qQR
q 0i
n
ij0j
iis
jisn
ij1i
jjs
ijsi KK& =+−= ∑∑
≠=
≠=
Non è possibile però ricavare informazioni sulle eventuali variabili di controllo (non entrano nell’equazione del tracciante). js
ijsij Q
Rk =
Equazioni della cinetica del tracciante: small signal perturbation.
L’equazione del tracciante è sempre lineare, ma in questo caso i parametri kijcorrispondono alla derivata del flusso rispetto alla quantità di tracee.
( ) ( ) L+⋅−+⋅−+=== jsijsj QQ
2j
ij2
2jsj
QQj
ijjsjijsij dQ
RdQQ
21
dQdR
QQRR
( ) ∑ ∑= = ==
=δ+⎟⎟
⎠
⎞
⎜⎜
⎝
⎛δ⋅+−
⎟⎟⎟
⎠
⎞
⎜⎜⎜
⎝
⎛δ⋅++=
δ+ n
1j0i
n
0ji
QQi
jijisj
QQj
ijijss0i
iis n,,2,1iRQdQdR
RQdQdR
RRdt
QQd
isijsj
K
( ) ∑ ∑= = ==
=δ+⎟⎟
⎠
⎞
⎜⎜
⎝
⎛δ⋅−
⎟⎟⎟
⎠
⎞
⎜⎜⎜
⎝
⎛δ⋅=δ
n
1j0i
n
0ji
QQi
jij
QQj
iji n,,2,1iRQ
dQdR
QdQdR
Qisijsj
K&
jsj QQj
ijij dQ
dRk
=
=
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4
( )( )
( )⎪⎪⎩
⎪⎪⎨
⎧
+⋅+⋅+−=⋅+⋅−=
⋅+⋅+−=+⋅+⋅+⋅++−=
2242414044
1313033
121242122
141421213121011
uqkqkkqqkqkq
qkqkkquqkqkqkkkq
&
&
&
&
( )( )
( )
⎥⎦
⎤⎢⎣
⎡=
⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢
⎣
⎡
=
⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢
⎣
⎡
+−−
+−++−
=
4
140442
0331
421221
1412312101
V1000
10000001
kk0k00k0k00kkk
k0kkkk
C
B
A
qCy
uBqAq
⋅=
⋅+⋅=dtd
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4
qAq
⋅=dtd
∑=
λ⋅α=n
1i
tii
ie)t( xq
( )
( )
( )
( ) ∑
∑
∑
∑
=
λ
=
λ
=
λ
=
λ
α=
α=
α=
α=
4
1i
ti4i4
4
1i
ti3i3
4
1i
ti2i2
4
1i
ti1i1
i
i
i
i
extq
extq
extq
extq
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4
( ) ( )
( ) ( ) ∑
∑
=
λ
=
λ
α==
α==
4
1i
ti4i2
u4
42
4
1i
ti4i1
u4
41
i
2
i
1
exV
tqty
exV
tqtyq1(0) = u1
q2(0) = q3(0) = q4(0) = 0
q4(0) = u2
q1(0) = q2(0) = q3(0) = 0
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4xxA ⋅λ=⋅
( ) 0=⋅λ− xIA
qAq
⋅=dtd
( )( )
( )⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢
⎣
⎡
++λ−−λ−
++λ−+++λ−
=⋅λ−
140442
0331
421221
1412312101
kk0k00k0k00kkk
k0kkkk
IA
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4
( )( )
( )⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢
⎣
⎡
++λ−−λ−
++λ−+++λ−
=⋅λ−
140442
0331
421221
1412312101
kk0k00k0k00kkk
k0kkkk
IA
( ) ( )⋅−λ−=λ− 03kDet IA
( )( )
( )=
⎥⎥⎥
⎦
⎤
⎢⎢⎢
⎣
⎡
++λ−++λ−
+++λ−
140442
421221
1412312101
kkk00kkk
kkkkkDet
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4
( ) ( )( ) ( ) ( )[ ]
( )[ ] 0kkkkkk
kkkkkkkkDet
421414041221
14044212312101
03
=⎭⎬⎫
⎩⎨⎧
⋅−−−λ−⋅⋅−++λ⋅++λ⋅+++λ−
⋅−λ−=λ− IA
( )
0kkkkkkkkkkkkkkkkkkkkkkkkkkk
kkkkkkkkkkkkkkkkkkkkkkkkkkkkkk
kkkkkkk
423114311412423104
422104311204421401141201420401120401
4231422142143114211431121412
42043104210412044201140112010401
4231211412040123
=+++++++++
⎟⎟⎠
⎞⎜⎜⎝
⎛+++++++
+++++++⋅λ
+++++++⋅λ+λ
( ) 0k03 =−λ− 031 k−=λ
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4
031 k−=λ ( ) 0ii =⋅λ− xIA( )
( )
( )
0
xxxx
kkk0k0000k00kkkk
k0kkkkk
41
31
21
11
14040342
31
42120321
141231210103
=
⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢
⎣
⎡
⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢
⎣
⎡
−−
−−−−−
( )( )
( )⎪⎪⎩
⎪⎪⎨
⎧
=⋅−−+⋅=⋅
=⋅−−+⋅=⋅+⋅+⋅−−−
0xkkkxk0xk
0xkkkxk0xkxkxkkkk
411404032142
1131
214212031121
411421121131210103
x11=0 x21=0 x41=0
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4
031 k−=λ
( )( )
( )⎪⎪⎩
⎪⎪⎨
⎧
=⋅−−+⋅=⋅
=⋅−−+⋅=⋅+⋅+⋅−−−
0xkkkxk0xk
0xkkkxk0xkxkxkkkk
411404032142
1131
214212031121
411421121131210103
x11=0 x21=0 x41=0
⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢
⎣
⎡
=
0100
1x
( ) ( )
( ) ( ) ∑
∑
=
λ
=
λ
α==
α==
4
1i
ti4i2
u4
42
4
1i
ti4i1
u4
41
i
2
i
1
exV
tqty
exV
tqty
Software per risoluzione dei modelli compartimentali
depts.washington.edu/saam2
www.webcom.com/rphair/bis/resources/kinweb.html
www.webcom.com/rphair/welcome.html#Free Resources
www.boomer.org/pkin/soft.html
courses.washington.edu/rfka
bmsr.usc.edu/index.html
Modello di varianzaCome varia il peso associato ad un dato
sperimentale a seconda delle opzioni scelte
ERRORE ESPRESSO COME
SD FSD
Dati Modello Dati Modello
ASSOASSOLUTOLUTO
RELARELATIVOTIVO
( ) ( )
( ) ( )22
22
22
22
sf1
yf1
sf1
yf1
d1
d1
d1
d1
⋅⋅ν⋅⋅ν
⋅⋅
⋅ν⋅ν
Criterio di convergenzaIn ogni iterazione, il programma cerca valori dei parametri che risultano in una diminuzione del
valore della funzione obiettivo. Se, per ogni parametro, la differenza tra il valore assunto dal p
nella i-esima iterazione e quello assunto nella iterazione precedente verifica la relazione:
allora si considera soddisfatto il criterio di convergenza.
( )LU1ii pppp −δ<− −
RESIDUI:
CHIQUADRO:
Iniettato Ingerito
Residuo Chiquadro Residuo Chiquadro0.147 0.022 0.202 0.041-0.735 0.540 -0.356 0.1270.365 0.133 0.622 0.3870.965 0.931 0.114 0.013-0.335 0.112 0.246 0.0610.653 0.426 0.160 0.0260.039 1.52e-3 0.517 0.267-0.286 0.082 0.101 0.010-1.876 3.519 -0.233 0.0541.090 1.188 -1.436 2.0620.476 0.227 0.494 0.2441.882 3.542 -0.748 0.560
10.724 3.851 14.575
( )i
ii ,xfywres_sσ
−=
p
( )∑ ⎟⎟⎠
⎞⎜⎜⎝
⎛σ
−=χ
i
2
i
ii2 ,xfy p
CHI QUADRO
Chiquadro calcolato: 14.575
Numero di gradi di libertà: numero di dati sperimentali
meno numero di parametri determinati: 24-7 = 17
Chiquadro ridotto: 0.857
Probabilità: 62.88 %(0.8: 70%; 1.0: 45%)
k21
k01
k12
1 2
k02
( ) ( )
( )⎪⎩
⎪⎨
⎧
⋅+−⋅=
−δ⋅+⋅+⋅+−=
212021212
00212121011
qkkqkdt
dq
ttqqkqkkdt
dq
( ) ( )0201 tttt
1
11 ebea
Vqy −⋅λ−⋅λ ⋅+⋅==
( ) ( )( ) ( )
( ) ( )( ) ( )
( ) ( ) ( )
( ) ( ) ( )2
kkkkkk4kkkkkkkk2
kkkkkk4kkkkkkkk
kkkkkk4kkkkV2
kkkkkk4kkkkkkkkqb
kkkkkk4kkkkV2
kkkkkk4kkkkkkkkqa
0201120102212
01210212012102122
0201120102212
01210212012102121
0201120102212
012102121
0201120102212
01210212021201210
0201120102212
012102121
0201120102212
01210212012102120
++⋅−+++−+++−=λ
++⋅−+++++++−=λ
++⋅−+++⋅⋅
++⋅−++++−−+=
++⋅−+++⋅⋅
++⋅−++++−−+=
0 2 4 6 8 10
Con
cent
razi
one
nel c
omp.
1 (u
.a.)
0
2
4
6
8
10
set 1set 2dati sperimentali
k21
k01
k12
1 2
k02
( ) ( )0201 tttt
1
11 ebea
Vqy −⋅λ−⋅λ ⋅+⋅==
0 2 4 6 8 10
Con
cent
razi
one
nel c
omp.
1 (u
.a.)
0
2
4
6
8
10
set 1set 2dati sperimentali
Tempo post-somministrazione (u.a.)
0 1 2 3 4
Con
centr
azio
ne
nel
com
p.
2 (
u.a
.)
0
5
10
15
20
25
30
k21
k01
k12
1 2
k02
( ) ( )0201 tttt
1
11 ebea
Vqy −⋅λ−⋅λ ⋅+⋅==
Un parametro pi si dice NON IDENTIFICABILEnell’intervallo [t0,T] se esiste un numero INFINITO di soluzioni. Se un modello ha anche un solo parametro NON IDENTIFICABILE, allora l’intera struttura si dice NON IDENTIFICABILE.
Un parametro pi si dice IDENTIFICABILEnell’intervallo [t0,T] se esiste un numero FINITO di soluzioni (diverse da quella identicamente nulla). Se tutti i parametri sono IDENTIFICABILI, allora l’intera strutturasi dice IDENTIFICABILE.
Un parametro pi si dice UNIVOCAMENTE IDENTIFICABILE nell’intervallo [t0,T] se esiste UNA E UNA SOLA soluzione. Se tutti i parametri sono UNIVOCAMENTE IDENTIFICABILI, allora l’intera struttura si dice UNIVOCAMENTE IDENTIFICABILE. Se anche un solo parametro non è UNIVOCAMENTE IDENTIFICABILE, allora l’intera struttura si dice NON-UNIVOCAMENTE IDENTIFICABILE.
( )
( ) ( )( ) ( ) ( )
( ) ( ) ( )⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢
⎣
⎡
=
− pBpApC
pBpApCpBpC
pJ
1n2M
⎥⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢⎢
⎣
⎡
∂∂
∂∂
∂∂
∂∂
p
n2
1
n2
p
1
1
1
pp
pp
JJ
JJ
L
MOM
L
2n sottomatrici di ordine mxr
p derivate per ogni termine di ogni sottomatrice
2nxmxrxp derivate!!!!!
METODOMETODO DELLA MATRICE DI MARKOVDELLA MATRICE DI MARKOV
n = numero compartimeniir = numero inputm = numero outputp = numero parametri
METODOMETODO DELLA MATRICE DI MARKOVDELLA MATRICE DI MARKOV
( )
( ) ( )( ) ( ) ( )
( ) ( ) ( )⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢
⎣
⎡
=
− pBpApC
pBpApCpBpC
pJ
1n2M
⎥⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢⎢
⎣
⎡
∂∂
∂∂
∂∂
∂∂
p
n2
1
n2
p
1
1
1
pp
pp
JJ
JJ
L
MOM
L
Il modello èidentificabile se e solo se IL RANGO DELLA MATRICEDELLE DERIVATE èuguale a p per ogni possibile valore del vettore p.
Il modello èidentificabile se e solo se IL RANGO DELLA MATRICEDELLE DERIVATE èuguale a p per ogni possibile valore del vettore p.
n = numero compartimeniir = numero inputm = numero outputp = numero parametri
mxr termini[ ][ ] [ ] )(B)(As)(
)t(uL),t(yL),s( 1
r,1jm,1ij
i ppIpCppH −
==
−=⎥⎥⎦
⎤
⎢⎢⎣
⎡=
( ) ( ) ( )( )
( )
( )∑
∑∑
=
−
=
−
= α+
β=
λ−== n
1k
1kijk
k
n
1k
1kijkn
1k k
ijk
ii
ss
s
sA
,sY),ty(Lp
p
pp
pp
( ) ( )[ ]( )[ ]
( )⎥⎦⎤
⎢⎣⎡=
⎥⎥⎦
⎤
⎢⎢⎣
⎡=
1,sY
tuL,tyL,sH i
j
iij
pppn = numero compartimeniir = numero inputm = numero outputp = numero parametri
METODOMETODO DELLA MATRICE DELLA FUNZIONE DELLA MATRICE DELLA FUNZIONE DI TRASFERIMENTODI TRASFERIMENTO
n = numero compartimeniir = numero inputm = numero outputp = numero parametri
Il modello èidentificabile se e solo se IL RANGO DELLA MATRICE G(p) è uguale a p per ogni possibile valore del vettore p.
Il modello èidentificabile se e solo se IL RANGO DELLA MATRICE G(p) è uguale a p per ogni possibile valore del vettore p.
( )
⎥⎥⎥⎥⎥⎥⎥⎥⎥⎥⎥⎥⎥⎥
⎦
⎤
⎢⎢⎢⎢⎢⎢⎢⎢⎢⎢⎢⎢⎢⎢
⎣
⎡
∂α∂
∂α∂
∂β∂
∂β∂
∂α∂
∂α∂
∂β∂
∂β∂
=
p
mrn
1
mrn
p
mr1
1
mr1
p
11n
1
11n
p
111
1
111
pp
pp
pp
pp
L
MM
L
MM
L
MM
L
pG
METODOMETODO DELLA MATRICE DELLA FUNZIONE DELLA MATRICE DELLA FUNZIONE DI TRASFERIMENTODI TRASFERIMENTO
2nxmxrxp derivate!!!!!
il sistema dev’essere “input-“ e “output-connectable”(OGNI COMPARTIMENTO E’ RAGGIUNGIBILE DA ALMENO UN INPUT ED E’ COLLEGATO AD ALMENO UN OUTPUT)
il sistema dev’essere “input-“ e “output-connectable”(OGNI COMPARTIMENTO E’ RAGGIUNGIBILE DA ALMENO UN INPUT ED E’ COLLEGATO AD ALMENO UN OUTPUT)
CONDIZIONICONDIZIONI NECESSARIE PER NECESSARIE PER LL’’IDENTIFICABILITAIDENTIFICABILITA’’
k21k01
k12
k14
k31
k03
k42 k04
1
3
2
4
il sistema dev’essere “input-“ e “output-connectable”(OGNI COMPARTIMENTO E’ RAGGIUNGIBILE DA ALMENO UN INPUT ED E’ COLLEGATO AD ALMENO UN OUTPUT)
il sistema dev’essere “input-“ e “output-connectable”(OGNI COMPARTIMENTO E’ RAGGIUNGIBILE DA ALMENO UN INPUT ED E’ COLLEGATO AD ALMENO UN OUTPUT)
CONDIZIONICONDIZIONI NECESSARIE PER NECESSARIE PER LL’’IDENTIFICABILITAIDENTIFICABILITA’’
k34
k43k32
1Stomach
2 Smallintestine
k21
k02 k03
4 Organs& tissues
3 Transfer compartment
nu ≤ nedove nu è il numero di parametri incogniti, e ne è dato dall’espressione:
n=numero di compartimenti
n’=numero di sottosistemi chiusi
whk = numero di compartimenti del sistema hk meno la lunghezza del percorso più breve (se l=0, si prende comunque =1)
zhk = si riferisce alle comuni parti in cascata; è uguale al numero dei compartimenti comuni meno la lunghezza del percorso più breve meno 1
nu ≤ nedove nu è il numero di parametri incogniti, e ne è dato dall’espressione:
n=numero di compartimenti
n’=numero di sottosistemi chiusi
whk = numero di compartimenti del sistema hk meno la lunghezza del percorso più breve (se l=0, si prende comunque =1)
zhk = si riferisce alle comuni parti in cascata; è uguale al numero dei compartimenti comuni meno la lunghezza del percorso più breve meno 1
CONDIZIONICONDIZIONI NECESSARIE PER NECESSARIE PER LL’’IDENTIFICABILITAIDENTIFICABILITA’’
∑∑==
==
==
==
−+−=mhrk
1k1h
hk
mhrk
1k1h
hke zw'nnn
∑∑==
==
==
==
−+−=mhrk
1k1h
hk
mhrk
1k1h
hke zw'nnn
1
7
2
3
6
8
5
4
2 percorsi 1->6: lunghezza 2 e 3
4 percorsi 1->4: lunghezza 2, 3 e 4
Compartimenti input connectable: 1,2,3,4,5,6,8
Compartimenti output connectable: 1,2,3,4,5,6,7
Sottosistema h=1, k=1: [1,2,3,6]
Sottosistema h=2, k=1: [1,2,3,4,5]
Sottosistemi chiusi: [6], [4,5]
Parti comuni in cascata: [1,2,3]
k34
k43k32
1Stomach
2 Smallintestine
k21
k02 k03
4 Organs& tissues
3 Transfer compartment
0232
321 kk
kf+
=
METODO DELLE FORCING FUNCTIONSHP: MODELLO CON COMPARTIMENTO CENTRALE COLLEGATO A STRUTTURE COMPARTIMENTALI
PERIFERICHE
k21k01
k12
k14
k31k41
1
3
2
4 Le strutture periferiche non sono note. Difficile caratterizzarle tutte insieme perché c’è una forte correlazione degli effetti conseguenti alle modifiche delle ipotesi cinetiche sulle varie strutture.
METODO DELLE FORCING FUNCTIONSSi considerano le singole strutture prese
separatamente.
In questo modo, è possibile definire le sottostrutture indipendentemente dal resto del modello.
Per far ciò, devo considerare nota la funzione che descrive l’andamento nel compartimento centrale.
k21k01
k12
k14
k31k41
1
3
2
4
METODO DELLE FORCING FUNCTIONS
La procedura seguita è la seguente:
Si definisce una funzione che descrive l’andamento nel compartimento centrale (in genere, una somma di esponenziali).
Si associa questa funzione al compartimento centrale utilizzando l’opzione “Forcing function”.
Per ogni compartimento periferico, si cerca la sottostruttura più adatta a descrivere i dati sperimentali misurati in quel compartimento, e che sia compatibile con la curva nel compartimento centrale (descritta dalla forcing function).
Una volta trovate tutte le sottostrutture, si ricostruisce il modello completo e si fa un fit complessivo per trovare i valori dei parametri anche per il compartimento centrale.
MODELLI DI VARIANZAVi sono situazioni in cui è giustificato pensare che gli errori determinati sperimentalmente sono insufficienti a indicare il grado di incertezza legato alle misure:
IN PARTICOLAR MODO NELLA MODELLIZIN PARTICOLAR MODO NELLA MODELLIZ--ZAZIONE DI PROCESSI METABOLICI PER ZAZIONE DI PROCESSI METABOLICI PER CUI LE LEGGI IPOTIZZATE SONO SOLO CUI LE LEGGI IPOTIZZATE SONO SOLO DELLE APPROSSIMAZIONI O DELLE DELLE APPROSSIMAZIONI O DELLE INTERPRETAZIONI DEL CASO REALE, E INTERPRETAZIONI DEL CASO REALE, E IN CUI LA VARIABILITA’ INTERIN CUI LA VARIABILITA’ INTER-- E E INTRAINDIVIDUALE SONO INTRAINDIVIDUALE SONO IMPREVEDIBILI.IMPREVEDIBILI.
MODELLI DI VARIANZA
In questi casi si ipotizza che l’errore sui singoli dati, da utilizzare nel processo di fit sia dipendente dalla previsione del modello.previsione del modello.
Ovviamente il risultato del fit dipenderà in maniera significativa dalle ipotesi che vengono fatte sul modello di varianza da prendere in considerazione.
CASO SEMPLICE
1 solo parametro 1 solo parametro (INTAKE I)(INTAKE I)
MODELLI DI VARIANZA
Si introduce questo valore nella formula della grandezza da minimizzare (Chi quadrato), e si va a IMPORRE che questa grandezza assuma il valore atteso, pari al NUMERO DI GRADI DI LIBERTA’ (cioè, pari al numero di dati sperimentali nn meno il numero i parametri derivati 11).
Ipotesi 1
ii2i FIyˆ ⋅⋅κ=⋅κ=σ
∑
∑
=
==n
1ii
n
1ii
F
yI
RATIO OF THE MEANS
MODELLI DI VARIANZA
Ipotesi 2
κ=σ2iˆ
∑
∑
=
=
⋅=
n
1i
2i
n
1iii
F
yFI
UNWEIGHTEDLEAST SQUARES
Ipotesi 3
( )2i2i FIˆ ⋅⋅κ=σ n
Fy
I
n
1i i
i∑==
SLOPES
i yi Fi1,00 25 502,00 12 253,00 10 204,00 7 155,00 5 126,00 4 107,00 3 88,00 2 69,00 1 4
10,00 1 26,95 15,20
0,46
i yi Fi Intake1,00 25 50 0,502,00 12 25 0,483,00 10 20 0,504,00 7 15 0,475,00 5 12 0,426,00 4 10 0,407,00 3 8 0,388,00 2 6 0,339,00 1 4 0,25
10,00 1 2 0,256,95 15,20 0,40
0,46
POPULATION KINETICS
Dati di diversi soggetti che cerco di analizzare con un modello comune.La population kinetics permette di identificare la VARIABILITA’ tra i soggetti (BSV: between subject variation) isolandola da tutte le altre “sorgenti” di incertezza (RUV: residual unknown variation).
SPK (https://spk.rfpk.washington.edu/info/)NONMEM (http://c255.ucsf.edu/nonmem0.html)PKBugs (http://ww1.imperial.ac.uk/medicine/about/divisions/ephpc/eph/bugs/pkbugs)WinNonmix (http://www.pharsight.com)NLME (http://stat.bell-labs.com/NLME/index.html)PopKinetics (http://depts.washington.edu/saam2/)
NPEM2 (http://www.lapk.org)
Rielaborazione presentazione del Prof. P.Vicini (UW-RFPK)
POPULATION KINETICS
Da individuo a popolazione
Molti individui (dati sparsi)
risposta di popolazione
Caratterizzare e modellare le fonti di variabilità
Time (a.u.)0 1 2 3 4 5
Dat
a (a
.u.)
0.01
0.1
1
exp(-0.05 t)exp(-0.25 t)exp (- t)
POPULATION KINETICS
Time (a.u.)0 1 2 3 4 5
Dat
a (a
.u.)
0.01
0.1
1
exp(-0.05 t)exp(-0.25 t)exp (- t) media dei dati
POPULATION KINETICS
y = (ey = (e--0.05t0.05t + e+ e--0.25t0.25t ++ ee--tt)/3)/3
Time (a.u.)0 1 2 3 4 5
Dat
a (a
.u.)
0.01
0.1
1
exp(-0.05 t)exp(-0.25 t)exp (- t) media dei datimedia dei parametri
POPULATION KINETICS
y = ey = e--(0.05+0.25+1) t/3(0.05+0.25+1) t/3
POPULATION KINETICS
Numero di soggetti MOLTI POCHI
Numero di dati
per soggetto
MOLTI Informazioni affidabili sia Informazioni affidabili solo per popolazione che per per individuoindividuo
POCHI Informazioni affidabili solo Informazioni inaffidabiliper popolazione
Notazioni individuali:
θ parametro incognito del modello
ω RUV incognita
Notazioni di popolazione
θ parametro incognito del modello
ω BSV incognita
σ RUV incognita
POPULATION KINETICS
Standard Two Stage (STS)
parametri cinetici stimati separatamente per ogni individuo, poi vengono valutati valor medio e covarianza
Iterative Two Stage (ITS)
Dopo STS, il valor medio e la covarianza sono usati come valore „bayesiano“ per i soggetti individuali.
Il processo viene iterato finchè non si giunge ad unaconvergenza
POPULATION KINETICS
Standard Two Stage (STS)
Modello risolto per ognuno dei soggetti e la media θ e la covarianza ω di ogni parametro vengono calcolate come la media e la covarianza del campione
La STS presuppone una distribuzione normale dei parametri
k01 = θk01+ η, con η ~ N(0,ωk01)
k01 ~ N(θk01, ωk01)
( )( )TSTSi
N
1i
STSi
STSN
1ii
STS ˆˆN1ˆ
N1
θ−θθ−θ=ωθ=θ ∑∑==
θk01 = valore tipico
ωk01 = BSV
POPULATION KINETICS
Iterative Two Stage (ITS)
Modello risolto per ognuno dei soggetti e la media θ e la covarianza ω di ogni parametro vengono calcolate come la media e la covarianza del campione ed utilizzate come stima Bayesiana a priori per una rianalisi dei dati individuali
E‘ molto utile e potente nel caso in cui vi siano singoliindividui con pochi dati
Fa variare i singoli parametri in un range „ristretto“ sullabase dei risultati dei fit individuali
( ) ( )ii Y,pY,p ωθ⇒⇒ωθ
POPULATION KINETICS
STS ITSk21 k02 V k21 k02 V
1 0.05395 1.7774 0.369262 0.10166 1.9427 0.440343 0.08142 2.4536 0.485834 0.08747 1.1715 0.427595 0.08844 1.4715 0.493066 0.09953 1.1637 0.513817 0.10225 0.6797 0.504618 0.09196 1.3756 0.505269 0.08663 8.8656 0.3773110 0.07397 0.6955 0.4386211 0.09812 3.8490 0.5834112 0.10558 0.8329 0.39779
0.0892 2.19 0.461
POPULATION KINETICS
STS ITSk21 k02 V k21 k02 V
1 0.05395 1.7774 0.36926 0.06565 1.6205 0.349882 0.10166 1.9427 0.44034 0.09542 1.9745 0.447223 0.08142 2.4536 0.48583 0.08142 2.4585 0.486324 0.08747 1.1715 0.42759 0.08606 1.1954 0.431965 0.08844 1.4715 0.49306 0.09912 1.3259 0.461296 0.09953 1.1637 0.51381 0.09708 1.1734 0.514997 0.10225 0.6797 0.50461 0.09326 0.7291 0.527838 0.09196 1.3756 0.50526 0.09552 1.3256 0.493039 0.08663 8.8656 0.37731 0.08452 5.4764 0.3756810 0.07397 0.6955 0.43862 0.07458 0.6966 0.4386011 0.09812 3.8490 0.58341 0.10098 3.6955 0.5749212 0.10558 0.8329 0.39779 0.08601 0.9774 0.44077
0.0892 2.19 0.461