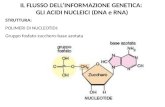
RNA POLIMERASI (direzione 3-5 lungo...
Transcript of RNA POLIMERASI (direzione 3-5 lungo...

La trascrizione è un sistema enzimatico che converte l'informazione genetica di un segmento di DNA a
doppia elica in una catena di RNA con una sequenza di basi complementari una delle due catene del DNA
che funge da stampo. Richiede eucromatina.
PROCARIOTI: dna stampo, fattore sigma di riconoscimento
EUCARIOTI: stampo di cromatina, piú sistemi di riconoscimento
RNA POLIMERASI (direzione 3-5 lungo DNA)
I: pre-rRNA
II: mRNA ----> 12 subunità, 500 kDa. La subunità maggiore possiede una lunga coda carbossi
terminale che consiste in 52 ripetizioni di una sequenza di 7 amminoacidi= CTD (YSPTSPS)
III: tRNA, rRNA 5s
RICHIEDE
Fattori generali di trascrizione: necessari alla polimerasi per iniziare la trascrizione. Hanno un
dominio di legame al DNA e uno di transattivazione per legare proteine che ne attivano la
trascrizione. Agiscono in modo sinergico. Alcuni sono ubiquitari, altri tissutali. VEDI: attivatori e
repressori.
Fattori specifici di trascrizione(proteine regolatrici dei geni): determinano la velocità alla quale un
particolare gene viene trascritto.
Rimodellatori di cromatina: per essere trascritto la struttura nucleosomica deve essere distrutta.
PROMOTORE: sequenza nucleotidica posta a monte o a valle del sito di trascrizione necessaria per
trascrizione di un gene.
Esempio CORE: TATA box, 24-32 basi prima del sito di inizio a 5'. Preceduta da Inr in cui si srotola DNA.
TBP: sequenza di riconoscimento del TATA, inclusa nel complesso TF2D. Il legame TBP-DNA è stabilizzato da
TF2A e TF2B. E' associata a 14 TAF: 1 con due bromodomini per interagire con lisine acetilate e attivare
promotore. Istoni sono deacetilati e non trascritti.
Complesso di preinizio: definito da 20 subunità, di cui 7 conservate. Dato da fattori generali di trascrizione,
DNA e polimerasi. Legame di TF2D a DNA promotore. ORDINE DI LEGAME: D, AB, E, F+Pol, H.
TF2H: attività elicasica che promuove lo svolgimento del DNA in prossimità del sito di inizio con idrolisi di
ATP; attività chinasica per fosforilazione CTD----> distacco TF2D ( tranne B) e scorrimento RNA polimerasi su
stampo. Defosforilazione per nuovo ciclo.
mRNA: coda di poliA in 3', capping di 7metilguanosina in 5'. Splicing di pre-mRNA nel nucleo per rimuovere
introni (non codificanti) e condensare esoni (codificanti): alternativo per ottenere diverse proteine dallo
stesso gene.
ENHANCER: sequenze 100bp bidirezionali che attivano la trascrizione reclutando TF. Possono essere lontani
centinaia o migliaia di bp dal promotore. Aumentano con la complessità fenotipica.
CO-ATTIVATORI: complessi proteici addizionali per trascrizione.
Esempio: MEDIATORE, 20-30 subunità, si lega a CTD stimolando la fosforilazione. Facilita interazione fra
Polimerasi, TF e altri componenti che regolano la trascrizione (metilazione, acetilazione, rimodellamento):
azione permessa dal DNA che si piega.
REGOLATORI ARCHITETTONICI: proteine non istoniche (HMG) che si legano al DNA e mediano la formazione
di anse, in modo che componenti distanti fra loro possano comunicare.
Esempio: Enhanceosoma del gene Interferon b, NfKB-Jun/ATF-Irf.
ISOLATORI: sequenze di DNA a cui si legano specifiche proteine che influenzano l’azione degli enhancer. Se
è fra promotore e enhancer spegne l'attività del primo sul secondo, se viene aggirato no.

CONTROLLO TRASCRIZIONE
Repressione: mediante un complesso con 3 componenti
o Subunità di legame con DNA (DBD): associata a URS1, sequenza repressoria
o Co-repressore: UME6
o HDAH: reprime trascrizione, reclutata da SIN3 (proteina adattatrice) e URS1
Attivazione
o NFkB: fattore di trascrizione coinvolto nel signaling associato a stress, ipossia…
Sub. P65 defosforilata: nel citoplasma associata a IkB
Sub. P65 fosforilata: dissociata da IkB (degradato), trascloca nel nucleo e attiva
geni responsivi a NFkB
o STAT: fattori di trascrizione associati a recettori citochine (EPO, GF) fosforilati da JAK
CLASSI STRUTTURALI di fattori di trascrizione
Elica-giro-elica: proteine omeotiche conservate nel dominio di legame al DNA e di transattivazione.
Presentano un omeodominio di 60 aa. Regolano geni omeotici , espressi durante lo sviluppo
embionale.
Elica-ansa-elica: formano omodimeri e eterodimeri
o MYO-D/ID:proliferazione muscolo se omodimero (MYO-D)---> eterodimero no legame DNA
BHLH: fattori di trascrizione con residui basici all’estremità N-terminale, legano
DNA (MYO-D)
HLH: fattori di trascrizione senza residui, non legano DNA (ID)
o MYC: per passare da G1-S. Potenziale oncogene, neuroblastoma. È un bHLH.
Eterodimeri con MAD tramite proteina MAX (sempre espresso nel ciclo cellulare,
bHLH)
MAD/MAX: agisce su SIN3 (repressione)
MYC-MAX: agisce su MIZ1 che non può reclutare p300/CBP (HAT) e quindi attivare
la trascrizione di p15, inibitore delle Cdk
Effetto complessivo: promozione proliferazione e apoptosi
Cerniera di Leucina: ansa di residui idrofobici. Un dominio lega il DNA e uno elica-ansa-elica è sulla
C-terminale.
o Jun: omodimeri e eterodimeri
o Fos: regolatore del ciclo cellulare, aumenta fra G1-S. Solo eterodimeri.
Zinc finger: il DBD è un’alpha elica stabilizzata da interazioni con Zn mediante due Hys (gli altri
legami di coordinazione dello Zn sono impegnati con Cys). Riconoscono sequenze di tre basi sul
solco maggiore del DNA. Esempio: recettori nucleari ormoni liposolubili.
La funzione dei fattori di regolazione della trascrizione è regolata da imput cellulari e segnali extracellulare:
de/fosforilazione, attacco del ligando, rilascio dell’inibitore, cambio del partner, scissione proteolitica.
IS

Non coding RNA: no traduzione, regolazione mrna
HOUSEKEEPING
Rrna
Trna
Snrna splicing
Snorna processamento rrna
Grna editing mrna
REGOLATI
Mirna endogeno
Sirna esogeno
Evidenze mirna:
• lin4, codifica per mirna di 22-66 nucleotidi---->mrna di lin14. Aumenta lin4, diminuisce lin 14.
• Let7
-C.elegans, sviluppo embrionale
-Petunie, iniezione rna ds=bianche
-Neurospore
-Mammiferi
MIRNA: ncrna di 22 nucleotidi, endogeno, sintetizzato a partire da precursori di dimensioni variabili
codificati da introni o esoni per altri mrna. Ne sono noti 851 con 34000 target.
SINTESI con 2 endonucleasi=rnasi III
• Drosha/DGCR8= taglio primirna (single strand, stem loop a forcina) in premirna (~80)
• Exportin 5/ Ran= trasporto nel citoplasma. Attività gtpasica= ran-gdp e exportin 5 tornano nel
nucleo, rilascio premirna.
• Dicer= Mirna quasi maturo accoppiato a piccola sequenza di mrna complementare
• Elicasi= rimuove mrna, mirna maturo si lega a Risc
• Risc= 3' UTR mrna complementare per degradazione o inibizione traduzione (interruzione,
decapping, deadenilazione e metilazione)
E’ espresso in modo regolato nei tessuti:
Iperespresso---> degradazione oncosoppressore
Downregolato---> no degradazione oncogene, cancro al polmone
MIR: gene codificante. Antimir come biomarcatori tumorali perchè estremamente stabili in fluidi e tessuti
(identificazione prima di procedure invasive).
RNA INTERFERENCE: meccanismo di silenziamento genico post trascrizionale per cui una molecola di rna
double strand puó interferire con l'espressione genica. Protezione nei confronti di rna ds estraneo (virus) o
endogeno (trasposoni). Controlla anche rimodellamento cromatina agendo a livello nucleare.
• Perfetta complementarietà (sirna): degradazione mrna
• Non perfetta complementarietà (mirna): repressione trascrizione
SINTESI
Dicer= Rna ds in sirna che si accoppia a mrna
Sirna è esogeno, contenuto in elementi estranei o silenziati nel genoma come virus e trasposoni. Piú facile
di knock-out (espressione fenotipica per rimozione di un gene) e studio di proteine.
IS

DEFINIZIONI
• Cromatina=DNA e proteine (istoniche e non)
• Cromosoma= particolare stato di condensazione della cromatina in metafase
• Nucleosoma=uguali masse di DNA ( parte esterna) e 8 istoni (coppie eterodimeriche H2A H2B/ H3
H4, parte interna) + H1 che puó essere rimosso senza influenzare la struttura nucleosomica dato
che è esterno. Il DNA compie 1,67 giri attorno all'ottamero istonico, per 10.2 bp/giro (10.5 in H2O).
• Distanza giri Dna: 80 bp
• Diametro nucleosomi: 10 nm
• Massa nucleosoma: 262 kDa
• Grandezza nucleosoma: 200 bp. Ripetute nel 95% del dna: nucleosoma monomerico=200
bp=un'unita di lunghezza di dna.
• Core del nucleosoma: 146 bp= unità minima per posizionamento
ISTONI: sono proteine, come tali possiedono domini proteici che servono per interagire con un substrato.
• Dominio histone fold: per ripiegare il DNA per la formazione dei complessi intermedi, ossia dimeri (
H2A e H2B) e tetrameri ( H3 e H4)
• Porzione ammino terminale: coda ricca di lisina
Le code degli istoni sono prive di ordine e fuoriescono sia da entrambe le facce del nucleosoma che fra i
due avvolgimenti del DNA: sono accessibili da parte delle proteasi e alle modifiche trascrizionali.
• Acetilazione: lisina. Si: ON. No: OFF. Fasi durante l'assemblaggio del nucleosoma.
• Metilazione: lisina e arginina
• Fosforilazione: serina e treonina
• Ubiquitinazione: lisina
CODICE ISTONICO= le code degli istoni modificati sono riconosciute da proteine con domini proteici specifici
• Bromodomini: istoni acetilati, BRD4
• Cromo domini: istoni metilati, HP1
• Domini Tudor
• Domini PhD
N.B. H2A.X distribuita nelle cellule eucariotiche: quando il DNA subisce una rottura questa variante viene
fosforilata e riconosciuta da enzimi che riparano il DNA.
CROMATINA: a seconda dell’ assemblamento nucleosomico
• Eucromatina: trascrizionalmente attiva, nucleosomi assemblati in modo lasso
• Eterocromatina: inattiva trascrizionalmente, nucleosomi compattati per inibire la trascrizione
ISOLATORE: barriera che impedisce la diffusione di eterocromatina e eucromatina, alterando zone di
trascrizione
Livelli di condensazione:
▪ Fibra da 10 nm: parzialmente svolta, costituita da una stringa di nucleosomi---->eucromatina
▪ Fibra da 30 nm: doppia elica, due file di nucleosomi avvolte----> eterocromatina.
H1=promuove assemblaggio fibra 30 nm.
Nella formazione del nucleosoma si hanno, nell’ordine, formazione di dimeri e tetrameri, associazione del
tetramero col dna, reclutamento del dimero. I nucleosomi si formano preferenzialmente sul DNA curvo e
sequenze AT (solco minore).
Si hanno due posizionamenti:
• Intrinseco: il DNA cattura l'ottamero in una precisa regione per motivi strutturali
• Estrinseco: qualche altro fattore (proteina non istonica) induce il posizionamento

REPLICAZIONE: separa i filamenti di DNA----> distrugge nucleosoma. Una volta che il DNA e’ stato replicato,
i nucleosomi si formano velocemente su entrambi i duplicati.Gli ottameri istonici non sono mantenuti
durante la replicazione (rimossi nella forca replicativa) mentre sono conservati i tetrameri e i dimeri:
trasferiti dietro le forche di replicazione e reinseriti nel nuovo DNA.
FACT: facilita la trascrizione della cromatina. La proteina smantella i nucleosomi riassemblandoli dietro l'
RNA polimerasi permettendo la trascrizione. Il meccanismo è necessario poiché l' rna polimerasi ha
dimensioni maggiori del nucleosoma. Gli ottameri istonici rimossi durante la trascrizione si riallacciano al
DNA non appena la polimerasi è passata.
-----------------------------------------------------------------------------------------------------------------------------------------------
L' eucromatina è dinamica: può essere rimodellata variando il posizionamento dei nucleosomi,
modificandoli covalentemente e inserendo varianti istoniche. Il rimodellamento ha come scopo il rendere
più accessibile la cromatina in modo da facilitare il legame e l' attività dei fattori di trascrizione che
regolano l'espressione del gene presente in quella regione o la inibiscono se i nucleo somi si spostano sul
promotore.
• SWI/SNF: rende nucleosomi più distanziati e disposti irregolarmente. Presenta un'attività ATPasica
e un bromodominio. (NURF)
• SWR1: inserimento varianti istoniche
• HAT: acetila e attiva la trascrizione. Si può legare a residui acetilati degli istoni tramite
bromodomini.
➢ Gruppo A: su istoni della cromatina, regolano la trascrizione
➢ Gruppo B: su istoni di nuova sintesi presenti nel citosol, regolano l’assemblaggio di nuovi
nucleosomi
• HDAC: toglie i residui acetilati e inibisce la trascrizione. Fa parte di complessi di repressione.
La proteine oncogeniche di adenovirus e polioma SV40 legano p300/CBP, impedendo acetilazione (H4) e
reprimendo la trascrizione di geni specifici inibitori del ciclo cellulare (oncosoppressori)---> meccanismo
tumorigenico di virus oncogeni a DNA.
SITI IPERSENSIBILI alla Dnasi: promotori, OR e centromeri. Sono generati dal legame di proteine che
escludono gli ottameri istonici.
EPIGENETICA: branca della biologia molecolare che studia le mutazioni genetiche e la trasmissione di
caratteri ereditari non attribuibili direttamente alla sequenza del DNA.
• Esistenza di una struttura molecolare che si autoperpetua e che non dipende dalla sequenza del
DNA. Esempio: mantenimento di strutture proteiche durante la replicazione.
• Capacità di diverse condizioni, che possono avere diverse conseguenze fenotipiche, di essere
ereditate senza alcuna modifica della sequenza di DNA. Esempio: due individui con la stessa
sequenza di DNA in un determinato locus, possono esibire due diversi fenotipi
CARATTERE EPIGENETICO: fenotipo stabilmente ereditato che risulta da modificazione di un cromosoma in
assenza di alterazioni della sequenza di DNA. Dipende da
• Segnali dall’ambiente extracellulare: mitogeni, temperatura (che agiscono anche su DNA!)
• Iniziatori epigenetici: DnaBF e ncrna, causano modificazione epigenetica
• Mantenitori: modificazioni istoni, meccanismi molecolari che mantengono la condizione
epigenetica nella prima e nelle successive generazione (mantengono metilazione del Dna figlio--->
metilazione=eredità epigenetica)
METILAZIONE: meccanismo di inattivazione trascrizione su

• Dna=in citosina, sempre inibitoria
• Istoni=metilati su Arg9 da istone metil trasferasi---> cromodominio di HP1. In generale puó inibire o
attivare la trascrizione a seconda dei residui metilati ( Arg2 volte, Lys 3 volte) e dal tipo di lisina.
N.B. metilazione in citosina-->metilcitosina-->mutazione in timina. Azacitidina non puó essere metilata.
Isole CpG: regioni in cui la densità del dimero citosina-fosfato/guanina è 10 volte + del resto del genoma. [In
generale la presenza di CpG è 80% minore di quella prevista su basi genomiche perchè una buona parte di C
è metilata]. Sono 45000 in totale, spesso su 5': metilazione dell'isola=no attivazione promotore=no
trascrizione gene.
ENZIMI
DNTM: enzima metilazione. Assenza gene=no vita.
• DNMT1: metiltrasferasi di mantenimento, replicazione
• DNMT2 non metila DNA, ma citosina 38 sulla sequenza anticodone del tRNAAsp.
• DNMT3A/3B: metilazione de novo, stabiliscono i pattern di metilazione durante lo sviluppo
embrionale.
PASSAGGI:
• Istone metiltrasferasi: metila istone su Arg9 e recluta HP1 (inibitore trascrizione) con cromodominio
• HP1: recluta DNMT1
• DNMT1: metila DNA e recluta altre proteine inibitorie come MECP2
N.B. EZH1 fa parte di PRC2 che metila Arg27 su H3
IMPRINTING GENOMICO
Nelle cellule germinali primordiali vi sono metilasi e quindi geni non espressi. All’inizio della gametogenesi
le metilazioni sono cancellate da una demetilasi.Durante la gametogenesi è avviato uno schema di
metilazione specifico per ciascuno sesso: gameti femminili (si) e gameti maschili (no). Fra la fecondazione e
la blastocisti, le Xen-cells vanno incontro a demetilazione generalizzata (eccetto i geni imprinted nella
gametogenesi).
La metilazione del DNA avviene DURANTE IL DIFFERENZIAMENTO dei diversi tessuti a partire dalle prime
fasi dell‘embriogenesi. La metilazione del cromosoma X è casuale.
CHIP: immunoprecipitazione della cromatina, per vedere se dna è metilato (uso anticorpi istoni)
POLYCOMB: reprimono geni che promuovono la risposta apoptotica di senescenza alle mutazioni
oncogeniche e permettono l'acquisizione da parte delle cellule tumorali di caratteristiche staminali.
Numerose proteine PcG sono sovraespresse nei tumori a causa di downregolazione di fattori di trascrizione
“cell fate” e di ncRNA.
MALATTIE EPIGENETICHE E CURE
• Linfoma cutaneo delle cellule T: SAHA, inibitore delle deacetilasi istoniche con potenziale anti-
tumorale.
• Sindrome Mielodisplastica: 5-azacitidina, inibitore delle DNA metil-transferasi
• Leucemia Acuta Promielocitica: causata da traslocazione di un recettore alfa dell’acido retinoico,
presente sul cromosoma 17,con il gene PML (da leucemia promielocitica) situato sul cromosoma
15, generando un mRNA di fusione PML/RAR-alfa che codifica per una proteina chimerica. Il tipico
blocco maturativo che caratterizza le cellule della LAP dipende dalla capacità di PML/RAR-alfa di
legarsi al complesso corepressore-istone deacetilasi (HDAC); tale legame determina una

repressione trascrizionale. Quando utilizzato a dosi terapeutiche l’ATRA (ligando naturale di RAR-
alfa) determina il rilascio del complesso PML/RAR-HDAC nonché la conversione di PML-RAR-alfa in
attivatore trascrizionale; ciò comporta l'induzione del differenziamento con remissione clinica della
malattia.
IS

CELLULA TUMORALE: Si divide e prolifera bypassando i controlli imposti alle cellule normali. Invade e
colonizza territori normalmente riservati ad altre cellule.
CARATTERISTICHE
Immortalizzazione: crescita incontrollata, evasione apoptosi.
Trasformazione: NON vengono mantenute queste caratteristiche base della cellula primaria che le
permettono, in coltura, di crescere in un monostrato
o Anchorage dependence: necessità di una superficie solida per la crescita.
o Serum dependence (growth factor dependence): il siero fornisce i fattori di crescita
necessari alla proliferazione.
o Density-dependent inhibition: il contatto fra le cellule ne inibisce la crescita, ovvero esiste
un limite di densità---> Nelle cellule tumorali si ha sovrapposizione di strati cellulari,
sconfinando in aree tissutali deputate ad altre cellule.
o Cytoskeletal organization:le cellule sono piatte e si estendono lungo la superficie su cui
crescono, presentano un network allungato di fibre actiniche--->cellule tumorali globose.
Metastasi: colonizzazione e neoangiogenesi. Quando i tumori metastatizzano, perdono la capacità
di aderire alla matrice.
La maggior parte dei tumori ha un' origine monoclonale, deriva ossia da un’unica cellula.
Evidenze: alterazioni a livello genetico che si ritrovano in tutte le cellule tumorali (traslocazioni
cromosomiche leucemie)
AGENTI CANCEROGENI
Mutagenici: sostanze chimiche e agenti fisici che possono causare mutazioni casuali che
o Aumentano la proliferazione cellulare: una popolazione espansa di cellule è piú sensibile a
successive mutazioni
o Diminuiscono la stabilità del genoma (ben visibile nei cariotipi aberranti): il tasso di
mutazione complessivo aumenta
Virus: possono alterare la funzione dei geni che controllano proliferazione, differenziamento e
morte cellulare (base della trasformazione neoplastica)
I tumori possono essere benigni (poliposi, APC), maligni (RB) o l’espansione di sindromi predisponenti
(atassia, xeroderma).
Mutazioni cellule germinali: carattere ereditario predisponente. La malattia interessa la progenie,
non il genitore. Per lo piú letale durante la vita prenatale (tipica di oncosoppressori).
Mutazione cellule somatiche: tumore (=tumefazione, per proliferazione incontrollata di cellule)
nell’organismo portatore.
MA una singola mutazione non è sufficiente a causare l’insorgenza di un tumore. Lo sviluppo di una cellula
cancerosa richiede multipli eventi di mutazione: se una singola mutazione fosse sufficiente a causare un
tumore ci si aspetterebbe che l’incidenza tumorale fosse indipendente dall’eta’. Con il tempo le mutazioni a
carico di una cellula si accumulano. Si parla pertanto di progressione del tumore.
Numero di eventi necessari: 6
Normale tasso di mutazione cellulare: 10-7
Numero di geni in una cellula: 106
Numero di cellule per persona: 10 13
Probabilità di sviluppare un tumore: 10 13 x 10-42 = 1/10 29
Il tumore è un disordine poligenico : il bersaglio non è un solo gene deputato alla proliferazione ma una
fitta rete di segnali che la cellula invia ai tessuti e allo stroma.

Si evidenziano una fase di
Iniziazione: fattori che predispongono a neoplasie
Promozione: GF, TF o ormoni alterati/in eccesso che inducono trasformazione/proliferazione
Progressione: somma di iniziazione e promozione su oncogeni o oncosoppressori
PROTO-ONCOGENE: gene fisiologicamente espresso nella cellula, codificante per proteine coinvolte nella
crescita e proliferazione cellulare, con funzione regolare e regolata. È un potenziale oncogene.
ONCOGENE: identificato in virus che causano tumori, è un proto-oncogene che per mutazione ha acquisito
proprietà di gene dominante.
POTENZIALI ONCOGENI
Fattori di crescita: EGF, PGDF, HGF, CSF (fattore stimolante le colonie)
Recettori di superficie: EGFR, PGDFR, Met
Trasduttori del segnale: src, abl, ras
Fattori trascrizionali: myc, jun, fos
Regolatori positivi del ciclo cellulare: ciclina D, CDK4
Inibitori di apoptosi: bcl-2
ATTIVAZIONE ONCOGENI
Mutazioni Puntiformi: produzione proteina anomala, costitutivamente attivata (gain of function)
Esempio: Ras (12) nel carcinoma vescicale
Amplificazione Genica:aumento del numero di copie di un gene (~tumori solidi)
Esempio: Myc nei carcinomi polmonari e epatici
ErbB nei tumori mammari e ovarici
Traslocazione Cromosomica: spostamento di un frammento cromosomico ad un altro.
Esempio: Bcl2 (BAX) proto-oncogene espresso nel mitocondrio dove media la permeabilità per
cytp450, coinvolto in apoptosi. Gli effetti sono simili a quelli dell’amplificazione (linfomi, leucemia)
Inversione Cromosomica: rotazione di un frammento di 180° sullo stesso cromosoma.
Esempio: Abl, tirosina chinasi. Se mutata continua a fosforilare causando tumore (leucemia)
ONCOSOPPRESSORI: in condizioni normali codificano per proteine che inibiscono la proliferazione cellulare.
Delezioni o mutazioni inattivanti (metilazione, deacetilazione/acetilazione= epigenetica) portano a tumori
MA solo in condizione di omozigosi: se uno degli alleli è normale avverrà una normale inibizione della
divisione cellulare. L’introduzione di oncosoppressori normali in una cellula neoplastica la guarisce. Quindi
la mancata attività di un oncosoppressore favorisce la crescita di un tumore, cosí come la perdita della
condizione di eterozigosi (spesso è già presente una mutazione germinale che predispone al cancro).
Sono divisi in:
*GATEKEEPER: regolano – proliferazione e + apoptosi. L’inattivazione di entrambi gli alleli porta a
perdita della funzione e a un vantaggio selettivo nella crescita.
*CARETAKER: controllano l’integrità del genoma (duplicazione e riparazione). L’inattivazione di
entrambi gli alleli determina aumento dell’instabilità genomica e maggiore frequenza di alterazione
di oncogeni e oncosoppressori.
ESEMPI
P53 (*G e *C): 4 domini, fattore di trascrizione per geni di ciclo cellulare e apoptosi.Nel 70% dei
tumori umani p53 è assente (nel 20% dei casi si ha mutazione di DMD).
o In c.n. p53 è inattiva a causa del legame con MDM2 che la ubiquitina e degrada tramite
proteasoma

o A seguito di danni al DNA o se stimolata da oncogeni (tramite p19ARF) è fosforilata da
ATM. Se i danni al DNA sono troppo estesi, p53 induce apoptosi (via FAS, IGFB3, BAX)
o P53 migra nel nucleo dove si lega a p21
o P21 inibisce il complesso ciclinaD/Cdk4, bloccando il ciclo cellulare prima della fase S e
consentendo la riparazione. Frena così la proliferazione di cellule con DNA alterato.
RB(*G) : lega, se defosforilato, E2F=responsabile dell’entrata nella fase S del ciclo cellulare. Se RB
viene deleto, E2F è libero e induce le cellule a proliferare costantemente, causando retinoblastoma.
o P53 induce p21
o P21 inibisce ciclinaE/Cdk2
o CiclinaE/Cdk2 fosforila Rb
o Rb rilasciaE2F
BRCA1/BRCA2: tumori ovaio e mammella
APC, WT1, NF1, VHL……..
NOTE:
Oncogene: Alterazione del DNA che converte i proto-oncogeni in oncogeni (cancer-causing genes). Una sola copia di oncogene attivo è sufficiente a produrre proteine stimolatrici della crescita cellulare o responsabili della trasformazione.
Oncosoppressore: Perdita o alterazione del DNA che inattiva i geni onco-soppressori: mancata produzione di proteina inibitrice della crescita. Entrambe le copie del gene sono perdute o inattivate.
CICLINE:
G1: aiutano G1/S
G1/S: attivano Cdk alla fine di G1--->progressione attraverso Start
S: legano Cdk dopo Start e stimolano duplicazione cromosomi (anche alcuni eventi mitosi)
M: attivano Cdk che stimolano la progressione oltre G2/M, in mitosi
CHINASI CICLINA DIPENDENTI: non presentano attività chinasica nella forma monomerica. Devono
associarsi a cicline. I livelli di Cdk sono costanti, quelli delle cicline variano a seconda della fase del ciclo
cellulare. Il complesso ciclina/Cdk è attivato al 100% solo quando fosforilato da CAK ( chinasi che attiva Cdk)
in un punto adiacente il sito attivo---> modificazione conformazionale per cui Cdk puó fosforilare i bersagli e
indurre eventi del ciclo cellulare. Per essere attiva Tyr15 è defosforilata, Tyr160 fosforilata.
INIBIZIONE
CKI: proteina inibitrice che induce modificazione conformazionale
APC/C: transizione metafase-anafase, inattivo in G1.Catalizza ubiquitinazione di securina (mantiene
uniti cromatidi fratelli) e cicline M/S---> defosforilazione Cdk
SCF: ubiquitina CKI (fosforilata da Cdk 4-6) in G1 controllando attivazione di S/Cdk e replicazione
FATTORI DI CRESCITA: spingono cellula da G0 a G1.
Immediate early genes: Jun/Fos (complesso A1). Sono attivati da GF tramite Ras-Raf1-Mek-Erk-TCF
(fattore del complesso ternario)-SRF---->Fos
Delayed genes: Myc. Indotto da Jun/Fos.Eterodimeri con Max.
TGFbeta: inibisce l’ingresso in fase S inducendo l’espressione di inibitori del ciclo cellulare e reprimento la
trascrizione di Myc. Fosforila inoltre Rb.
INK4: p15, p16, p18, p19---> inibiscono ciclinaD/Cdk4
Cip/Kip: p21, p27, p57---> inibiscono ciclinaE/Cdk2
IS