Nozioni base di Biologia -...
-
Upload
truongnhan -
Category
Documents
-
view
213 -
download
0
Transcript of Nozioni base di Biologia -...
Informatica e Bioinformatica – A. A. 2013-2014 1
Ripercorriamo velocemente i principali concetti di biologia indispensabili per
capire la Bioinformatica: verranno approfonditi in altri corsi.
Gli organismi viventi possiedono tutti un genoma, il quale contiene tutta
l’informazione biologica necessaria alla costruzione e alla sopravvivenza
di un individuo.
L’informazione biologica contenuta in un genoma è codificata nell’acido
deossiribonucleico (DNA) ed è suddivisa in unità discrete chiamate geni.
I geni codificano tutte (e non solo) le proteine di un organismo, comprese
quelle che si legano in appropriati punti del genoma regolando una serie di
reazioni definite espressione genica.
Nozioni base di Biologia
Informatica e Bioinformatica – A. A. 2013-2014 2
Procarioti: organismi unicellulari con una organizzazione relativamente semplice.
Non contengono particolari organelli, caratteristici degli eucarioti.
Il materiale genetico (DNA) non è racchiuso in una particolare struttura.
Gli Eucarioti hanno un nucleo, dove è contenuto il DNA, ed hanno dei
compartimenti interni, racchiusi da membrane, chiamati organelli, che assolvono
particolari compiti biologici (complesso del Golgi, lisosomi, mitocondri, ecc.).
Procarioti ed Eucarioti
Informatica e Bioinformatica – A. A. 2013-2014 3
Gli acidi nucleici (DNA: acido deossiribonucleico e RNA: acido
ribonucleico) sono dei polimeri organici costituiti da monomeri
chiamati nucleotidi.
I nucleotidi sono formati da tre elementi fondamentali:
un gruppo fosfato
una molecola di zucchero pentoso (deossiribosio nel DNA o ribosio nell’RNA)
una base azotata che si lega allo zucchero con legame N-glicosidico.
Gli acidi nucleici sono formati solo da
quattro tipi di basi azotate:
adenina, guanina, citosina (comuni al
DNA e all’RNA);
la timina presente solo nel DNA;
l’uracile solo nel RNA.
Acidi nucleici: DNA e RNA
Informatica e Bioinformatica – A. A. 2013-2014 4
Il DNA esiste prevalentemente in forma di doppia elica antiparallela, in cui due filamenti
sono appaiati e avvolti tra loro.
Le basi si appaiano all’interno della doppia elica
secondo la regola:
Adenina con Timina (A-T)
Guanina con Citosina (G-C)
Due basi in grado di appaiarsi tra loro vengono
dette complementari.
Di conseguenza, se si conosce la sequenza di
un’elica si può ricavare anche la sequenza
dell’elica complementare.
Nota:
Negli eucarioti, il DNA si dispone all'interno del nucleo in strutture chiamate cromosomi.
Negli altri organismi, privi di nucleo, esso può essere organizzato in cromosomi o meno e
risiede nel citoplasma.
Informatica e Bioinformatica – A. A. 2013-2014 5
La direzionalità delle molecole di DNA
5' nnCGATGCTAGTAGTTGTACGCAnn -> 3‘OH
|||||||||||||||||||||
HO-3‘<- nnGCTACGATCATCAACATGCGTnn - 5'
Una rappresentazione in formato testo di una doppia elica di DNA
5‘-CGATGCCACCAAGTTGTACGCA-> 3‘OH
Nota: il DNA può essere costituito da catene lunghissime di basi. Il genoma
umano nei suoi 46 cromosomi, contiene circa 3.3 miliardi di basi!!
La complementarietà delle basi consente di poter archiviare solo una delle
due eliche di una molecola di DNA, che viene in genere rappresentata in
direzione 5’ -> 3’
Qual è il numero di tipologie di basi azotate che vi attendete?
Informatica e Bioinformatica – A. A. 2013-2014 6
Le Proteine
Fin dall'inizio del secolo scorso sappiamo che le proteine sono responsabili di molti
processi biochimici.
Le proteine sono polimeri lineari di aminoacidi, uniti chimicamente l'uno all'altro
tramite legame peptidico. Le proteine sono costituite essenzialmente da 20 possibili
aminoacidi diversi.
La sequenza con cui gli aminoacidi si succedono l'uno all'altro determina le proprietà di
ogni proteina. Esistono proteine di lunghezze molto diverse, da pochi aminoacidi (in
questo caso sono generalmente chiamate peptidi) a diverse migliaia di aminoacidi.
In una proteina la sequenza di aminoacidi ha una direzione. Per convenzione la sequenza
si scrive a partire dall'estremità NH2-terminale (che è la prima ad essere sintetizzata) e si
procede verso l’estremità COOH-terminale.
Informatica e Bioinformatica – A. A. 2013-2014 7
Strutture delle proteine
Struttura
primaria:
sequenza degli
aminoacidi
Struttura
terziaria:
la struttura
tridimensionale
della proteina.
Struttura quaternaria:
determinata dal fatto che
molte proteine sono
costituite da più subunità.
Struttura secondaria: le proteine tendono ad
assumere conformazioni
locali particolari molto comuni,
come α-eliche e foglietti-β
Informatica e Bioinformatica – A. A. 2013-2014 8
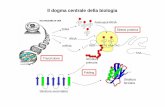
Il dogma centrale della Biologia
Duplicazione
Porta alla formazione di nuove
molecole di DNA e al trasferimento
del materiale genetico.
Trascrizione
L’informazione contenuta nel
DNA passa alle molecole di RNA.
Traduzione
Processo finale in cui dall’RNA si
arriva alla sintesi delle proteine.
Come l’informazione biologica presente nel genoma è “utilizzata”
per la sintesi delle proteine?
Informatica e Bioinformatica – A. A. 2013-2014 9
La trascrizione del DNA
Processo nel quale l’RNA (acido ribonucleico)
è sintetizzato a partire dal DNA stampo. L’RNA è un acido nucleico, il cui scheletro contiene
zucchero ribosio coniugato con le basi Adenina,
Guanina, Citosina e Uracile (U, al posto di T).
La sintesi dell’RNA avviene in direzione 5’ 3’
ad opera di enzimi detti RNA polimerasi, che
generano una copia ad RNA del DNA stampo.
Le molecole prodotte possono essere diverse. Si
distinguono (lista parziale) in:
RNA messaggero o mRNA
RNA transfer o tRNA
RNA ribosomali o rRNA
ognuna delle quali è coinvolta, con ruoli diversi,
nella sintesi proteica.
Informatica e Bioinformatica – A. A. 2013-2014 10
L’azione delle RNA polimerasi è in qualche modo “guidata”dalla presenza sul DNA
di particolari box di sequenza che vengono riconosciuti da fattori di trascrizione.
Non tutto il DNA viene trascritto in RNA.
Alcune parti del DNA forniscono informazioni su:
inizio (segnale d’inizio) e fine (segnale di stop) della trascrizione;
regolazione della trascrizione (nello stesso organismo non tutti i geni sono
trascritti in tutte le cellule) promotori, repressori della trascrizione.
Informatica e Bioinformatica – A. A. 2013-2014 11
La struttura del gene
La definizione di “gene” nel corso degli anni è cambiata (e continua a cambiare),
ma possiamo comunque rimarcare una sostanziale differenza tra i geni dei
procarioti e degli eucarioti.
5’UTR 3’UTR
Nei procarioti
Il gene corrisponde in genere all’intera sequenza
presente nell’mRNA.
L’mRNA include la porzione che corrisponde
alla sequenza amminoacidica, che viene chiamata
regione codificante (CDS), ma anche delle
sequenze aggiuntive ad entrambe le estremità,
non codificanti, indicate come 5’ UTR e 3’ UTR.
Informatica e Bioinformatica – A. A. 2013-2014 12
Generalmente i geni degli eucarioti (in particolare degli eucarioti superiori) sono
interrotti da introni.
Trascrizione: si genera il trascritto primario
Maturazione dell’RNA
Splicing degli introni (la loro eliminazione),
e l’unione degli esoni a formare il trascritto
maturo (RNA messaggero maturo).
esoni
introni
Rappresentazione schematica del DNA
genomico del gene della -globina
Messaggero maturo
Negli eucarioti
Informatica e Bioinformatica – A. A. 2013-2014 13
>gi|21359948|ref|NM_021245.2| Homo sapiens myozenin 1 (MYOZ1), mRNA
GTTTCTCCCTAAGTGCTTCTTTGGATCTCAGGCTCTAGGTGCAATGTGAAGGGGAGTCCCTGGGCAGACTGATCCCTGGCTCAGACAGTTC
AGTGGGAGAATCCCAAAGGCCTTTTCCCTCCTTCCTGAGCCTCCGGGCAAGGAGGGAGGGATCTTGGTTCCAGGGTCTCAGTACCCCCTGT
GCCATTTGAGCTGCTTGCGCTCATCATCTCTATTAATAACCAACTTCCCTCCCCCACTGCCAGTGCTGCCCCCACGCCTGCCCAGCTCGTG
TTCTCCGGTCACAGCAGCTCAGTCCTCCAAAGCTGCTGGACCCCAGGGAGAGCTGACCACTGCCCGAGCAGCCGGCTGAATCCACCTCCAC
AATGCCGCTCTCAGGAACCCCGGCCCCTAATAAGAAGAGGAAATCCAGCAAGCTGATCATGGAACTCACTGGAGGTGGACAGGAGAGCTCA
GGCTTGAACCTGGGCAAAAAGATCAGTGTCCCAAGGGATGTGATGTTGGAGGAACTGTCGCTGCTTACCAACCGGGGCTCCAAGATGTTCA
AACTGCGGCAGATGAGGGTGGAGAAGTTTATTTATGAGAACCACCCTGATGTTTTCTCTGACAGCTCAATGGATCACTTCCAGAAGTTCCT
TCCAACAGTGGGGGGACAGCTGGGCACAGCTGGTCAGGGATTCTCATACAGCAAGAGCAACGGCAGAGGCGGCAGCCAGGCAGGGGGCAGT
GGCTCTGCCGGACAGTATGGCTCTGATCAGCAGCACCATCTGGGCTCTGGGTCTGGAGCTGGGGGTACAGGTGGTCCCGCGGGCCAGGCTG
GCAGAGGAGGAGCTGCTGGCACAGCAGGGGTTGGTGAGACAGGATCAGGAGACCAGGCAGGCGGAGAAGGAAAACATATCACTGTGTTCAA
GACCTATATTTCCCCATGGGAGCGAGCCATGGGGGTTGACCCCCAGCAAAAAATGGAACTTGGCATTGACCTGCTGGCCTATGGGGCCAAA
GCTGAACTTCCCAAATATAAGTCCTTCAACAGGACGGCAATGCCCTATGGTGGATATGAGAAGGCCTCCAAACGCATGACCTTCCAGATGC
CCAAGTTTGACCTGGGGCCCTTGCTGAGTGAACCCCTGGTCCTCTACAACCAAAACCTCTCCAACAGGCCTTCTTTCAATCGAACCCCTAT
TCCCTGGCTGAGCTCTGGGGAGCCTGTAGACTACAACGTGGATATTGGCATCCCCTTGGATGGAGAAACAGAGGAGCTGTGAGGTGTTTCC
TCCTCTGATTTGCATCATTTCCCCTCTCTGGCTCCAATTTGGAGAGGGAATGCTGAGCAGATAGCCCCCATTGTTAATCCAGTATCCTTAT
GGGAATGGAGGGAAAAAGGAGAGATCTACCTTTCCATCCTTTACTCCAAGTCCCCACTCCACGCATCCTTCCTCACCAACTCAGAGCTCCC
CTTCTACTTGCTCCATATGGAACCTGCTCGTTTATGGAATTTGCTCTGCCACCAGTAACAGTCAATAAACTTCAAGGAAAATGAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAA
Nell’mRNA sono presenti anche regioni non tradotte
Solo la parte centrale (in blu e sottolineata) codifica la relativa proteina (MYOZENIN 1).
La regione a monte è la 5’UTR, la regione a valle la 3’UTR.
Sequenza di un trascritto (MYOZ 1), in formato FASTA, ottenuta interrogando un
database di sequenze nucleotidiche
Informatica e Bioinformatica – A. A. 2013-2014 14
Durante lo splicing, gli esoni degli eucarioti possono essere combinati (riarrangiati) in
modi diversi tra loro.
Si ottengono così differenti mRNA che codificano differenti proteine a partire dallo
stesso gene.
Questo meccanismo consente di amplificare la quantità di informazione contenuta
nel genoma. Nell’uomo si stimano circa 20-25000 geni che, con questo meccanismo,
possono codificare più di 100.000 differenti proteine.
Esempio di splicing alternativo
L’uso alternativo dello splicing nel gene dei vertebrati
per la calcitonina/CGRP, genera un ormone che
regola l’omeostasi del calcio nella ghiandola tiroide
(in verde) o un neuropeptide vasodilatatore nel
sistema nervoso (in rosso).
Solo negli eucarioti Splicing alternativo
Come passare dagli mRNA alle proteine?
Informatica e Bioinformatica – A. A. 2013-2014 15
La traduzione (translation)
L’informazione codificata nell’mRNA, utilizzando “solo” 4 tipi di nucleotidi
deve poter generare ben 20 differenti aminoacidi.
Consideriamo la sequenza lineare di RNA: 5-AUGAUCAGAAUCG……3
Se leggiamo 1 base alla volta (A, U, G, A, U, C,…….) esisterebbero solo 4 aminoacidi
2 basi (AU, GA, UC, AG,……): 42 combinazioni = 16 aminoacidi, non basta!
3 basi (AUG, AUC, AGA,…...): 43 combinazioni = 64 aminoacidi, anche troppi, ma è
proprio così.
La cellula possiede un sistema di interpretazione di questo codice genetico.
La regione codificante è letta tre basi alla volta, senza sovrapposizioni:
ogni gruppo di tre basi è chiamato tripletta o più propriamente codone.
Tutti gli organismi hanno essenzialmente lo stesso codice genetico con qualche
piccola eccezione in casi molto particolari (ad esempio i mitocondri):
- è perciò definito universale.
Informatica e Bioinformatica – A. A. 2013-2014 16
Le quattro lettere sulla colonna di
sinistra indicano la prima base di ogni
codone, le lettere poste in alto indicano
la seconda base.
All'interno della tabella, accanto ad
ogni codone è indicato l'aminoacido
corrispondente.
Si può vedere che molti aminoacidi
sono codificati da più codoni:
il codice genetico è degenere perché
molti amminoacidi sono codificati da più
di un codone.
Tre codoni codificano lo "STOP",
ovvero la fine della proteina.
Il codone ATG codifica la metionina
ma indica anche l’inizio della regione
codificante la proteina.
Come leggere il codice genetico
Questo significa che, noto un mRNA, potete
in linea di principio ottenere la corrispondente
sequenza amminoacidica!
Informatica e Bioinformatica – A. A. 2013-2014 17
Ad esempio la sequenza:
A C T G T A C C G T T A A G C A T A….
può dare origine a:
ACT GTA CCG TTA AGC ATA…...
CTG TAC CGT TAA GCA TA…..
TGT ACC GTT AAG CAT A….
Data la sequenza di un trascritto esistono tre possibili modi di tradurre tale sequenza
in proteina, a seconda del punto di inizio:
le tre possibilità sono chiamate reading frame (fasi di lettura).
evidenza che la sequenza deve essere codificante
Quando la fase di lettura è costituita esclusivamente da codoni che rappresentano aminoacidi
si parla di open reading frame (ORF).
Una sequenza tradotta in proteina ha una fase di lettura che comincia con un codone di inizio
(in genere AUG) e si estende lungo i codoni finché raggiunge uno dei tre codoni di
terminazione.
In genere, solo una delle tre possibili fasi di lettura è aperta in una data sequenza di DNA.
Una lunga open reading frame è difficile che sia presente per caso. Se non fosse tradotta
non ci sarebbe nulla che impedisce l’accumulo dei codoni di terminazione:
Informatica e Bioinformatica – A. A. 2013-2014 18
Lo sviluppo di tecnologie strumentali sempre più sofisticate ha portato ad una enorme produzione di dati
biologici. Per la gestione di questi dati è quindi necessario disporre di potenti sistemi di archiviazione e
strumenti per accedere alle loro informazioni.
DATABASE BIOLOGICI
Sono essenzialmente dei contenitori ordinati di informazioni costruiti per introdurre e mantenere dati di tipo biologico e permetterne una facile consultazione (query)
Raccolgono informazioni e dati derivati dalla letteratura e da analisi effettuate in laboratorio
oppure attraverso l’applicazione di analisi bioinformatiche o analisi in silico.
Sono generalmente accessibili liberamente e possono essere consultati via web.
Ogni banca dati è caratterizzata da un elemento centrale attorno al quale viene costruita la
entry della banca dati.
Ad esempio, l’elemento centrale per le banche dati di sequenze di acidi nucleici è la
sequenza nucleotidica di DNA o di RNA
Informatica e Bioinformatica – A. A. 2013-2014 19
L’oggetto principale è la ENTRY, una unità riconoscibile grazie ad un identificatore
univoco, che possiede una descrizione organizzata in campi standardizzati riconoscibili
grazie ad HEADERS (“etichette”), univoci nella banca dati; esempio Identificatore,
Autore, Data, ecc.
Organizzazione di un database biologico
Un RECORD biologico
LOCUS un codice
DEFINITION
descrizione della sequenza
ACCESSION un codice
ORGANISM
l’organismo a cui appartiene la
sequenza (e tassonomia)
REFERENCE
Riferimenti bibliografici a quella
sequenza o chi l’ha sottomessa
Informatica e Bioinformatica – A. A. 2013-2014 20
In genere le banche dati presentano 2 versioni delle entries:
Flat-file: un file di testo semplice, formattato, meno “accessibile”
HTML (o XML): interattivo, di facile consultazione
L’interattività ha un ruolo centrale per una banca dati: permette di
navigare facilmente tra le sue entries e quelle di altri database.
Sia i flat-file che le pagine HTML sono ricchi di cross-references, riferimenti che
rimandano ad altre banche dati generiche o specializzate.
Si ottiene così per ogni entry una serie di informazioni spesso ridondanti, tra
cui è bene sapersi orientare, anche perché alcune sembrano in contraddizione.
Esempi:
- una proteina può avere dei riferimenti a sequenze codificanti diverse;
- una entry può avere più nomi per descriverla o corrispondere a più autori.
Informatica e Bioinformatica – A. A. 2013-2014 21
Collegamenti tra i database Caratteristica importante di questi database è il fatto che sono collegati (in modo intricato) tra di loro. Da un record di un database è possibile saltare, mediante un link ipertestuale, ai record ad esso correlati degli altri database integrati nel sistema.
Informatica e Bioinformatica – A. A. 2013-2014 22
Tipologie di interrogazioni delle Banche Dati
RICERCHE TESTUALI (QUERY)
Utilizzano programmi di RETRIEVAL (di ricerca, reperimento dati) per restituire i record di un database che soddisfano i criteri richiesti.
sfruttano una ricerca di tipo letterale ed individuano termini uguali.
Ricordo che i database devono essere tutti standardizzati (tag, separatori, headers, segni di punteggiatura ecc): questo rende rapida la ricerca.
RICERCHE PER SIMILARITÀ (su sequenze nucleotidiche o proteiche)
Restituiscono le sequenze di un database più simili ad una sequenza fornita come query.
Non sono delle vere e proprie query in quanto richiedono l’esecuzione di programmi anche piuttosto complessi (ad esempio BLAST o FASTA).
Domande:
Trovare la sequenza nucleotidica del gene ‘telethonin’ è una ricerca testuale o di similarità?
Identificare in topo il gene omologo alla DHFR umana è una ricerca testuale o di similarità?
Ricercare una sequenza proteica di homo sapiens è una query o una ricerca per similarità?
Informatica e Bioinformatica – A. A. 2013-2014 23
OPERATORI BOOLEANI
Questi potreste già conoscerli dalla matematica!
ATTENZIONE! Oltre a quello
nucleare, esiste anche il genoma
mitocondriale, nei database sono
depositate sequenze derivanti da
entrambe le sorgenti!!!
Informatica e Bioinformatica – A. A. 2013-2014 24
RICERCA BIBLIOGRAFICA
Le modalità con cui si esegue una ricerca bibliografica sono di esempio
per una qualsiasi ricerca testuale o query.
Struttura di un articolo scientifico
- Rivista dove è pubblicato (nome, data di
pubblicazione, volume, pagina )
- Titolo
- Autori
- Abstract (Riassunto dell’articolo)
- Introduzione
- Opzionali: ringraziamenti
- Reference (Bibliografia)
Materiali e metodi
Risultati, discussione, conclusione
Descrizione del lavoro
Informatica e Bioinformatica – A. A. 2013-2014 25
Titolo
Autori
Abstract
(riassunto)
Introduzione Risultati