Metilazione del DNA
description
Transcript of Metilazione del DNA

Metilazione del DNA
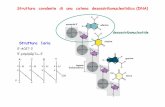
Nei vertebrati la metilazione interessa solamente la
Citosina sul dinucleotide CpG : l’enzima citosina
metiltransferasi aggiunge un gruppo metile al C5 della citosina:il risultato è la 5-
metilcitosina.
Human Molecular Genetics 2

Metilazione del DNA Le sequenze CpG sono sotto-rappresentate nel genoma (probabilmente
per la tendenza della 5-metilcitosina a venire deaminata e mutata in T)
ma abbondanti nelle regioni promotrici dei geni

In cellule non embrionali, 80% dei CpG sono metilati, ad eccezione delle isole CpG dei promotori
Dnmt1 ha particolare affinità per le sequenze emi-metilate: tende quindi a metilare il nuovo filamento che si è formato su uno stampo metilato-> mantenimento del pattern di metilazione

CH3 CH3 CH3 CH3
MeCP2 Repres.
CorepressoriHDAC 1
HDAC 2
Deacetilazione delle lisine di H3 e H4

Metilazione e regolazione genica: Dnmt metilano il DNA Il DNA metilato è legato da proteine che legano il metile
(MeCP2, MBD1-4) Queste a loro volta sono in grado di reclutare diversi HDAC ->
repressione della trascrizione

Inibitori delle HDAC: una nuova terapia anti-cancro
Diverse sostanze che causano differenziamento, arresto della crescita e/o apoptosi di cellule trasformate -> inibitori di HDAC
(es. DMSO, Tricostatina A (TSA), butirrato)
Questi inibitori causano arresto del ciclo cellulare in G1 o G2, differenziamento e/o apoptosi in vitro, documentati in tutti i tipi cellulari trasformati incluse linee cellulari derivate sia da tumori ematologici (leucemie, linfomi, mielomi) che epiteliali (tumore dei polmoni, delle ovaie, del seno, della prostata)
Le concentrazioni biologicamente attive correlano con quelle richieste per causare accumulazione di istoni acetilati

SI IGNORA quali siano gli HDAC la cui attività deve essere bloccata
Gli inibitori causano aumento dell’ acetilazione sugli istoni (H2A, H2B, H3 e H4) anche in cellule normali, ma l’attività di soppressione della crescita sembra essere limitata alle cellule trasformate.
Geni bersaglio: CDKN1A (codificante per l’inibitore dell’attività cinasica ciclina-
dipendente p21/WAF1) (tramite un sito di SP1 sul promotore) CDKN2A (p16) Ciclina E



Ingestione di fibre vegetali non digeribili
Nella parte terminale dell’intestino vengonousate come fonte di energia da batteri simbionti
Formazione di acido butirrico come prodotto
CH3CH2CH2COOH

Costituisce la fonte energetica primaria delle cellule del colon

La metilazione del promotore e' uno dei meccanismi principali coinvolti nell'inattivazione di geni
oncosoppressori
Nel 10% dei tumori colorettali sporadici e’ inattivato il gene responsabile della sintesi di una proteina
addetta al “mismatch repair” (MLH1)
Mismatch repair : riparo di una coppia di basi male appaiataEs. T/G probabilmente a causa di una mutazione

Cellule tumorali del colon mostrano una ipermetilazione delPromotore di MLH1 che e’ cosi inattivo
Trattamento combinato con 5-azadeossicitidina e butirrato
Ipometilazione di MLH1
Espressione del gene che codifica per MLH1
Apoptosi

Normale Tumorale
DAF
DAFDAF
DAF
DAF
La presenza di DAF nelle cellule tumorali delColon rende inefficace il sistema del Complemento

EFFETTO DEL BUTIRRATO SUL mRNA di DAF

EFFETTO DEL BUTIRRATOSULLA PROTEINA DAF

EFFETTO DEL BUTIRRATO SULL’ATTACCO DEL COMPLEMENTO

EFFETTO DEL BUTIRRATO SUL PROMOTORE di DAF

EFFETTO DEL BUTIRRATOSULLA STABILITA’ DEL mRNA di DAF

Cellule tumorali del colon sembrano avere una ipometilazione
Di RB1 (una proteina regolatrice del ciclo cellulare)

Il butirrato aumenta la metilazione diRB1

Quali sono i mediatori?

Kruppel-like factor-4 (KLF4/GKLF) e’ un fattore di trascrizione che mostra proprieta’ simili a quelle del butirrato in cellule del colon
QuickTime™ and aYUV420 codec decompressor
are needed to see this picture.

-Il butirrato aumenta l’mRNA di KLF4
-Questo aumento e’ dovuto ad una maggiore attivita’ del promotore
-HDAC (Istone Deacetilasi) inibisce questo aumento

Fibre alimentari Butirrato Acetilazione del promotore KLF4
KLF4
Apoptosi