La maturazione dell'RNA -...
Transcript of La maturazione dell'RNA -...

T
4213311ESICIEEERE
AAGAAG T ITGGGTGCTTGGAGAC GAG GTCTGCTTTCCAGCAGA AC T AACTT T
EBEEDEECIDDENEEECCEEE% 44k CEREINEEEECIEELICEEEEldA
Lur Le u Leu Gly Asn Met te Val Ile Val Leu Gly Lys Asp Phe Thr Pro Ala Ala Gin Ala Ala Phe Giri Lys Val Val Ala Gly Val Ala Thr Ala Leu Ala His Lys Tyr His TerffintERECEICEMEDIECIIEEEE ENBERECEOCEESECEICIEDEZEHE 7233CEECIEDECEESEICEICEEMICEMCENIZEBEECEEIZEICEEEEICEEDEFECISEIGIEECEENECIETEEMEZZEie
4
ATTTGATGACTGT TTA A T A
pranscsnermacmcznacemorman
NbliCEICIEC3EDECELICIEE12 A ELEEIZZIEEI EICE211212EMICIECEEEECIELICICEINEEZEINEREEIREECINEEDIEREEREEIVA
ATCAGC T T CCCCTCCTC C1T T TIC CTCC CI A G TIC CI T TICIT CITIC TICIT CITIC T T1T CITICIT AA T CCT T TCC T T TCCCTCA T
IliAIAIAITICIAITITITITIAICIGITITIGIAICICITITITIAIAITITITITICIGIAIA TG T T TCT TTCTCCTT T AC
T
A
lawei
mencempogoseacceaard
A T A T T T T A A T A A
A1TT
CT TITAlC!C TIT CIA TIA1T TIAIAf
caamonammanocomememA
1311111MIECIDE 111131111111311111E0BEEMBEZETEMI 111151111D11111
:aUn gene che codifica per una proteina è una sequenza di nucleotidi (A, C, G, T) nel DNA. Lasequenza riportata qui si riferisce al gene per la globina beta nel topo. La globina beta è unadelle due catene amminoacidiche dell'emoglobina. La sequenza effettivamente codificante delDNA è interrotta da due «introni» (in grigio). I tre segmenti di sequenza codificante sono «esoni»(in colore). Gli amminoacidi della globina beta sono indicati con le rispettive sigle vicino ai
corrispondenti nucleotidi. Perché il messaggio genetico possa esseretradotto nella catena amminoacidica della proteina, l'RNA deve venirtrascritto dal DNA. Le sequenze dette introni devono quindi essererimosse in modo che l'RNA messaggero (m-RNA) abbia la giusta se-quenza codificante. Gli introni sono caratteristici dei geni delle cellule
eucarioti, che, diversamente da quelle batteriche, hanno un nucleo bendefinito. I nucleotidi ATG all'inizio della sequenza (in alto a sinistra)sono il segnale di inizio della sintesi proteica. I nucleotidi TAA alla finesono il segnale per la fine della sintesi proteica. La sequenza è statadefinita da Philip Leder e collaboratori alla Harvard Medical School.
Ser Cys Leu Trp Gly Lys Val
p
Val His Leu Thr Asp Ala Glu Lys Ala Ala Val
U
no dei problemi fondamentali del-la biologia molecolare è quellodi scoprire in che modo due cel-
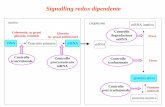
lule con la stessa dotazione di geni possa-no, ciascuna, produrre una differente se-rie di proteine. Con ogni probabilità,tranne poche eccezioni, tutte le cellule diun organismo pluricellulare hanno la stes-sa informazione genetica, codificata nellesequenze di basi nucleotidiche che costi-tuiscono il loro DNA. La sequenza nu-cleotidica nel filamento di DNA funge dainsieme di istruzioni per l'assemblaggiodelle proteine che dirigono tutte le fun-zioni cellulari. Si potrebbe pensare, per-tanto, che tutte le cellule di un organismopluricellulare sintetizzino le stesse pro-teine. Nella realtà, invece, ogni tipo dicellula produce solo una parte caratteri-stica di tutte le proteine codificate nel suoDNA. Come viene controllato, allora,l'apparato genetico di ogni tipo di cellula,perché produca solo una serie specifica diproteine e nelle giuste quantita?
Le cellule degli organismi pluricellularisono eucarioti, cioè hanno un nucleo bendefinito. In esse, la sequenza dei nucleo-tidi all'interno di un gene, cioè il segmen-to di DNA che codifica per una sola pro-teina, viene trascritto in una molecola diRNA, che fuoriesce dal nucleo e passa nelcitoplasma circostante, dove viene tradot-ta in proteina. Così, nella cellula eucario-te, sono possibili parecchi livelli di regola-zione genetica. Potrebbe essere esercitatoun controllo per individuare quali seg-menti del DNA vengono trascritti inRNA, quali sequenze trascritte vengonotrasportate nel citoplasma e la velocitàalla quale viene tradotto un particolareRNA. Da tempo si è dato per scontatoche il controllo della trascrizione e il con-trollo della traduzione svolgano una fun-zione nella regolazione dei geni degli eu-carioti. Tuttavia, ancora nel 1975 la cono-scenza di ciò che avviene nell'RNA tra latrascrizione e la traduzione era lacunosa.
Alla fine degli anni settanta, divenneevidente che sono necessarie parecchieoperazioni significative per trasformare ilprodotto di trascrizione del DNA, l'RNAprimario, o appena formato, nell'RNAmessaggero (m- RNA) maturo, necessa-
rio per la traduzione. Alle due estremitàdel filamento di RNA vengono aggiuntedifferenti strutture. Certi nucleotidi lungola catena subiscono una modificazionechimica. Tuttavia, è stata del massimointeresse la scoperta che il prodotto ditrascrizione, l'RNA primario, viene fre-quentemente tagliato e nuovamente sal-dato per dare un m-RNA più corto. Inalcuni casi, questo RNA può essere ta-gliato e nuovamente saldato in più di unModo e dà origine così a differentim-RNA e quindi a differenti proteine.Questo taglio e successiva saldatura diffe-renziali potrebbero anche essere unaforma di controllo genetico e, in alcunicasi, è stato dimostrato che è proprio così.
Met
Glu Asn Phe Arg
CCTCC
T T
sono quelli dell'adenina, della citosina,della guanina e dell'uracile, le cui abbre-viazioni sono A, C, G e U. (Nel DNA ilposto di U è occupato dalla timina, T.).Ogni nucleotide consta di una base azota-ta e di uno zucchero a cinque atomi dicarbonio. Questi cinque atomi sono ingenere designati con numeri. Ogni coppiadi nucleotidi adiacenti è unita da un grup-po fosfato che forma un legame tra il car-bonio 5' del residuo di zucchero di unnucleotide e il carbonio 3' del residuo dizucchero del nucleotide adiacente. Così lamolecola di RNA ha una sua direzionali-tà: un'estremità è la terminazione 5' el'altra la terminazione 3'.
La trascrizione del DNA in RNA vienerealizzata da una RNA-polimerasi, unodei tre tipi di polimerasi presenti in unacellula eucariote. L'enzima si lega alDNA in corrispondenza di un giusto sito«di inizio» per l'RNA e sceglie il primonucleotide, che diventa l'estremità 5' delfilamento di RNA. Quindi si sposta rapi-damente lungo il filamento di DNA, ag-giungendo i giusti nucleotidi al filamentodi RNA. L'RNA poi, appena formato, sisepara dal DNA: per prima si stacca l'e-stremità 5' e il complesso della molecolanon si allontana fino a quando non si èformata anche l'estremità 3'.
Quando la RNA-polimerasi copia untratto di filamento del DNA, il nucleo-tide che viene aggiunto all'RNA è com-plementare a quello presente sul DNA. Acausa della struttura molecolare dei nu-cleotidi. ciascuno di essi può formare le-gami a idrogeno con uno solo degli altritre. Così C e G possono formare legami a
idrogeno solo tra loro, eA e U (o T) lostesso. Sono proprio questi legami a idro-geno tra basi (o meglio nucleotidi) com-plementari che fanno sì che i due filamen-ti di DNA formino una doppia elica. Ana-logamente, un segmento di RNA puòformare con un segmento di DNA unamolecola a duplice filamento se le basi suidue filamenti sono complementari.
Pertanto, l'RNA trascritto è comple-mentare al filamento di DNA da cui pro-viene. Poiché conserva l'informazione diquella parte del genoma (l'intero com-plemento d'informazione genetica), essopuò incorporare il codice per la catena diunità amminoacidiche che costituisce unaproteina. Il codice viene letto sull'RNA atre nucleotidi alla volta e ogni tripletta dinucleotidi. che codifica per uno dei 20diversi amminoacidi, è chiamata codone.Nel processo di traduzione, la lettura delcodone porta all'aggiunta del corrispon-dente amminoacido alla molecola protei-ca che si sta formando.
I tre RNA
L'RNA messaggero, l' RNA che ha unafunzione codificatrice nei riguardi delleproteine, non è il solo tipo di RNA. Ve nesono perlomeno due altri tipi principali,ognuno dei quali ha una funzione signifi-cativa nella sintesi proteica. L'RNA ribo-somale (r-RNA) è un componente delribosoma, l'organello citoplasmatico chefunge da piattaforma per la traduzione. Ilribosoma consta di due subunità, designa-te come grande e piccola: ogni subunitàincorpora una molecola di r-RNA e tra 25
e 50 molecole proteiche. L'RNA di tra-sporto (t-RNA) funziona come un uncinomolecolare per attaccare l'amminoacidosuccessivo alla fine di un filamento pro-teico; ciascuno dei diversi t-RNA ricono-sce un particolare amminoacido.
Sul ribosoma, l'RNA di trasporto vienea trovarsi assieme all'RNA messaggero eagli enzimi, che servono a connettere ilgiusto amminoacido alla molecola protei-ca in via di formazione e a far progredirel'intero apparato per leggere il codonesuccessivo. Così, nella sintesi di una pro-teina, il t-RNA e l'r-RNA hanno ruoliripetitivi che sono realizzati seguendo leistruzioni dell'm-RNA. Nelle celluleumane, vi è all'incirca un milione di copiedi ogni tipo di t-RNA assieme a circacinque milioni di copie di ciascuno dei duetipi principali di r-RNA: una per ogniribosoma presente nella cellula.
A causa della loro abbondanza, gliRNA di trasporto e gli RNA ribosomalisono più facili da studiare degli RNAmessaggeri ed erano già stati intensiva-mente analizzati prima che si fosse rag-giunta una buona conoscenza anche del-l'm-RNA. Il primo esempio di matura-zione dell'RNA è stato trovato proprionell'RNA ribosomale. Nel 1961, KlausScherrer e Harriet Latham hanno colla-borato con me a una ricerca condotta inun nuovo laboratorio per lo Studio dellacellula animale e dei virus animali pres-so il Massachusetts Institute of Techno-logy. Abbiamo cominciato ad analizzarel'RNA di cellule umane, sviluppatesi suun terreno colturale. Volevamo, in par-ticolare, confrontare gli RNA appena
La maturazione dell'RNAIl DNA viene trascritto in RNA messaggero, poi tradotto in proteina.Nelle cellule dotate di nucleo, tra cui le cellule umane, molti eventisi svolgono nell'RNA nel periodo tra la trascrizione e la traduzione
di James E. Darnell, Jr.
È probabile che il processo di maturazio-ne dell'RNA, successivo alla trascrizione,sia necessario per molti m-RNA, ma chesia una forma di regolazione solo per po-chi. La sua scoperta ha nondimeno ag-giunto una nuova dimensione al quadrodell'espressione genica, il che è in sé unpasso avanti significativo. Inoltre ha per-messo di formulare interessanti ipotesisulla primissima storia dei geni e dellecellule.
La trascrizione del DNA
Come il DNA, l'RNA è una catena po-limera, le cui unità monomere sono nu-cleotidi. Nell'RNA i quattro nucleotidi
A TGGTGCACCT GACTGATGC TGAGAAGGCTGC T G T T GCCTGTGGGGAAAGGTG T TiGGTGGTGAGGCCCTGGGC AGAI BEICEEEEEIDEEEXECEEREEDEICIECCEISEElinEEMEIDEDIEEEIDEIEEDEGIEWCAVal Gly Gly Glu Ala Leu Gly Arg Leu Leu Val Val Tyr Pro Trp Thr Gin Arg Tyr Phe Asp Ser Phe Gly Asp Leu
SernailiEXEMEEICEENEICELELIEUEEESEEEEMECEIDEEECErle nririEEDIERTIMEED3ECCEDRECEERTECUPMECEEICEDEICITEEECIDECC313122151EIZEEEEGIDIENECDEVEMEIMEE EILEIECCEEMEEIZEICIEUF
,sp Va .His L Lys Giu S i i Ala Phe Thr Ciy Lys H --: tV, Asp Asn Phe T r Ile Vai Lys' s HisPro
Lys Ala Asn Gly Met q P Ala Ser Ada
22 23

RNA, PRODOTTOPRIMARIODI TRASCRIZIONE
TRASCRIZIONE
MATURAZIONE DELL'RNA
V
m-RNA MATURO
TRASPORTO NEL CITOPLASMA
CATENADI AMMINOACIDI
RIBOSOMA
TRADUZIONE
v/ 7-\
La regolazione genetica in una cellula eucariote avviene a parecchi livelli. Nel nucleo il DNA vienedapprima trascritto in un RNA primario, che è una copia complementare di un segmento di DNA.Questo prodotto di trascrizione primario viene quindi trasformato in m-RNA maturo: cioè l'RNAmessaggero al quale sono state asportate le sequenze non codificanti. L'm-RNA è poi trasportatodal nucleo al citoplasma, dove viene tradotto dai ribosomi nella catena amminoacidica di unaproteina. L'espressione dei geni può essere controllata durante la trascrizione e la maturazione nelnucleo, durante la traduzione nel citoplasma e durante il trasporto dal nucleo al citoplasma.
C A T T A C A
RNA-POLIMERASI
3
trascritti nella cellula con i tipi di RNAstabile presenti in grandi quantità: it-RNA e i due tipi di r-RNA.
I batteri sono procarioti, cioè le lorocellule non hanno un nucleo ben definito.In esse la trascrizione e la traduzione sisvolgono simultaneamente: l'RNA mes-saggero comincia a essere tradotto men-tre è ancora in fase di trascrizione sullamolecola di DNA. Come le cellule euca-rioti, tuttavia, le cellule batteriche dipen-dono dai ribosomi e dai t- RNA; pertanto,buona parte dei primi lavori sui tre tipistabili di RNA fu compiuta con cellule diquesto genere.
I tre tipi di RNA stabile e abbondantepossono venir distinti sulla base delle lorodimensioni. Tutti i t-RNA sono piccoli:sono lunghi all'incirca 80 nucleotidi. I dueprincipali tipi di RNA ribosomale sono,invece, sostanzialmente più grossi. Ladimensione di una molecola di RNA simisura spesso in base alla velocità con cuila molecola precipita quando viene cen-trifugata a velocità elevata in un'ultracen-
trifuga. Questo metodo si chiama analisidella sedimentazione zonale e la velocitàdi sedimentazione si esprime in unitàSvedberg, abbreviate con S. Quanto piùlungo è l'RNA, tanto più elevato è ilnumero di S. Le due forme principali dir-RNA nelle cellule eucarioti vengonodesignate come 18S e 28S.
Nei nostri primi esperimenti, Scherrer.Latham e io abbiamo cercato di trovare sei prodotti iniziali, nella trascrizione del-FRNA, hanno le stesse dimensioni dei tipiabbondanti di RNA o, alternativamente,se l'RNA viene trascritto in unità chesono più lunghe o più corte delle molecoleabbondanti. I n ucleosidi, cioè i precursoridei nucleotidi, possono essere marcaticon vari atomi radioattivi. Se alle cellulein accrescimento sono forniti nucleosidiradioattivi, le subunità marcate entreran-no nei pool di nucleotidi che forniscono ilmateriale costruttivo per i filamenti diRNA. Le molecole radioattive sarannoquindi aggiunte a nuovi filamenti di RNAper opera delle RNA-polimerasi. La
marcatura offre un mezzo per identificarel'RNA appena formato: dopo che gliRNA marcati sono stati estratti dalla cel-lula, possono venir separati in base allaloro dimensione, analizzandone la sedi-mentazione, e confrontati con RNA nonmarcato, già nella cellula in precedenza.
La maturazione dell ir-RNA e del t-RNA
Inizialmente le cellule hanno ricevuto inucleosidi marcati per soli cinque minuti.Dopodiché, la radioattività non è com-parsa nell'RNA con le dimensioni corri-spondenti ai tre preesistenti tipi stabili.Essa è comparsa, invece, nel nucleo dellecellule in un gruppo assortito di molecolecon un'ampia gamma di dimensioni. Mol-te molecole marcate erano parecchio piùlunghe anche del più lungo RNA riboso-male, che consta di 5000 nucleotidi.Come vedremo, tali grosse molecole, chesi marcavano rapidamente, hanno dimo-strato di avere un notevole significato nel-l'attività dell'RNA messaggero. I prodot-ti di trascrizione che erano rimasti marcatientro i cinque minuti erano di tante di-mensioni diverse da non poter essere in-clusi in poche categorie sulla base dellaloro dimensione. Quando il tempo duran-te il quale i nucleosidi marcati venivanoforniti alla cellula fu aumentato da cinquea 60 minuti, gruppi distinti di molecolemarcate e dotate di differenti dimensionirisultarono, invece, evidenti. Una dellemolecole più grosse, che comparve dopo25-30 minuti di marcatura, aveva un va-lore di sedimentazione di 45S.
La composizione in basi dell'RN A 45S(cioè le quantità relative di A, C, G e U)era molto simile a quella degli RNA 18S e28S. La molecola 45S risultò presto corre-lata con gli RNA ribosomali in un mododiverso ed estremamente interessante. Latrascrizione può essere interrotta rapi-damente per opera dell'antibiotico acti-nomicina, che si lega strettamente alDNA. Abbiamo fornito l'antibiotico allecellule dopo che il marcatore era già statoassorbito dalla molecola 45S. A questopunto dal DNA non veniva più trascrittoRNA; pertanto il marcatore si trovavasolo in quelle molecole che erano statetrascritte in precedenza. Poco dopo lasomministrazione dell'actinomicina essoscompariva dalle molecole 45S e ricom-pariva negli RNA 18S e 28S.
Abbiamo avanzato l'ipotesi che gliRNA 45S siano un lungo prodotto prima-rio di trascrizione che viene tagliato perdare i due RNA ribosomali finiti. Da allo-ra essa è stata convalidata da molti espe-rimenti. Metodi perfezionati di definizio-ne delle sequenze nucleotidiche hannoreso possibile la determinazione dellesequenze di basi in ambedue i geni perl'RNA ribosomale e per gli r-RNA stessi.È evidente oggi che in molti tipi di celluleeucarioti ivi incluse quelle di lievito, dellepiante, delle rane e dei topi, un lungoprecursore dell'r-RNA viene tagliato efornisce molecole finite più corte. Espe-rimenti recenti hanno mostrato che que-sto accade perfino nei batteri.
Alla fine degli anni sessanta, inoltre,
Roy H. Burdon dell'Università di Gla-sgow e Deborah Bernhardt nel mio labo-ratorio al MIT hanno trovato che anchel'RNA di trasporto si forma in seguito aun processo di maturazione. Nel t-RNA,però, la differenza di lunghezza tra pro-dotto di trascrizione primario e molecolafinita è solo tra 20 e 30 basi: un precurso-re di circa 100 nucleotidi viene accorciatoe dà il prodotto finito che consta di 80nucleotidi.
All'inizio degli anni settanta, in moltitipi di cellule il processo di maturazionedell'RNA ribosomale e dell'RNA di tra-sporto era stato ormai stabilito in manieradefinitiva. Si delineò a quel punto la que-stione se anche l'RNA messaggero fossefatto maturare in modo analogo. Sembra-va improbabile che esso lo fosse nelle cel-lule procarioti, visto che nei procarioti iribosomi traducono l'RNA messaggeromentre è ancora in fase di trascrizione.Negli eucarioti, d'altra parte, dove la tra-scrizione e la traduzione sono separatenel tempo e nello spazio, la maturazionedell'm-RNA era un evento ben distinto einteressante.
Gli RNA nel poliribosoma
Molte più cose andavano tuttavia 'tro-vate a proposito dell'RNA messaggeroprima che il problema della sua matura-zione potesse essere affrontato diretta-mente. Quando, negli anni sessanta, ven-ne scoperto il processo di maturazionedell'RNA ribosomale, l'RNA messagge-ro delle cellule eucarioti non era statoancora isolato. Era già stato dimostratoperò che gli amminoacidi sono congiuntiin proteine nei poliribosomi (o polisomi),grappoli di ribosomi presenti nel citopla-sma. Si presunse, dunque, che l'RN Amessaggero fosse presente in essi, masenza ancora disporre di prove.
In un tentativo di trovare l'ipoteticomessaggero, Sheldon Penman, YechielBecker e Scherrer nel mio laboratorio alMIT hanno coltivato le cellule tumoraliumane, chiamate cellule HeLa, in presen-za di nucleosidi marcati, i quali venivanoincorporati nell'RNA; in seguito, questoRNA marcato era estratto dai poliribo-somi. Una parte dell'RNA estratto eranaturalmente RNA ribosomale, ma que-sto dopo una breve esposizione ai precur-sori radioattivi non era rimasto marcato.Vi era, invece, un altro gruppo di moleco-le di RNA nei poliribosomi che era statomarcato rapidamente: queste molecoleavevano una composizione in basi diversada quella dell'r-RNA. Gli RNA poliribo-somali, come vennero chiamati, avevanouna lunghezza variabile tra 500 e 3000nucleotidi. Abbiamo avanzato l'ipotesiche ciascun RNA poliribosomale inclu-desse una varietà di tutti gli RNA mes-saggeri presenti nella cellula.
La composizione in basi degli RNApoliribosomali è stata confrontata conquella delle lunghe molecole trovate nelnucleo durante la ricerca sul processo dimaturazione dell'RNA ribosomale. L'in-sieme di RNA non ribosomali marcatirapidamente era stato denominato RNA
nucleare eterogeneo (hn-RNA) e non fudifficile dimostrare che tale RNA avevauna composizione in basi molto simile aquella dell'RNA poliribosomale, breve-mente marcato. Sembrava dunque chel'hn-RNA potesse essere un precursoredella molecola di messaggero utilizzatanella traduzione che aveva luogo nei poli-ribosomi.
Il guaio con la suddetta ipotesi era cheuna parte degli hn-RNA aveva 5000 nu-cleotidi o più, mentre i più lunghi fra gliRNA poliribosomali avevano una esten-sione di soli 3000 nucleotidi. Tenendopresente l'esempio della maturazione delt-RNA e dell'r-RNA, abbiamo suggeritoche l'hn-RNA venisse accorciato nel nu-cleo in un processo di maturazione, primadi essere trasportato nel citoplasma. Tut-tavia, non fu possibile per alcuni anniprovare questa possibilità in modo diret-to. La ragione è che le cellule eucariotisintetizzano migliaia di tipi di RNA mes-saggero e. dato che non era stato isolatoalcun m- RNA singolo, non fu possibileosservare la sintesi di una molecola dim-RNA per dire se era stata o no sottopo-sta a un processo di maturazione.
Un modo per aggirare questa difficoltàconsiste nel basarsi sulla semplicità degliorganismi molto meno organizzati dellecellule, e cioè i virus. Pertanto ci siamorivolti allo studio di cellule eucarioti infet-tate da virus. L'infezione di una cellulaanimale da parte di un virus è parsa uneccellente sistema sperimentale per stu-diare le proprietà biochimiche dell'RNAmessaggero e. in ultima analisi, la sua sin-tesi. Il genoma di un virus, che è contenu-to nella parte centrale della particella vi-rale, è una molecola di DNA o di RNA.Quando il genoma virale entra nella cellu-la ospite, usurpa l'apparato sintetico dellacellula in modo che essa sintetizzi gli acidinucleici e le proteine virali. Alla fine delprocesso infettivo, nuove molecole viralisi combinano nelle particelle virali chepossono infettare altre cellule. In con-fronto alla cellula ospite di mammifero, ilvirus ha poche proteine e, pertanto, pochiRNA messaggeri. Questo fatto semplificale cose. Dato che I'm-RNA virale deveessere tradotto dai ribosomi e dai t- RN Adell'ospite, esso può essere esaminatoquasi come se fosse I'm-RNA di una cellu-la eucariote. Inoltre, alcuni virus a DNApenetrano nel nucleo cellulare, dove sisvolge la sintesi enzimatica dell'm- RNAvirale; pertanto gli stadi nella produzionedi RNA possono anche essere studiatimediante un virus.
Il virus come strumento
La semplicità del virus venne sfruttatainizialmente per dimostrare che l'RN Apoliribosomale che si marca in breve tem-po è RNA messaggero. Penman e Beckernel mio laboratorio hanno trovato che nel-le cellule infettate con il virus della polio-mielite, che è un virus a RNA, questoRNA ha preso il posto nei ribosomi del-l'RNA marcato in breve tempo. Parecchiricercatori hanno quindi utilizzato un se-condo metodo per dimostrare che I'm-
1
5
C A
T T A i C
A 3'
3 O T C A A T
C T 5'
FILAMENTO DI DNA
2
RNA TRASCRITTO
3
C A
T T A C
A
3
5'
4
5'
C A Gj T A C A
3'
O T A T C T
C A U A C A
La trascrizione del DNA in RNA conserval'informazione genetica codificata nel DNA.Ogni nucleotide di quest'ultimo può formarelegami a idrogeno con un altro nucleotide sol-tanto, che viene detto il suo complementare:l'A (il nucleotide adenilico) si appaia con T (ilnucleotide titnilico) e C (il nucleotide citosili-co) con G (il nucleotide guanilico). L'elica diDNA consta di due filamenti complementaritenuti uniti da tali legami (1). L'RNA è unfilamento singolo costituito da nucleotidi e incui T è sostituito da U (nucleotide uracilico). Ifilamenti sia di DNA sia di RNA sono direzio-nali: un'estremità viene indicata con 5' e l'altracon 3'. Un filamento di DNA viene trascritto inRNA dall'enzima RNA-polimerasi (2). Que-sto si sposta lungo un filamento del DNA, leg-gendo i nucleotidi uno alla volta e aggiungendoil nucleotide complementare a un filamento diRNA che si allunga dall'estremità 5' alla 3' (3).Quando quest'ultima si è completata, il pro-dotto di trascrizione si separa dal DNA (4).
24
25

RIBOSOMA
AMMINOACIDOPROTEINA
PICCOLA SUBUNITA
1
t RNA
ANTICODONE
< r-RNA A G C A G UUA1CG;1/41
CODONE
m-RNA
PROTEINA
GRANDE SUBUNITA
25-30 MINUTI
3
RNA28S
RNA18S
DIVERSIt-RNA111111
• le•
I.• •15\
PRECURSORIRADIOATTIVI DELL'RNA
2
H-5
OEo
CITOPLASMAoo
O
RNACITOPLASMATICO
NUCLEOLORNA NUCLEOLARE
NUCLEO
RNANUCLEARE
GRANDE < > PICCOLA
5 MINUTI RNA MARCATO
RNA
45S
GAG CA G U U A C GA UCA
La traduzione dell'RNA in proteina richiede due tipi di RNA oltre almessaggero che porta l'informazione genetica. Gli RNA ribosomali(r-RNA) sono componenti strutturali del ribosoma (1). Gli RNA ditrasporto (t-RNA) fungono da uncino per aggiungere un nuovo ammi-noacido alla catena proteica in formazione; ogni t-RNA è specifico perun dato amminoacido. Ogni amminoacido nella proteina è specificatoda un codone, sequenza di tre nucleotidi nel filamento dell'RNA mes-
saggero. La molecola di t-RNA si lega all'm-RNA mediante un antico-done, gruppo di tre nucleotidi complementare al codone (2). Quando ilribosoma si sposta lungo la molecola del messaggero, l'anticodonericonosce il codone e forma con esso legami a idrogeno; l'amminoacidoall'altra estremità del t-RNA si attacca alla catena proteica. Quando ilribosoma si sposta sul codone successivo, il t-RNA viene estromesso esostituito da un altro t-RNA che corrisponde al nuovo codone (3).
28S
18S
-RNA virale penetra nei poliribosomi.Tale metodo è chiamato ibridazione mo-lecolare e fu impiegato per la prima voltanei virus a DNA che infettano i batteri daSol Spiegelman e Benjamin D. Hall del-l'Università dell'Illinois. L'ibridazionemolecolare si basa sul fatto che i filamentidi RNA e di DNA possono ibridarsi, ov-vero formare una struttura a doppio fila-mento, solamente se le loro sequenze nu-cleotidiche sono complementari. Se siforma un ibrido, si può concludere chel'RNA, in origine, è stato trascritto a par-tire da quel DNA.
L'RNA estratto dai poliribosomi di cel-lule infettate da virus a DNA è stato me-scolato con il DNA del virus e della cellu-la ospite. Fatto questo, l'RNA poliribo-somale si è ibridato con il DNA del virusinfettante, ma non con quello del mammi-fero ospite o di altri virus. Brian R.McAuslan della Princeton University hacompiuto un passo in più, mostrando chela struttura centrale del virus del vaiolobovino, un grosso virus a DNA, può sinte-tizzare l'RNA da precursori nucleotidici.L'ibridazione molecolare venne impiega-ta per dimostrare che l'RNA sintetizzatodalla porzione centrale del virus era lostesso dell' RNA poliribosomale presentenelle cellule infettate con il virus del vaio-lo bovino. L'RNA poliribosomale era sta-to pertanto identificato in maniera incon-trovertibile come RNA messaggero: sisapeva che era presente in corrisponden-za del sito di traduzione ed era stato ap-paiato a filamenti complementari di DNApresenti nel genoma virale.
L'identificazione del messaggero hacostituito un decisivo progresso. Studian-do gli m-RN A virali è stato possibile se-
guire la sintesi di specifiche molecole dimessaggero per vedere quali operazionisono eseguite sul prodotto di trascrizione.Sono stati rapidamente identificati parec-chi tipi significativi di maturazione del-l'RN A messaggero. Joseph R. Kates del-l'Università del Colorado a Boulder hanotato che un segmento di ogni m- RNAsintetizzato dal virus del vaiolo bovino èricco di nucleotidi dell'adenina (A). Me-diante analisi chimica, Kates ha dimostra-to che tale segmento si trova in corrispon-denza dell'estremità 3' del messaggero.Poco dopo, quattro gruppi sotto la guidadi Kates, di Mary Edmonds dell'Universi-tà di Pittsburgh, di George Brawermandella Tufts University e mia hanno dimo-strato indipendentemente l'uno dall'altroche quasi tutti gli m-RNA poliribosomalidi cellule animali hanno una sequenza di150-200 nucleotidi adenilici A attaccatiall'estremità 3'. Tale sequenza non è pre-sente, invece, negli RNA di trasporto oribosomali.
La «coda» di nucleotidi adenilici in cor-rispondenza dell'estremità 3' dell'RNAmessaggero è stata denominata sequenzapoli-A e il processo nel quale essa vieneaggiunta è detto poliadenilazione. Si saoggi che, net- nucleo cellulare, la coda è.aggiunta all'hn-RNA (RNA nucleareeterogeneo) per opera di un enzima quasisubito dopo che l'hn-RNA è stato tra-scritto. Anche se la sequenza di eventimolecolari nella poliadenilazione non èstata ancora ricostruita in provetta, si èaccertato che la RNA-polimerasi procedeben oltre il sito della sequenza poli-A:cioè la sede, sul DNA, che corrispondeall'estremità 3' dell'hn-RNA dove vieneaggiunta la coda. Il filamento di RNA
AAU
viene tagliato circa 20 nucleotidi dopouna sequenza segnale e parte di tale se-gnale è il gruppo AAUAAA, che è statotrovato in più di 100 m-RNA vegetali eanimali. L'estremità 3' libera viene quindiallungata per aggiunta di circa 200 nu-cleotidi A e ciò grazie all'intervento di unenzima scoperto dalla Edmonds.
Teste e code
Quasi tutti gli RNA messaggeri chevengono trasportati verso il citoplasmahanno una coda poli-A. Tuttavia, unm-RNA non deve necessariamente avereuna coda poli-A per essere tradotto. Lafunzione della coda sembra essere quelladi proteggere l'm-RNA dalla demolizio-ne operata dagli enzimi presenti nel cito-plasma. Gli RNA messaggeri senza codapoli-A durano solo alcuni minuti nel cito-plasma, mentre quelli con la coda posso-no essere stabili per ore o per giorni. Gliunici m-RNA noti che entrano nel cito-plasma senza una coda poli-A sono quelliche vengono tradotti in quelle piccoleproteine che si chiamano istoni e che silegano al DNA. Questi m-RNA per gliistoni, designati come poli-A durano nelcitoplasma meno di 30 minuti.
Oltre al suo interesse teorico, la se-quenza poli-A ha avuto un notevole effet-to pratico sulle ricerche relative al proces-so di maturazione dell'RNA messaggero.La ragione è che una molecola di m-RNApuò essere afferrata per la sua coda po-li-A . Ciò avviene sintetizzando un omo-polimero polinucleotidico, cioè un fila-mento costituito da un solo tipo di nucleo-tide. Una colonna verticale di vetro vieneriempita di fibre di cellulosa che hanno,
aderente a esse. tale polimero; viene ge-neralmente utilizzato un omopolimeroconsistente o di tutti T o di tutti U. Vienequindi fatto passare attraverso la colonnal'intero corredo di RNA della cellula. Lesequenze poli-A alla fine degli m-RNApossono ibridarsi con gli omopolimeri erimanere nella colonna. L'RNA senzapoli-A, principalmente RNA di trasportoe ribosomale, viene eliminato. Questatecnica, chiamata cromatografia per affi-nità, è stata il primo mezzo pratico diisolamento in forma chimicamente puradell'm-RNA di una cellula eucariote.
La disponibilità di m-RNA purificatoha dato un notevole impulso alle ricerchesulla maturazione dell'RNA. Sono stati inbreve tempo trovati due altri procedi-menti. Nel 1974 Robert P. Perry dell'In-stitute for Cancer Research ha scopertoche l'm-RNA della cellula animale non ècomposto solamente dei quattro nucleo-tidi regolari. Alcuni dei nucleotidi presen-ti nel filamento sono metilati, cioè presen-tano gruppi metilici supplementari (CH3)che sono stati aggiunti a essi. BernardMoss del National Institute of Allergyand Infectious Diseases, Aaron J. Shatkindel Roche Institute of Molecular Biologye Kin-Ichiro Miura dell'Istituto nazionaledi genetica del Giappone hanno trovatoche anche alcuni nucleotidi degli m-RNAvirali sono metilati.
Gli enzimi chiamati ribonucleasi sonostati utilizzati per determinare dove sitrovano, nell'RNA messaggero, le strut-ture metilate più significative. Le ribonu-cleasi scindono i legami tra nucleotidiadiacenti del filamento di RNA: ne risultaun insieme di singoli nucleotidi, ognunocon un gruppo fosfato attaccato. Invece,quando le ribonucleasi vennero utilizzateper l'RNA virale metilato, è stata trovatauna struttura complessa che includevaparecchi gruppi fosfato, in aggiunta aisingoli nucleotidi.
Sulla struttura resistente alle ribonu-cleasi sono stati realizzati molti test chi-mici e oggi se ne conosce la costituzione.Tale struttura, detta «testa» in contrap-posizione a «coda», e da alcuni «cappuc-cio» (cap), si trova all'estremità 5' di tuttigli m-RNA che sono stati osservati nellecellule eucariote, a eccezione di unm-RNA di un unico virus. La componen-te più caratteristica della testa è un nu-cleotide guanilico (G), al quale è statoaggiunto un gruppo metilico. Il nucleoti-de guanilico viene unito all'estremità 5'dell'RNA mediante un legame che inclu-de tre gruppi fosfato.
Alcuni insegnamenti dall'adenovirus
Il gruppo di Perry. quello di Shatkin e ilmio (oggi alla Rockefeller University)hanno cercato di determinare se la testaviene aggiunta all'interno del nucleo.come avevamo fatto quando si era trovatoche la coda poli-A era una componentecomune degli m- RNA cellulari. Abbiamocosì scoperto che le teste sono aggiunteall'estremità libera 5' del filamento diRNA che si sta allungando a opera dienzimi presenti nel nucleo prima che la
4 RIFORNIMENTO DI ACTINOMICINA
••
La dimostrazione di un processo di maturazione si è avuta per la prima volta con l'r-RNA. Nellacellula di mammifero la maggior parte dell'RNA stabile si trova nel citoplasma; una quantitàmolto più piccola di esso è presente, invece, nel nucleolo, un organello del nucleo. La dimensionedi una molecola di RNA si misura in base alla velocità con cui precipita in una provetta quandoviene centrifugata ad alta velocità; l'unità di grandezza determinata in questo modo è lo Sv edberg(S). I tre tipi di RNA stabile sono gli r-RNA 185 e 285 e i più piccoli t-RNA. Negli esperimenti, iprecursori nucleotidici radioattivi possono venir incorporati in nuovo RNA e le molecole marcatepossono dare informazioni sulla dimensione dell'RNA trascritto. Negli esperimenti sulla matura-zione dell'RNA, cellule di mammifero sono state messe in presenza di precursori radioattivi (1).Dopo cinque minuti questi marcatori sono comparsi in RNA di ,aria lunghezza nel nucleo (2).Dopo 25-30 minuti, buona parte del marcatore è stata trovata in una molecola nucleare delledimensioni di 45S (3). L'antibiotico actinomicina, che arresta la sintesi dell'RNA, è stato sommini-strato alle cellule marcate (4): a questo punto il marcatore è scomparso dalle molecole 45S ed ècomparso, invece, nel citoplasma nelle molecole 18S e 28S. Pertanto l'RNA 45S è risultato unprecursore che viene accorciato per dare, prima del trasporto nel citoplasma, i tipi stabili di r-RNA.
26 27

SITO DI SCISSIONE
vV
m' G-P-P-P < >
Una «coda» e una «testa» sono aggiunte all'RNA trascritto dal DNAnelle prime fasi di maturazione dell'm-RNA. Quando la RNA-poli-merasi ha trascritto solo circa 20 nucleotidi, gli enzimi presenti nelnucleo aggiungono una struttura molecolare all'estremità 5' libera delprodotto di trascrizione (1, 2). Questa testa consiste di una guanina(G) modificata, attaccata al prodotto suddetto mediante un legameche include tre gruppi fosfato (P). Allorché la polimerasi si sposta
più lontano lungo il DNA, trascrive la sequenza AAUAAA, che svolgeun ruolo nell'aggiunta della serie di nucleotidi A, chiamata poli-A(3). Il prodotto di trascrizione viene tagliato circa 20 basi dopo lasequenza AA UAAA (4). Lo stesso enzima, o un altro, aggiunge quindii 150-200 nucleotidi A del poli-A all'estremità 3' della catena (5).In fasi successive del processo di maturazione, le strutture terminali siconservano, mentre la parte interna dell'RNA trascritto si modifica.
A A A AA AA A A AAA A
3
m 7 G-P-P-P
m 7 G-P-P-P
cb,m 7 G-P-P-P
AAl A AAAAAA A AAIAIA1A AIAIA**, P1Pm' G-P-P-P
2 ENZIMA
polimerasi abbia trascritto una ventinacirca di basi. Il gruppo di Moss, invece, haisolato di recente l'enzima che aggiunge latesta. A differenza della coda poli-A,questa viene aggiunta a tutti gli m- RNAtrascritti. La funzione della testa non ècompletamente chiara, ma sembra che
essa serva a promuovere la traduzione.Dato che viene aggiunta a tutti glim-RNA, non pare che l'addizione possaessere una forma significativa di regola-zione genetica.
La scoperta della poliadenilazione edell'aggiunta della testa ha indirizzato di
nuovo la nostra attenzione alla relazioneesistente tra l'hn-RNA e I'm-RNA. Sisapeva già che i due tipi di molecola han-no una composizione in basi simile.Quando l'hn-RNA e l'm- RNA poliribo-somale vennero purificati mediante cro-matografia per affinità, si trovò che in
3'
28S
ambedue i gruppi le molecole avevanouna testa 5' e una coda poli-A 3'. Pertantose l'hn-RNA è il precursore dell'm--RNA, le estremità della molecola ven-gono preservate nel corso della matura-zione e del trasporto nel citoplasma. Però,gli hn-RNA erano in media cinque voltepiù lunghi degli m-RNA poliribosomali.Una possibile spiegazione di questa diffe-renza sembra intuitivamente ovvia: unsegmento potrebbe essere tagliato dalcentro dell'hn-RNA e ciò che resta dellamolecola potrebbe poi venir saldato dinuovo lasciando intatte le estremità. Tut-tavia, nel 1976, l'idea sembrava di diffici-le realizzazione o addirittura irrealizzabi-le agli esperti di biologia molecolare.
In meno di due anni, invece, risultò chetale lontana possibilità aveva effettiva-mente luogo in una cellula eucariote.Buona parte delle ricerche effettuate perdimostrare che l'hn-RNA viene accorcia-to per diventare m- RNA ha utilizzatocome parte del sistema sperimentale l'a-denovirus, uno dei molti virus che puòprovocare infezioni delle vie respiratoriesuperiori nell'uomo. Il decorso dell'infe-zione da adenovirus è stato studiato afondo. In particolare sono oggi ben com-prese la collocazione cronologica delladuplicazione del DNA virale nel nucleodella cellula, la composizione delle pro-teine virali e la sequenza secondo la qualele proteine sono prodotte. Pertanto l'a-denovirus risultò un buon organismo dastudiare per trovare in che modo l'RN Aspecifico del virus viene sintetizzato nelnucleo cellulare e fatto maturare per ot-tenere m-RNA.
Il genoma dell'adenovirus è una mole-cola di DNA a duplice filamento, di lun-ghezza pari a 36 000 nucleotidi . La posi-zione di una particolare sequenza di nu-cleotidi nel genoma virale è generalmentedescritta in base a una mappa lineare di100 unità in cui ogni unità corrisponde a360 nucleotidi e dove O si riferisce all'e-stremità sinistra della mappa e 100 alladestra. Da otto a dieci ore dopo che laparticella virale è penetrata in una cellulaumana, la cellula comincia a produrrecopie del DNA virale. Le proteine viralivengono classificate in rapporto al tempodella duplicazione del DNA: quelle «pre-coci» sono prodotte prima che il DNA siduplichi; il piccolo gruppo di quelle «in-termedie» all'incirca nello stesso periodoin cui si duplica in DNA; infine, le «tardi-ve» principalmente dopo che la duplica-zione si è messa in corso. La maggior par-te delle proteine che formano la strutturadella particella virale sono proteine tardi-ve; tra di esse vi sono le proteine degliesoni, le più abbondanti nell'involucroesterno (o capside) del virus.
Le ricerche sul processo di maturazionedell'RNA messaggero si sono concentra-te sulle proteine tardive dell'adenovirus.Il lavoro sperimentale si è basato moltointensamente sulle endonucleasi di restri-zione, un gruppo di enzimi che ha avutouna profonda influenza sulla biologiamolecolare quando, negli anni settanta, èstato scoperto. Ogni endonucleasi di re-strizione taglia il DNA o l'RNA in corri-
spondenza di una specifica sequenza diquattro-sei nucleotidi. In un particolaregenoma, i siti di restrizione, cioè i punti incui l'enzima taglia il DNA, sono fissi e.pertanto, le endonucleasi di restrizionepossono essere utilizzate per tagliare unamolecola di DNA in segmenti riproduci-bili. Quando i siti di restrizione sono statiidentificati, i segmenti si possono rag-gruppare in una serie ordinata, con ognisegmento in una posizione nota sullamappa lineare del genoma virale.
Un passo avanti nell'ibridazione
Si può vedere facilmente che il fatto dipossedere una simile serie di pezzi diDNA ordinati aumenta notevolmente ilpotere della tecnica di ibridazione mole-colare. Se le molecole marcate di RNAvengono mescolate con una serie ordinatadi segmenti di DNA, gli ibridi che ne risul-tano indicano con grande precisione laposizione nel genoma da cui l'RNA è sta-to trascritto. Con questa tecnica formida-bile abbiamo cercato di determinare se gliRNA messaggeri per le proteine tardivedell'adenovirus vengono trascritti nelnucleo cellulare come una singola unitàlunga e quindi tagliati in segmenti più pic-coli o se, invece, sono trascritti in unitàche hanno all'incirca le dimensioni dellemolecole del messaggero.
Quando la trascrizione dell'RNA viraleaveva preso inizio, le cellule infettate conl'adenovirus sono state esposte a nucleo-sidi radioattivi per un tempo così breveche solo alcuni precursori marcati si sonoaggiunti a ogni filamento di RNA. In unapopolazione di cellule infettate, vi sonoRNA-polimerasi pressoché in ogni posi-zione del DNA virale che funge da stam-po per la trascrizione dell' RN A. Se allecellule viene fornito il marcatore, alleestremità di un insieme di molecole diRNA di differenti lunghezze si troveran-no nucleotidi radioattivi. Gli RNA mar-cati più corti si troveranno vicini al puntodi inizio della trascrizione. Si osserveran-no anche molecole più lunghe e la piùlunga in assoluto si troverà vicino al puntoterminale della trascrizione.
L'RNA marcato è stato estratto dallecellule e purificato. I vari RNA purificatipoi sono stati separati in base alle dimen-sioni e ibridati con una serie ordinata disegmenti di DNA di adenovirus. L'ibri-dazione molecolare ci ha fornito un modoper saggiare le ipotesi su come viene pro-dotto l'RNA messaggero. Se gli m- RNAper le proteine tardive venissero trascrittiin piccole unità corrispondenti ai singolim- RNA, l'ibridazione darebbe un quadrocomplesso in cui RNA corti sarebberoibridati con il DNA vicino a ciascuno deimolteplici siti di inizio della trascrizione.
Si consideri ora che cosa accadrebbe segli RNA messaggeri per le proteine tardi-ve venissero trascritti come una singolaunità e quindi tagliati in segmenti. Vi sa-rebbe soltanto un punto di inizio per latrascrizione e gli RNA più corti si ibride-rebbero solo con il DNA vicino all'unicopunto di inizio. Gli RNA marcati più lun-ghi si ibriderebbero, nell'unità di trascri-
zione, in corrispondenza delle giuste po-sizioni intermedie. Quando l'RNA nu-cleare brevemente marcato e provenientedalle cellule infettate è stato ibridato conil DNA dell'adenovirus. gli RNA più cortisi sono ibridati solo in corrispondenza diun'unica posizione: l'unità di mappa 16.Gli RNA più lunghi si sono dispersi, inve-ce, in base alle loro dimensioni, alla destradell'unità 16. Pertanto abbiamo conclusoche gli RNA messaggeri per le proteinetardive vengono trascritti come unità sin-gola a partire da una posizione vicina aquella di mappa 16.
Posizioni sulla mappa genica
Due gruppi di giovani ricercatori. unoche operava presso il Cold Spring HarborLaboratory e l'altro al Massachusetts In-stitute of Technology, hanno compiutouna serie di esperimenti che hanno rivela-to in che modo avviene il processo di ma-turazione dell'RNA primario. cioè appe-na trascritto dall'unità di trascrizione perla proteina tardiva. Richard J. Roberts eRichard E. Gelinas del Cold Spring Har-bor Laboratory hanno trovato che tutti gliRNA messaggeri per le proteine tardivedell'adenovirus avevano gli stessi 11 nu-cleotidi confinanti con la testa in corri-spondenza dell'estremità 5'. Philip A.Sharp. Susan M. Berget e Claire Mooredel MIT, lavorando con I'm-RNA per unadelle proteine degli esoni, hanno trovatoche la sequenza comune adiacente allatesta non si ibridava con la porzione prin-cipale dell'm-RNA. Quando I'm- RNAper l'esone veniva mescolato con il DNA,formava in modo costante un ibrido, conlunghezza corrispondente a circa 4500basi, nel mezzo del genoma virale chepoteva essere reso visibile al microscopioelettronico. Dalla struttura centrale aduplice filamento si estendevano, tutta-via, due code. All'estremià 3' vi era lasequenza poli-A , dalla quale non ci si at-tendeva che si ibridasse perché non deri-vante dal DNA virale. All'altra estremitàdell'm-RNA, la sequenza comune 5' siestendeva anch'essa dall'ibrido comecoda a filamento singolo.
Ambedue i gruppi hanno riconosciutorapidamente la possibilità che la sequenza5' provenisse da un'altra parte del geno-ma dell'adenovirus. Sharp al MIT e, inol-tre, Thomas R. Broker e Louise T. Chowal Cold Spring Harbor Laboratory hannomescolato molti RNA messaggeri per leproteine tardive con la serie ordinata disegmenti del DNA e hanno ottenuto unrisultato sorprendente: ogni m-RNA siibridava, nel genoma virale, in quattroposizioni distinte. Ognuno formava unibrido in un unico sito corrispondente allasequenza codificante per la proteina.Inoltre, tutti gli m-RNA, indipendente-mente dalla posizione della sequenza co-dificante, hanno formato degli ibridi incorrispondenza delle posizioni 16, 20 e27. Il risultato ha suggerito che ognim-RNA per le proteine tardive dell'ade-novirus è un mosaico che consta di tresequenze comuni con un unico brano diinformazione genetica.
18SSPAZIATORE
SPAZIATORE
Il precursore dell'RNA ribosomale nelle cellule eucarioti include tre«spaziatori» che vengono rimossi nel processo di maturazione. Metodiavanzati per determinare le sequenze nucleotidiche del DNA e dell'RNAhanno mostrato come l'RNA primario, che nelle cellule umane ha unvalore di sedimentazione di 45S, viene trasformato negli r-RNA 185 e28S. La struttura generale di quasi tutti gli RNA ribosomali di cellule
RNA TRASCRITTO
5,8SSPAZIATORE
eucarioti, prodotti primari di trascrizione, è simile: quello illustrato quiproviene da cellule tumorali umane in coltura, dette cellule HeLa. Nelprimo stadio della maturazione intervengono gli enzimi che provocanouna scissione tra i segmenti 185 e 5,8S. Gli spaziatori sono poi rimossi daaltri enzimi; l'RNA 5,8S si attacca all'RNA 28S mediante legami aidrogeno, producendo le due forme stabili di RNA presenti nei ribosomi.
RNA-POLIMERASI
DNA
28 29

4
DNA VIRALE
PROTEINE INTERNE CAPSIDE DEL VIRUS
Un adenovirus, virus che provoca negli esseri umani infezioni delle vie respiratorie superiori, èillustrato in questa sezione trasversale schematica. Nelle prime indagini sulla maturazione del-I'm-RNA, esso è stato un importante oggetto di studio, perché a quell'epoca dalle cellule dimammifero non era ancora stato isolato alcun m-RNA. L'involucro esterno dell'adenovirus consta dimolecole proteiche, la maggior parte delle quali sono chiamate esoni. Le principali proteine degliesoni sono designate come 2, 3, 4 e IX. All'interno dell'involucro vi è una molecola di DNA a duplicefilamento, con una lunghezza di circa 36 000 nucleotidi. Quando il virus penetra in una cellulaumana, il suo DNA si duplica e da esso viene poi trascritto un numero relativamente piccolo dim-RNA. Il virus usurpa quindi l'apparato di sintesi proteica della cellula ospite per produrre pro-teine corrispondenti a questi m-RNA. Le proteine degli esoni fanno parte di un gruppo «tardi-vo» che viene sintetizzato principalmente dopo che il DNA virale ha cominciato a duplicarsi.
UNITÀ DI TRASCRIZIONE TARDIVA
L1 L2
L3 L4 L5
VT
VT
o
20 40
60
80
100
TRASCRIZIONE E AGGIUNTA DELLA TESTA
SCISSIONE IN CORRISPONDENZA DEL SITO POLI-A
X
AGGIUNTA DI POLI-A
T
T (V
\/,\)17
"
I A200
SEQUENZA CODIFICANTE PER LO SPL1CINGE SEQUENZE LEADER
)~IIM,200
m-RNA MATURO
VT
L'unità di trascrizione tardiva dell'adenovirus ha sia siti poli-A multipli sia siti multipli per losplicing. La si può trasformare così che dia degli m-RNA che codificano per più di una dozzina diproteine tardive dell'adenovirus. Una unità di trascrizione è un filamento di DNA che vici..continuamente trascritto. L'unità tardiva dell'adenovirus si estende dalla posizione 16 pratica-mente fino alla posizione 100 su una mappa lineare del DNA virale, in cui ogni unità corrisponde a360 nucleotidi. In tutti gli m-RNA tardivi vi sono sequenze complementari alle posizioni 16, 20 e27. Dalla posizione 30 fino a quasi la posizione 100 vi sono sequenze che codificano per le proteinetardive. raggruppate in famiglie designate con 1,1, L2, L3, L4 e L5. Ogni famiglia include leregioni codificanti per parecchie proteine affini. All'estremità destra di ogni famiglia vi è unsingolo sito poli-A, comune a tutti gli m-RNA presenti in quel gruppo. Subito dopo che ha inizio latrascrizione, all'estremità 5' si aggiunge una struttura, detta testa (T); per finire la RNA-polimera-si trascrive l'intera unità tardiva. Allorché attraversa i cinque siti poli-A, uno di essi vieneprescelto; altri enzimi tagliano via in questo punto i nucleotidi e viene aggiunto il poli-A. Qui èstata presa in esame I'L2: le sequenze tra le posizioni 16, 20 e 27 e tra la posizione 27 e la famigliaL2 vengono rimosse. Uno dei segmenti codificanti della famiglia L2 viene quindi prescelto; non ènota però la base della scelta per il sito poli-A e per il sito in cui avviene lo splicing. L'm-RNAmaturo include la testa aggiunta in corrispondenza dell'estremità 5', le sequenze comuni nelleposizioni 16, 20 e 27, il codice per una particolare proteina tardiva e la coda all'estremità 3'.
Ricerche recenti da parte di vari esper-ti, tra cui Heschel J. Raskas della Schoolof Medicine della Washington Universitye Joseph R. Nevins ed Edward Ziff dellaRockefeller University hanno illustrato inche modo gli m-RNA dell'adenovirus rie-scono a incorporare sia le sequenze co-muni sia quelle uniche. In questo lavorol'unità di trascrizione per le proteine tar-dive dell'adenovirus è apparsa come unapezzo estremamente complicato dellamacchina biologica. Si sa oggi che in unacellula di mammifero, infettata da unadenovirus, il principale prodotto di tra-scrizione ottenuto dopo la duplicazionedel DNA è l'unità di trascrizione per leproteine tardive, che porta sequenze co-dificanti per le 13 o 14 proteine tardive.L'unità di trascrizione si estende dallaposizione di mappa 16 pressoché alla po-sizione di mappa 100, all'estremità destra
della mappa stessa. Vicino all'unità 16, viè un filamento di nucleotidi chiamato ilpromotore tardivo, che induce la polime-rasi ad avviare la trascrizione. In corri-spondenza delle posizioni 16, 20 e 27 visono le sequenze comuni, osservate daSharp, Broker e Chow. Dalla posizione27 alla posizione 100 vi sono le sequenzecodificanti.
Le sequenze codificanti nell'unità ditrascrizione tardiva sono disposte in cin-que gruppi, designati come Li, L2, L3,L4 e L5 (nell'ordine da sinistra a destrasulla mappa). Ogni gruppo numerato in-clude le sequenze codificanti per una fa-miglia di proteine affini. All'estremitàdestra di ogni gruppo vi è un sito di attac-co poli-A. Per esempio il gruppo L2 siestende da circa la posizione 40 alla posi-zione 50; il suo sito poli-A è in corrispon-denza della posizione 50. Così vi sono, in
corrispondenza della posizione 16, un sitoche funge da testa e, nell'unità di trascri-zione tardiva, un totale di cinque poli-A.
Lo «splicing» del messaggero
Per ogni famiglia di proteine, le se-quenze codificanti hanno una disposizio-ne analoga, che si sovrappone. Tutte lesequenze codificanti si sovrappongonoall'estremità destra dell'unità numerata, etutte includono i nucleotidi adiacenti alsito poli-A . Nel mezzo del gruppo, invece,le sequenze codificanti divergono: cia-scuna include un differente segmento deinucleotidi che si trovano nel mezzo del-l'unità. Quando una molecola matura diRNA messaggero si forma dall'unità ditrascrizione tardiva viene inclusa una solasequenza codificante.
Da molti esperimenti è risultato chiaroche l'm-RNA maturo viene prodotto nelseguente modo. La trascrizione è avviatain corrispondenza del promotore tardivoe si muove verso destra a partire dallaposizione 16. Quando alcuni nucleotidisono stati trascritti, l'estremità 5' ondeg-giante dell'RNA viene completata. Lapolimerasi quindi si sposta verso la posi-zione 100, superando tutti e cinque i sitipoli-A . Durante il loro passaggio gli en-zimi tagliano il prodotto di trascrizione incorrispondenza di un sito poli-A e attac-cano qui la coda poli-A . La prima moleco-la di RNA negli stadi della maturazioneinclude, pertanto, tutte le sequenze dallaposizione 16 al sito poli-A prescelto. Peresempio, se viene prescelto il sito L2,l'RNA includerà le sequenze dalla posi-zione 16 alla posizione 50; la famiglia L idi sequenze codificanti viene inclusa conla famiglia L2, ma non con le famiglie L3,L4 e L5. L'RNA a valle del sito L2 (cheinclude i gruppi L3, L4 e L5) viene chia-ramente distrutto nel nucleo e non vienemai utilizzato come messaggero.
L'RNA nucleare, contenente il poli-Ae parzialmente maturato, include pertan-to parecchi «spaziatori», che non fannoparte dell'm-RNA maturo. Gli spaziatorivengono rimossi da enzimi in un processoche può richiedere anche 20-30 minuti.Vengono asportate anche le sequenze L 1e vengono rimossi i segmenti compresi trala posizione 16 e la posizione 20 e tra laposizione 20 e la posizione 27; rimangonocosì solo le sequenze comuni in corri-spondenza delle posizioni I 6, 20 e 27. Nerisulta una molecola con un gruppo disequenze codificanti affini. In questo casoè stata selezionata la sequenza L2. Lafamiglia della L2 ha tre sequenze codifi-canti. Una delle tre viene scelta da enzimiche rimuovono un segmento variabile diRNA dall'interno della famiglia di L2. Siottiene così m-RNA maturo, con la testacostituita dalle sequenze 5' comuni, lacoda poli-A e una sequenza codificantesolamente.
I nucleotidi dalla posizione 16 alla po-sizione 100 nel genoma dell'adenoviruscostituiscono un esempio estremo di unitàdi trascrizione complessa. Questa unitàpuò subire un processo di trasformazioneper dare alla fine più di un tipo di messag-
gero maturo. D'altra parte, un'unità ditrascrizione semplice fornisce solamenteun tipo di m-RNA. Grazie alla combina-zione di molteplici siti poli-A e di unosplicing (rottura e successiva saldatura)differenziale, si può trasformare, parten-do da una singola unità di trascrizionedell'adenovirus, una formidabile serie diproteine tardive.
Si può vedere facilmente che o i molte-plici siti poli-A o lo splicing differenzialeda soli potrebbero portare a una unità ditrascrizione complessa. Solo poche diqueste unità complesse sono state trovatenelle cellule eucarioti e di esse tutte tran-ne una hanno soltanto siti poli-A molte-plici. Soltanto una ha sia i siti poli-A mol-teplici sia lo splicing differenziale: essa è,nel moscerino della frutta (Drosophila),l'unità per la catena pesante della protei-na miosina.
Nel genoma del ratto, una singola unitàdi trascrizione con due siti poli-A è statatrovata da Ronald Evans, Michael Rosen-feld e collaboratori al Salk Institute forBiological Studies. Essa include la se-quenza codificante per due proteine:l'ormone calcitonina, sintetizzato nellatiroide, e un neuropeptide recentementescoperto, un ormone del sistema nervososintetizzato nell'ipofisi. Le due proteinehanno teste (l'estremità della catena pro-teica definita ammino-terminale o N--terminale) identiche, ma code (l'estremi-tà definita carbossiterminale o C-termi-nale) diverse. Nelle cellule della tiroideviene selezionato il primo sito poli-A e siottiene la calcitonina; nelle cellule dell'i-pofisi viene selezionato il secondo sitopoli-A e viene prodotto il neuropeptide.
Le ricerche effettuate dal 1975 a oggihanno dimostrato che i geni nelle celluleeucarioti possono venir regolati attraver-so il processo di maturazione dell'RNA.Non è ancora chiaro, tuttavia, quanto si-gnificativo risulterà essere tale processocome forma di regolazione genetica. Perla maggior parte dei geni degli eucarioti ilmetodo di regolazione principale risultaessere ancora il controllo della trascrizio-ne. Inoltre, la maggioranza delle unità ditrascrizione eucarioti che sono state esa-minate sono semplici unità, che possonocodificare solo per una proteina.
Esoni e introni
Anche in semplici unità di trascrizione,però, si svolge un processo di lavorazioneche serve a eliminare gli spaziatori. Dopoche la ricerca con l'adenovirus ha traccia-to il primo solco nel terreno, è risultatochiaro che, nei geni degli eucarioti, lesequenze espresse (esoni) sono anch'essecomunemente interrotte da sequenzenucleotidiche che non vengono tradotte(introni). Geni discontinui sono stati tro-vati in eucarioti così poco affini tra lorocome i lieviti e gli esseri umani. In tutte lecellule è necessario che si verifichi unosplicing perché si formi un RNA messag-gero senza gli introni e con gli esoni unitiper la traduzione. Così la maturazionedell'RNA è molto diffusa tra gli eucarioti.I geni discontinui degli eucarioti rappre-
sentano una notevole innovazione rispet-to ai geni dei procarioti. In una cellulaprocariote, i geni sono continui e segmen-ti supplementari di DNA vengono subitoscartati dal genoma. Le opinioni un tem-po prevalenti sull'evoluzione cellularesostenevano che gli eucarioti si erano evo-luti da organismi unicellulari molto similialle attuali cellule procarioti. Se ciò è giu-sto, ne consegue che gli introni vennerointrodotti nei geni dei procarioti nel corsodell'evoluzione. D'altra parte, Cari R.Woese e collaboratori dell'Università del-l'Illinois a Urbana-Champaign hannoavanzato l'ipotesi che gli eucarioti nonsiano derivati dai procarioti e che vi sianodue linee di discendenza procarioti
ugualmente antiche. Woese sostiene an-che che le tre linee di discendenza sianoderivate da un unico precursore, che eglichiama «progenote».
L'analisi particolareggiata degli introninei geni degli eucarioti conferma l'ideache gli eucarioti non si siano evoluti daiprocarioti. Innanzitutto le sequenze in-tercalate in alcuni geni degli eucarioti esi-stono al loro posto da moltissimo tempo.Per esempio, nell'emoglobina, una mole-cola che trasporta ossigeno, vi sono duecatene proteiche: la globina alfa e la glo-bina beta. Le sequenza amminoacidichedelle due catene sono diverse, ma le duecatene sono comunque ripiegate in mododa formare, nella molecola dell'emoglo-
30
31

PROCESSO DI MATURAZIONE ESEMPIO
ISITO DI INIZIO SITO DI FINE
--------------_____t_1) F DNAi TRASCRITTO
PROTEINA ISTONICA
[ 1m-RNA
I
I(A) F
I IINTERFERON ALFA,
MOLTI m-RNA DI LIEVITO\ SITO POLI-A
i
I
I
(S)1
I
(S),
I
(A) F
I i GLOBINE ALFAE BETA,
MOLTI m-RNA DICELLULE EUCARIOTI
//N
N\
SITO PER LO SPLIC1NG
I[/
I I
Una semplice unità di trascrizione può essere trasformata in modo da dare un solo tipo dim-RNA. A sinistra, viene rappresentato in forma schematica il processo di maturazione di unitàsemplici, mentre a destra sono riportati esempi di tali unità e organismi nei quali si trovano. Ilsito di inizio è dove ha inizio la trascrizione; il sito di fine è dove termina la trascrizione. Il sitopoli-A corrisponde al punto in cui la coda 3' di poli-A viene inserita. In alcuni casi, la RNA-poli-merasi si arresta in corrispondenza del sito poli-A (1). In altri casi, invece, la trascrizione procedeal di là di esso (2). Il prodotto di trascrizione può venir tagliato e saldato così da dare un m-RNAfinito di minore lunghezza, se nel suo interno si trovano delle sequenze non codificanti (3). Lagrande maggioranza delle unità di trascrizione trovate nelle cellule dei mammiferi sono semplici.
PROCESSO DI MATURAZIONE ESEMPIO
/SITO DI INIZIO
(A),I
(A)2
I
SITO DI FINE
CATENE PESANTIDELL'IMMUNOGLOBULINA,
CALCITONINA
,
i
2
I
I(S)1 (S)2
I I
(S),
i
(A)
i
F
iGENI IN PREVALENZA
VIRALI. SV 40, POLIOMA,RETROVIRUS
..----...., ,
r- Il I
Un'unità di trascrizione complessa può venir trasformata così da dare più di un m-RNA e pertantopiù di una sola proteina. La presenza di due o più siti poli-A può sfociare in una unità complessa(1). Se questa viene tagliata in corrispondenza del primo sito poli-A, si produce un solo m-RNA;se viene tagliata, invece, in corrispondenza di un altro sito, si produce un differente m-RNA. Inuna unità di trascrizione con un solo sito poli-A, vi possono essere più di un unico sito per losplicing. L'm-RNA che viene prodotto dipende da quali siti per lo splicing siano prescelti (2).Unità complesse con più di un sito poli-A sono comuni nei virus e sono state trovate anche nellecellule di mammifero. Unità con splicing differenziale sono state notate in virus e un esempioanche in una cellula eucariote. Unità di trascrizione complesse, con molteplici siti per lo splicing,possono venir integrate nei geni della cellula ospite ed espresse assieme ai geni di quest'ultima.
bina, analoghe strutture tridimensionali.Tali strutture sono anche simili alla strut-tura della mioglobina, la proteina che silega all'ossigeno e che è presente nellecellule muscolari.
Malgrado queste somiglianze struttura-li, le differenze nelle sequenze amminoa-cidiche della globina alfa, della globinabeta e della mioglobina fanno pensare chei geni per le tre proteine abbiano comin-
ciato a divergere da un precursore comu-ne forse un miliardo di anni fa. Nel con-tempo, tutti e tre i geni hanno tre esoniseparati da due introni che si trovano inposizioni analoghe. Laddove le sequenzedegli amminoacidi hanno cominciato adivergere, sembra che la disposizionedegli introni si sia conservata, cosicchéessi hanno potuto rimanere al loro postoper un miliardo di anni.
Inoltre, quando si esamina la strutturatridimensionale di una proteina, si trovaspesso che la catena proteica può esseresuddivisa in segmenti con differenti fun-zioni; tali segmenti prendono- il nome dicampi funzionali. Molti introni dividono igeni in vari pezzi, ciascuno dei quali codi-fica per un differente campo funzionale.Pertanto, se gli introni venissero introdot-ti a caso in un genoma preesistente,avrebbero il notevole effetto di dividere igeni in pezzi corrispondenti a funzioniseparate. Sembra più probabile che essi sitrovassero già al loro posto forse fin dal-l'inizio dell'evoluzione e che gli esoni co-dificanti per i campi funzionali si sianoevoluti in maniera indipendente l'uno dal-l'altro.
All'inizio era l'RNA
Sulla base di queste argomentazioni edi altre ancora, W. Ford Doolittle dellaDalhousie University nella Nuova Scozia,Darryl C. Reanney della Latrobe Univer-sity in Australia e io stesso abbiamo pro-posto che, anche in tempi precellulari,l'informazione per le proteine non sia sta-ta continua. Si ritiene da più parti che,anche se il DNA viene prima dell'RNAnella sequenza che porta alla sintesi delleproteine nelle cellule attuali, il primo aci-do nucleico codificante non fu DNA, maRNA. Tra le varie ragioni si annovera ilfatto che le primitive catene di RNA pos-sono essere sintetizzate senza enzimi.Inoltre, l'RNA può immagazzinare in-formazioni codificanti ed è necessario perla traduzione. Se l'RNA si è evoluto comecatena nucleotidica casuale, la sua infor-mazione utile potrebbe non essere statacontinua. Lo splicing dell'RNA per riuni-re pezzi d'informazione discontinui sa-.rebbe stato così un evento evolutivo pre-coce e propizio. Di recente, Thomas R.Cech dell'Università del Colorado aBoulder ha dimostrato che l'RNA puòessere tagliato e saldato di nuovo senza ilconcorso di enzimi, perlomeno nel pro-tozoo Tetrahymena. Pertanto non è daescludere che le sequenze intercalari esi-stessero fin dai tempi più antichi e che losplicing dell'RNA sia stato una caratteri-stica precoce dell'espressione genica.
Queste congetture sull'evoluzione nontroveranno subito una soluzione. Nel 'frat-tempo rimane da rispondere ad alcunipressanti e precisi interrogativi. Si stacompiendo uno sforzo molto intenso peridentificare gli enzimi che tagliano e risal-dano il prodotto di trascrizione. Quandosi troveranno, sarà possibile scoprirecome un tipo di splicing venga prescelto alposto di un altro. Analogamente si dovràanche conoscere perché un sito poli-Avenga selezionato al posto di un altro. Aun livello più generale si pone la questio-ne di sapere quanto sia significativo ilprocesso di maturazione dell'RNA nellaregolazione genetica in complesso. Diqualunque natura saranno le risposte, leindagini sulla maturazione dell'RNA con-tinueranno ad avere un effetto profondosul nostro modo di capire in che modo igeni si esprimano nelle cellule eucarioti.
32