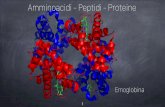
AMMINOACIDI 3 - Server · 2015-05-18 · AMMINOACIDI Gli amminoacidi comuni o standard che tutti...
Transcript of AMMINOACIDI 3 - Server · 2015-05-18 · AMMINOACIDI Gli amminoacidi comuni o standard che tutti...

AMMINOACIDIGli amminoacidi comuni o standard che tutti gli organismi viventi utilizzano perl’assemblaggio delle PROTEINE sono 20 e sono codificati dal codice genetico.
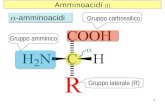
Sono tutti α-amminoacidi possiedono un gruppo amminico e uncarbossilico entrambi legati allo stesso atomo di carbonio (il carbonio α)
α-amminoacido generico scritto nella sua forma completamente protonata
COOH
H3N C H
R
+ α
COOH
H3N C H
R
+
COOH
H3N C H
R
++ α
Gli amminoacidi differiscono tra loro per il tipo di catena laterale R legata all’atomo di Cα. Dalla natura della catena laterale R dipendono: carica,
idrofobicità, dimensioni, reattività, capacità di formare legami idrogeno.

STRUTTURA DEI 20 AMMINOACIDI STANDARD
1) Amminoacidi con catena laterale R alifatica
Gly (G) Ala (A)GLICINA ALANINA
PROLINA
Pro (P)
Val (V)Leu (L) Iso (I)
LEUCINA ISOLEUCINA VALINA
COOH+ l
H3N―C ―Hl
H
COOH+ l
H3N―C ―Hl
CH3
COO-
lC ―H
+H2N CH2
l lH2C CH2
COOHlC ―H
+H2N CH2
l lH2C CH2
COO-
+ lH3N―C ―H
lCH
CH3 CH3
COOH+ l
H3N―C ―Hl
CH
CH3 CH3
COO-
+ lH3N―C ―H
lCH2lCH
CH3 CH3
COOH+ l
H3N―C ―Hl
CH2lCH
CH3 CH3
COOH+ l
H3N―C ―Hl
H―C―CH3lCH2l
CH3
1) Amminoacidi con catena laterale R alifatica
Gly (G) Ala (A)GLICINA ALANINA
PROLINA
Pro (P)
Val (V)Leu (L) Iso (I)
LEUCINA ISOLEUCINA VALINA
COOH+ l
H3N―C ―Hl
H
COOH+ l
H3N―C ―Hl
H
COOH+ l
H3N―C ―Hl
CH3
COOH+ l
H3N―C ―Hl
CH3
COO-
lC ―H
+H2N CH2
l lH2C CH2
COOHlC ―H
+H2N CH2
l lH2C CH2
COO-
lC ―H
+H2N CH2
l lH2C CH2
COOHlC ―H
+H2N CH2
l lH2C CH2
COO-
+ lH3N―C ―H
lCH
CH3 CH3
COOH+ l
H3N―C ―Hl
CH
CH3 CH3
COO-
+ lH3N―C ―H
lCH
CH3 CH3
COOH+ l
H3N―C ―Hl
CH
CH3 CH3
COO-
+ lH3N―C ―H
lCH2lCH
CH3 CH3
COOH+ l
H3N―C ―Hl
CH2lCH
CH3 CH3
COO-
+ lH3N―C ―H
lCH2lCH
CH3 CH3
COOH+ l
H3N―C ―Hl
CH2lCH
CH3 CH3
COOH+ l
H3N―C ―Hl
H―C―CH3lCH2l
CH3
COOH+ l
H3N―C ―Hl
H―C―CH3lCH2l
CH3
AMMINOACIDI CON CATENA LATERALE IDROFOBICA

2) AMMINIACIDI CON CATENA LATERALE POLARE NON CARICA
CISTEINA Cys (C)
COO-
+ lH3N―C ―H
lCH2lSH
H
SERINA Ser (S)
COO-
+ lH3N―C ―H
lCH2lOH
H COO-
+ lH3N―C ―H
lH―C―OH
lCH3
H
TREONINA Thr (T)
GLUTAMMINA Gln (Q)
COOH+ l
H3N― C ―HlCH2lCH2lC
H2N O
COOH+ l
H3N― C ―HlCH2lC
H2N O
ASPARAGINA Asn (N)
METIONINA Met (M)
COO-
+ lH3N―C ―H
lCH2l
CH2lS l
CH3
H

Contengono elettroni π delocalizzati
Hanno un massimo di assorbimento a 280 nm nell’UV, che è utile per rilevare la presenza di proteine in soluzione e per determinare la loro concentrazione.
TIROSINA, FENILALANINA E TRIPTOFANO SONO AMMINOACIDI AROMATICI
TIROSINA Tyr (Y)
COO-
+ lH3N―C ―H
lCH2l
lOH
COO-
+ lH3N―C ―H
lCH2l
lOH
H
FENILALANINA Phe (F)
COO-
+ lH3N―C ―H
lCH2l
COO-
+ lH3N―C ―H
lCH2l
H COO-
+ lH3N―C ―H
lCH2l
N H
COO-
+ lH3N―C ―H
lCH2l
N H
H
TRIPTOFANO Trp (W)

3) AMMINOACIDI CON CATENA LATERALE IONIZZABILE POSITIVAMENTE
LISINA Lys (L)ARGININA Arg (R)
ISTIDINA His (H)
COO-
+ lH3N― C ―H
lCH2l
CH2l
CH2l
CH2l
NH3+
HCOO-
+ lH3N― C ―H
lCH2l
CH2l
CH2l
CH2l
NH3+
H COO-
+ lH3N― C ―H
lCH2l H
C―N +CH
C―NH H
COO-
+ lH3N― C ―H
lCH2l H
C―N +CH
C―NH H
HCOO-
+ lH3N― C ―H
lCH2lCH2lCH2lNHlC
NH2 NH2+
COO-
+ lH3N― C ―H
lCH2lCH2lCH2lNHlC
NH2 NH2+
H
Asp (D)
AC. ASPARTICO
Glu (E)
AC. GLUTAMMICO
COO -+ l
H3N― C ―H
lCH
2lCOO -
HCOOH+ l
H3N ―C ―H
lCH 2
lCOOH (COO-)
COO -+ l
H3N― C ―H
lCH
2lCH
2lCOO -
HCOOH+ l
H3N ― C ―H
lCH2l
CH2lCOOH (COO-)
4) AMMINOACIDI CON CATENA LATERALE IONIZZABILE NEGATIVAMENTE

Il 21° amminoacido: selenocisteina (SEC).Incorporato in alcune proteine di varie specie
e codificato da un codone di stop in particolaricondizioni

Stereoisomeria degli amminoacidi
In tutti gli AA standard (ad eccezione della Glicina) il C-α è CHIRALE o ASIMMETRICO
Glicina (Gly) unico AA non chirale
Gli AA chirali possono esistere come stereoisomeri, per ognuno di essi esistono 2ENANTIOMERI: l’isomero D e quello L
TUTTI GLI AMMINOACIDI CHE FORMANO LE PROTEINE SI TROVANO NELLACONFIGURAZIONE L (TUTTAVIA gli isomeri D degli amminoacidi sonopresenti in alcuni antibiotici e nella parete cellulare di alcunimicrorganismi ma non sono utilizzati per sintetizzare le proteine deimammiferi)
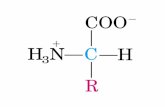
COOH
H3N C H
R
+ α
COOH
H3N C H
R
+
COOH
H3N C H
R
++ α
COOH
H3N C H
R
+
COOH
H3N C H
R
+ α
COOH
H3N C H
R
++
COOH
H3N C H
R
++ α

AMMINOACIDI NON COMUNITutte le specie viventi contengono, oltre agli amminoacidi standard, diversi altri amminoacidi in configurazione L che sono intermedi nelle vie metaboliche o precursori degli AA standard.
ORNITINA (omologo inferiore della Lisina)
COO-
+ lH3N―Cα―H
lCβH2
lCγH2
lCδH2
lNH3+
COO-
+ lH3N―C ―H
lCH2
lCH2
lCH2
lNHlC
NH2 O
COO-
+ lH3N―C ―H
lCH2
lCH2
lCH2
lNHlC
NH2 O
COO-
+ lH3N―C ―H
lCH2
lCH2
lCH2
lNHlC
NH2 O
CITRULLINA

DERIVATI AMMINOACIDICIMolti amminoacidi standard sono modificati chimicamente da enzimi specifici per produrre sostanze di grande importanza biologica.
Ac. γ-amminobutirrico (GABA)Derivato del glutammato
3
+
IstaminaDerivato dell’istidina
Adrenalina Derivato della tirosina
H3N+ CH2
CH2
CH2
COOH
H3N+ CH2
CH2
CH2
COOHlOH
OH OH
CH CH2 NH CH3
OH
CH CH2 NH CH3
OH
CH CH2 NH CH3

DERIVATI AMMINOACIDICIMolti amminoacidi standard sonomodificati chimicamente da enzimi specificidopo essere stati incorporati nellacatena polipeptidica.Influenzano la funzionalità della proteina in cui sonoinseriti.
FOSFOSERINA
+ HH3N― C―COOH
lCH2l
Ol
HO – P =Ol
OH
COO-
lC ―H
+H2N CH2
l lH2C C-H
lOH
1
2
3
5 4
4-IDROSSI-PROLINA
COO-
lC ―H
+H2N CH2
l lH2C C-H
lOH
COO-
lC ―H
+H2N CH2
l lH2C C-H
lOH
1
2
3
5 4
4-IDROSSI-PROLINA
COO-
+ lH3N―C ―H
lCH2
lCH2
lH-C-OH
lCH2
lNH3+
5-IDROSSILISINA
1
2
3
4
5
6
COO-
+ lH3N―C ―H
lCH2
lCH2
lH-C-OH
lCH2
lNH3+
5-IDROSSILISINA
1
2
3
4
5
6

ossidazione
riduzione

GLI AMMINOACIDI SONO ANFOLITI: IN SOLUZIONE SI COMPORTANO SIA DA BASI SIA DA ACIDI DEBOLI
HANNO GRUPPI IONIZZABILI CAPACI DI SCAMBIARE PROTONI CON LA SOLUZIONE ACQUOSA:
α-AMMINICO
α-CARBOSSILICO
Come faccio a stabilire come sarà ionizzato un amminoacido ad un dato valoredi pH?
In che modo ionizzano questi 2 gruppi?
O¯C = O
HH – N –
H
+
OHC O
HH N C H
HCH3
+ α
OHC O
HH N C H
HCH3
+ α
OHC O
HH N C H
HCH3
+
OHC O
HH N C H
HCH3
++ α

Acido debole Base coniugata
Esiste una relazione che lega pH, pKa e le concentrazioni di un acido debole e della sua base coniugata in soluzione.Equazione di Henderson-Hasselbalch
Ka = costante di dissociazione dell’acido. Più è grande maggiore è la dissociazione dell’acido.
La capacità di un acido di dissociare e scambiare il protone con la soluzione dipendenon solo dalla sua Ka ma anche dal pH della soluzione.
Può essere espressa in formalogaritmica: pKa = -log KaQuanto più è piccolo il pKa tanto piùl’acido è forte
HA H+ + A-HA H+ + A-Ka
Ka =[H+] [A-]
[HA]Ka =
[H+] [A-]
[HA]
pH = pKa + log [A-][HA]
pH = pKa + log [A-][HA]
DISSOCIAZIONE DI UN ACIDO DEBOLE

Forma completamenteProtonata
-COOH-NH3
+
Carica netta: +1
Ionizzazione di un Amminoacido diprotico (CON 2 GRUPPI CAPACI DI DISSOCIARE ILPROTONE):-2 gruppi protonabili, α-amminico e α-carbossilico-2 costanti di dissociazione acida, quindi 2 pKa-in soluzione saranno presenti 3 forme diversamente ionizzate in equilibrio tra loro.
OHC O
HH N C H
HCH3
+ α
OHC O
HH N C H
HCH3
+ α
OHC O
HH N C H
HCH3
+
OHC O
HH N C H
HCH3
++ α
pKa1
H+
H+
H+
H+
pKa2
Forma ZWITTERIONICA
-COO-
-NH3+
Carica netta: 0
Forma completamenteDeprotonata
- COO-
-NH2
Carica netta: -1
O-
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+
C OH
H N C HH
CH3
++
O-
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+
C OH
H N C HH
CH3
++
O-
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+
C OH
H N C HH
CH3
++
O-
C O
H N C HH
CH3
α
C O
H N C HH
CH3
α
C O
H N C HH
CH3
C O
H N C HH
CH3
O-
C O
H N C HH
CH3
α
C O
H N C HH
CH3
α
C O
H N C HH
CH3
C O
H N C HH
CH3
Da cosa dipende la prevalenza in soluzione di una forma sulle altre?

AA+1 AA0 AA-1
ZWITTERIONEEntrambi i gruppiprotonati
[AA+1]= [AA0] [AA0]= [AA-1]
pH = pKa1 pH = pKa2Tymoczko et al., PRINCIPI DI BIOCHIMICA zanichelli editore S.p.A Copyright © 2010
100
fraz
ion
e m
ola
re (
%)
50 -
Risposta: dal valore dei pKa e dal pH della soluzione

Devo considerare la dissociazione del gruppo α-COOH e quindi il pKa1 = -log Ka1 (costante di dissociazione acida del gruppo α-COOH).
A quale valore di pH nella soluzione di alanina la sua forma cationica AA+1 e quella zwitterionica AA0 saranno presenti in uguale concentrazione?
pH = pKa1 + log [α-COO-][α-COOH]
pH = pKa1pH = pKa1 + log [α-COO-][α-COOH]
pH = pKa1 + log [α-COO-][α-COOH]
pH = pKa1
pKa1
H +
H +
H +H +
H +H +
O-
C O
C HH
CH3
+ α
C O
C HH
+ α
C O
C HH
+
C O
C H++
O
C O
C H+ α
C O
C H+ α
C OHN C H+
C O
C H++HHH
O
C O
C HH
CH3
+ α
C O
C HH
+ α
C O
C HH
+
C O
C H++
OH
C O
C H+ α
C O
C H+ α
C OHN C H+
C O
C H++HHH
[AA+1]=[AA0] quando [α-COOH]=[α-COO-]Allora il valore del pH è uguale al valore del pKa1

Devo considerare la dissociazione del gruppo α-NH3+, e quindi il
pKa2 = -log Ka2 (costante di dissociazione acida del gruppo α-NH3+)
Il valore del pKa2 è uguale a quel valore di pH in corrispondenza del quale: [α-NH3
+]=[α-NH2] e quindi [AA0]=[AA-1]
Che valore di pH deve avere la soluzione di alanina affinché la sua forma zwitterionica AA0 e quella anionica AA-1 siano presenti in uguale concentrazione?
AA0 AA-1
OHC O
HH N C H
HCH3
+ α
OHC O
HH N C H
HCH3
+ α
OHC O
HH N C H
HCH3
+
OHC O
HH N C H
HCH3
++ α
pKa1 O-
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+
C OH
H N C HH
CH3
++
H+
H+
O-
C O
H N C HH
CH3
α
C O
H N C HH
CH3
α
C O
H N C HH
CH3
C O
H N C HH
CH3
H+
H+
pKa2OHC O
HH N C H
HCH3
+ α
OHC O
HH N C H
HCH3
+ α
OHC O
HH N C H
HCH3
+
OHC O
HH N C H
HCH3
++ α
pKa1 O-
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+
C OH
H N C HH
CH3
++
O-
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+ α
C OH
H N C HH
CH3
+
C OH
H N C HH
CH3
++
H+
H+
H+H+
H+H+
O-
C O
H N C HH
CH3
α
C O
H N C HH
CH3
α
C O
H N C HH
CH3
C O
H N C HH
CH3
O-
C O
H N C HH
CH3
α
C O
H N C HH
CH3
α
C O
H N C HH
CH3
C O
H N C HH
CH3
H+
H+
H+H+
H+H+
pKa2
pH = pKa2 + log [α-NH2][α-NH3
+]pH = pKa2pH = pKa2 + log
[α-NH2][α-NH3
+]pH = pKa2 + log
[α-NH2][α-NH3
+]pH = pKa2

Acido aceticopKa COOH è circa 4,8
MetilamminapKa NH3
+ è circa 10,6
α-Amminoacido (Gly)Per effetto di repulsione tra il
gruppo NH3+ e il protone del COOH
>>> si abbassa il pKa del COOH ad un valore di 2.3
È stabilizzato lo zwitterione
α-Amminoacido (Gly)Gli ossigeni elettronegativi del
gruppo COO- attraggono elettroni dal gruppo amminico abbassando il
suo pKa ad un valore di 9,6

PUNTO ISOELETTRICOpI = corrisponde a quel valore di pH al quale l’amminoacido in soluzione ha carica netta = 0
Forma ionica prevalente: zwitterione ~ 100%
Forma anionica e forma cationica sonopresenti in soluzione in uguale concentrazione
rapporto 1:1 fra Ala-1 e Ala+1
Per un amminoacido con due gruppi dissociabili si calcola:
Per es.: Gly >>> pI = (2.34+9.60)/2 = 5.97
(pKa1(α-COOH) + pKa2(α-NH3+))
pI =pI =2
Ala0

AA+ AA0 AA-
Entrambi i gruppiprotonati
pH = pI
I valori dei punti isoelettrici degli amminoacidi con due gruppi ionizzabili sono molto simili e compresi fra 5,0 e 6.5. In una soluzione a pH neutro la forma ionica prevalente degli amminoacidi diprotici è quella zwitterionica con carica netta zero.
Qual è la forma ionica prevalentemente presente a valori intorno alla neutralità (7.0)?
7
AA0

pH 1 2.34 5.97 9.6 14
Determinazione sperimentale dei pKa e del pI di un amminoacido diprotico: curva di titolazione della glicina
pKa1 pKa2
AA-1
anione
COO-
|H2N- C – H
|H
COO-
+ |H3N- C – H
|H
COOH+ |
H3N- C – H|
H
Vale la regola che a valori di pH superiori al punto isoelettrico l’aminoacido diprotico è prevalentemente nella forma ionica negativa (deprotonata)
Al di sotto del p. isoelettrico è prevalentemente nella sua forma ionica positiva (protonata)
Il punto isoelettrico corrisponde ad un punto di equivalenza della curva di titolazione mentre i pKa corrispondono a punti di semiequivalenza
AA+1
CationeAA0
Zwitterione
pI

13
pH
7
00 0,5 1,0 1,5 2,0
Moli OH-/mole di amminoacido
13
pH
7
00 0,5 1,0 1,5 2,0
Moli OH-/mole di amminoacido
13
pH
7
00 0,5 1,0 1,5 2,0
Moli OH-/mole di amminoacido
A pH inferiori al pka1 prevale la forma AA+1 A valori di pH compresi fra il
pKa1 e il pKa2 prevale la forma ionica AA0 e raggiunge la massima concentrazione a pH uguale al p. Isoelettrico
A pH uguale al pka1
abbiamo un’uguale concentrazione delle forme ioniche AA+1 e AA0
COOH+ Ι
H3N–C–HΙ
H
COOH+ Ι
H3N–C–H Ι
H
=
COO–
+ ΙH3N–C–H
ΙH
COO–
+ ΙH3N–C–H
ΙH
=
COO–
ΙH2N–C–H
ΙH
COO–
+ ΙH3N–C–H
ΙH
pH = pka2 → abbiamo un’uguale concentrazione delle forme ioniche AA0 e AA-1
A pH superiori al pka2
prevale la forma AA-1
COO–
ΙH2N–C–H
ΙH

GLI AMMINOACIDI FUNZIONANO DA TAMPONIESISTONO ALMENO DUE REGIONI NELLA SCALA DEL pH IN CUI
L’AMMINOACIDO è UN BUON TAMPONE
>>>>> pH = pKa ± 1
Es.: Glicina>> pKa1 = 2.34 (50% di AA+ e AA0)
Tra pH 1.34 e pH 3.34 la glicina funziona da tampone(a pH 1.34 avremo il 90% di AA+ e il 10% di AA0
a pH 3.34 avremo il 10% di AA+ e il 90% AA0)
>> pKa2 = 9.60 (50% di AA- e AA0)Tra pH 8.60 e 10.60 la glicina funziona da tampone
(a pH 8.60 avremo il 90% di AA0 e il 10% di AA-
a pH 10.60 avremo il 10% di AA0 e il 90% di AA-)


AMMINOACIDO TRIPROTICO (3 gruppi ionizzabili): in soluzione saranno presenti4 forme ioniche dell’AA in equilibrio tra loro.Sono 3 i pKa da considerare.Es.: Acido glutammico (Glu, E)
CATIONE
- αCOOH
- αNH3+
(R) - COOH
ZWITTERIONE
- αCOO-
- αNH3+
(R) - COOH
ANIONE -1
- αCOO-
- αNH3+
(R) - COO-
ANIONE -2
-αCOO-
- αNH2
-(R) - COO-
AA+1
pKa1
COOH+ |
H3N – C – H|CH2
|CH2
|COOH
COO-
+ |H3N – C – H
|CH2
| CH2
|COOH
COO-
+ |H3N – C – H
|CH2
|CH2
|COO-
COO-
|H2N – C – H
|CH2
|CH2
|COO-
AA0 AA-1 AA-2
pKaR pKa2

Quante moli di OH- devo aggiungere per avere l’ac. glutammico al suo p. isoelettrico?
Come calcolo il punto isoelettrico di un amminoacido acido?
0 0.5 1.0 1.5 2.0 2.5 3.0
10
8
6
4
2
pH
Moli di OH-/moli amminoacido
1
pI = (2.19 + 4.25)/2 =3.22AA+1
AA+1= AA0
AA0
AA0=AA-1
AA-1
AA-1= AA-2
pKa2 =9.67
pKaR = 4.25
pKa1 =2.19
3.22
7
AA-2
pKa1 + pKaR
2 pI =
pKa1 + pKaR
2 pI =
A quali valori di pH avrò la massima concentrazione di AA+1, AA0, AA-1, AA-2?
AA+1
pKa1
COOH+ |
H3N – C – H|CH2
|CH2
|COOH
COO-
+ |H3N – C – H
|CH2
| CH2
|COOH
COO-
+ |H3N – C – H
|CH2
|CH2
|COO-
COO-
|H2N – C – H
|CH2
|CH2
|COO-
AA0 AA-1 AA-2
pKaR pKa2

QUALI SONO E QUANTE SONO LE REGIONI TAMPONANTI? Individuare gli intervalli di pH
0 0.5 1.0 1.5 2.0 2.5 3.0
10
8
6
4
2
pH
Moli di OH-/moli amminoacido
Le regioni tamponanti sono 3:pKa1 ± 1;pKaR ± 1;pKa2 ± 1
pKa2 = 9.67
pKaR = 4.25
pKa1 = 2.19
7
AA+1
pKa1
COOH+ |
H3N – C – H|CH2
|CH2
|COOH
COO-
+ |H3N – C – H
|CH2
| CH2
|COOH
COO-
+ |H3N – C – H
|CH2
|CH2
|COO-
COO-
|H2N – C – H
|CH2
|CH2
|COO-
AA0 AA-1 AA-2
pKaR pKa2

IstidinapKR = 6,0
Deprotonazione del gruppo imidazolicoÈ un ibrido di risonanza
La carica + è delocalizzatafra i due atomi di azoto
COO-
+ lH3N― C ―H
lCH2l H
C―N +CH
C―NH H
COO-
+ lH3N― C ―H
lCH2l H
C―N +CH
C―NH H
H
COOH+ l
H3N―C ―Hl
CH2l H
C―NCH
C―NH H
+
COOH+ l
H3N―C ―Hl
CH2l H
C―NCH
C―NH H
+
COOH+ l
H3N―C ―Hl
CH2l H
C―NCH
C―NH H+
COOH+ l
H3N―C ―Hl
CH2l H
C―NCH
C―NH H+

pKR = 6,0
A pH 7,0 (pH 7.0 = pKR + 1) in soluzione avremo ~ il 10 % della forma AA+1 (imidazolo protonato) e ~ il 90% di quella AA0 (imidazolo deprotonato), a pH vicini a quello fisiologico sono entrambi presenti in concentrazioni significative e l’istidina può fungere da tampone.
È un ottimo tampone nell’ambiente cellulareLa catena R di un residuo di His che fa parte di una proteina può esseresia protonata che deprotonata >>> il suo pKR può variare, dipende dallecondizioni che si creano nel microambiente in cui si trova
COO-
+ lH3N―C ―H
lCH2l H
C―NCH
C―NH
COO-
+ lH3N―C ―H
lCH2l H
C―NCH
C―NH
COO-
+ lH3N―C ―H
lCH2l H
C―NCH
C―NH H
+
COO-
+ lH3N―C ―H
lCH2l H
C―NCH
C―NH H
+
AA+1 AA0

AA+2 AA+1 AA0 AA-1
pKa1 pKaR pKa2
0 0.5 1.0 1.5 2.0 2.5 3.0
10
8
6
4
2
pH
Moli di OH-/moli amminoacido
pKa1 = 1,82
pKaR = 6,0
pKa2 = 9.17
AA+2
AA+2=AA+1
AA0
AA0=AA+1
AA+1
AA0=AA-1
pI = 7,59
pI = (6,0 + 9,17)/ 2pI = 7,59
AA-1
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COO-
+ |H3N- C – H
|CH2
|C ―N| CH
HC ―N+
H
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COO-
+ |H3N- C – H
|CH2
|C ―N| CH
HC ―N+
H
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COO-
+ |H3N- C – H
|CH2
|C ―N| CH
HC ―N+
H
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COO-
+ |H3N- C – H
|CH2
|C ―N| CH
HC ―N
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COO-
+ |H3N- C – H
|CH2
|C ―N| CH
HC ―N
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COO-
+ |H3N- C – H
|CH2
|C ―N| CH
HC ―N
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COO-
|H2N- C – H
|CH2
|C ―N| CH
HC ―N
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COO-
|H2N- C – H
|CH2
|C ―N| CH
HC ―N
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COO-
|H2N- C – H
|CH2
|C ―N| CH
HC ―N
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
COOH+ |
H3N- C – H|CH2
|C ―N| CH
HC ―N+
H
H
pKaR + pKa2pI =
2
pKaR + pKa2pI =
2















![MOLECOLE DI INTERESSE BIOLOGICO: CHIMICA DELLA ......-3) Le macromolecole biologiche sono: [ ] A – amminoacidi, vitamine e proteine [ ] B – glicerolo, trigliceridi e acidi nucleici](https://static.fdocumenti.com/doc/165x107/5ff7fdb5ae4cd27c6133ae2f/molecole-di-interesse-biologico-chimica-della-3-le-macromolecole-biologiche.jpg)