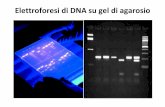
Gel di agarosio
description
Transcript of Gel di agarosio

Gel di agarosio
DNA è una molecola carica negativamente e quindi in un campo elettrico migra verso il polo positivo. La velocità di migrazione dipende da: 1. La sua forma2. Il suo rapporto carica/massa
Le molecole di DNA hanno la stessa forma e molto simili rapporti carica massa quindi si usa le gel elettroforesi che separa i frammenti di DNA in base alla loro dimensione


Elettroforesi di DNAPermette di separare, identificare e purificareframmenti di DNA. Contengono pori attraverso i quali il DNA migra al polo positivo. Più piccole sono le molecole, più veloce migrano sul gelPoliacrilammide DNA 5-500 bpAlto potere risolutivo (1bp) – Gel verticali – E costanteAgarosio DNA 200-50000 bpVarie concentrazioni. Minore potere risolutivoGel orizzontali – E costante
L’agarosio, in concentrazione opportuna, viene fuso insieme alla soluzione tampone.La soluzione fusa viene versata nella vaschetta e lasciata solidificare.Quando un campo elettrico viene applicato al gel il DNA, carico negativamente, si muove verso l’anodo.

Fattori che influenzano la velocità di migrazione del DNA sul gel
Dimensione della molecola di DNALa velocità di migrazione è inversamente proporzionale al lg10bp
Concentrazione dell’agarosioEsiste una relazione lineare tra la concentrazione del gel () e logaritmo della mobilità elettroforetica () log = log °-Kr ° = mobilità elettroforetica liberaKr = coefficiente di ritardo (dipende dalle proprietà del gel e dalle dimensioni e forma delle molecole)
Usando gel di conc. differenti è possibile separare una vasta gammadi molecole di DNAConc. di agarosio Range di separazione (%) (Kb) 0.3 5-60 0.9 0.5-7 1.2 0.4-6 1.5 0.2-3 2.0 0.1-2


Fattori che influenzano la velocità di migrazione del DNA sul gel
Conformazione del DNAIl DNA plasmidico puo’ trovarsi in forma superavvolta, parzialmente denaturata, o lineare.Le 3 forme, a parità di dimensioni, migrano con velocità diverse
VoltaggioLa velocità di migrazione è proporzionale al voltaggio applicato. Per avere ottima risoluzione il gel deve essere sottoposto ad un voltaggio 5V/cm
Composizione del tampone di corsaLa presenza di ioni in soluzione permette la conducibilità elettrica e quindi la mobilità elettroforetica del DNA. Soluzioni tampone contengono EDTA (1-2 mM) pH 8, Tris-borato(TBE)/Tris-acetato(TAE), 50 mM. L’agarosio viene sciolto nello stesso tampone di corsaLoading buffer 6x0.25 % blue di bromofenolo (migra alla stessa vel. di un frammento di 300 bp)0.25% xilene cianolo (migra alla stessa vel. di un frammento di 4000 bp)30% gliceroloBande corrispondenti ai frammenti di DNA si visualizzano utilizzando EtBr (1 g/ml) un colorante fluorescente che si intercala alle basi del DNA e può essere rivelato attraverso luce UV.
NH2
N+
NH2 CH3
+BrTBE 10x pH 8.8
Acido borico 27.5 gTrizma base 162.0 gEDTA 9.5 gH2O fino a 0.5 l





Isolamento da gel del frammento della PCR
Preparazione gel agarosio 2%Sciogliere 1.0 g di agarosio in 50 ml TBE 1× scaldando su piastra. Quando l’agarosio è fuso (diventa limpido) aggiungere 5.0 l di soluzione di EtBr 10 mg/ml (già pronta, maneggiare con guanti). Versare la soluzione tiepida nella vaschetta da gel in cui è stato inserito il pettine. Caricare su gel i seguenti campioni: 1) 100.0 l frammento PCR + 20.0 l loading buffer 2) 6.0 l marker (100 bp) containing loading buffer Far correre a 100 V per 30-40’. Osservare la banda corrispondente al frammento di gene amplificato corrispondente a 473 bp. Tagliare con il bisturi il pezzetto di gel contenente la banda e trasferirlo in un eppendorf sterile (dopo aver determinato la tara di quest’ultimo).Estrarre il DNA dal gel con il QIAquick Gel extraction kit.

Preparazione di un gel di agarosio

Estrazione del DNA da gel Il prodotto viene estratto da gel con il QIAquick gel extraction kit.Si utilizza per DNA da 70 bp a 10 Kb. In ogni colonna si possono processare fino a 400 mg di agarosio.
Si basa sullo stesso principio del protocollo di purificazione della PCR.In questo caso il gel deve essere fuso in precedenza in presenza di una soluzione (buffer QG) che solubilizza il gel e assicura il successo legame del DNA alla membrana di gel di silice.

Estrazione del DNA da gel

Digestione del prodotto della PCR con enzimi di restrizione
gene
vettore di espressione
SalIHindIII
Il prodotto della PCR deve essere tagliato con gli enzimi di restrizione prima di essere clonato nel vettore pPHSOD1Iq, tra i siti HindIII e SalI
5’-attcccaagctttagcgatggcgacgaaggc…
3’-taagggttcgaaatcgctaccgctgcttccg..
…………………………………….
gatcgcccaataagtcgacgcgtgacc-3’
ctagcgggttattcagctgcgcactgg–5’
HindIII
SalI

Digestione del prodotto della PCR con enzimi di restrizione
Digestione del prodotto della PCR con gli enzimi HindIII e SalI
H2O 14.0 l
Buffer 2 (10) 5.0 lFrammento PCR 30.0 lHindIII (20 U/ml) 1.0 l
50.0 lO/N 37°C
H2O 38.5 l
Buffer x Sal (10) 10.0 lFrammento PCR tagliato 50.0 lBSA100 10 mg/ml 0.5 lSalI (20 U/ml ) 1.0 l
100.0 lO/N 37°C

Tabella di compatiblità per la doppia digestione
seq = sequenziale

Digestione enzimatica “close to the end of DNA fragments”

La ligasiCosa fa:
Quando si usa nel gene cloning:

La ligasiScarsa efficienza perché la ligasi è incapace di tenere unite le sue molecole di DNA, quindi dobbiamo avere alta conc. di DNA per avere alta resa
Molto più efficiente perché si forma un o stabile intermedio sul quale la ligasi può reagire. Più lunghe sono le sticky ends e maggiore è l’efficienza della reazione

La DNA ligasi più utilizzata è quella codificata dal batteriofago T4, un enzima, ATP-dipendente, ottenuto da cellule di E. coli infettate dal fago T4. La T4 DNA ligasi è capace di legare tra loro estremità coesive e, seppur a minor efficienza, è capace di ligare tra loro anche estremità piatte.
La DNA ligasi di E.coli, un enzima NAD+-dipendente presente in E. coli, può legare tra loro soltanto estremità coesive ed è relativamente poco utilizzata.
Le estremità “coesive”, che contengono cioè corti filamenti a singolo filamento complentari tra loro, che vengono generate da molti enzimi di restrizione, facilitano il compito della ligasi, perché le estremità tendono ad appaiarsi tra loro. In queste condizioni le ligazioni si effettuano generalmente tra 4 e 37°C, per un tempo variabile tra 1 e 24 ore.
Se, invece, bisogna ligare, estremità “blunt” la ligazione è più difficile e si effettua a bassa temperatura e a più elevata concentrazione di enzima e di frammenti da legare per favorire l’incontro delle molecole da legare.

Il meccanismo della ligazione avviene in 3 fasi:
Per l’attività della ligasi sono necessari:•ATP•Un gruppo fosfato al 5’•Un gruppo -OH al 3’
1) La ligasi reagisce con l’ATP per formare un complesso covalente ligasi-AMP
2) La ligasi-AMP reagisce con il P al 5’ e con una reazione di transesterificazione lega l’AMP al fosfato rilasciando l’enzima
3) Il gruppo -OH reagisce con il 5’P e e determina la formazione di un nuovo legame fosfo- diesterico

Ottimizzazione della ligazione
Per ottenere buoni risultati la ligazione deve essere accuratamente ottimizzata rispetto alla:
• temperatura e al tempo di reazione• alla concentrazione totale del DNA• alla concentrazione dell’inserto e del vettore.
Per quanto riguarda la concentrazione è importante sia quella del DNA totale T, che quella dell’inserto I e del vettore V. In linea di massima basse concentrazioni di DNA favoriscono le reazioni di primo ordine come la ricircolarizzazione del vettore, la cui velocità è linearmente proporzionale alla sua concentrazione. D’altra parte, aumentare la concentrazione dell’inserto aumenta la probabilità di avere di avere vettori con inserti multipli .
Riguardo alla temperatura bisogna considerare che si contrappongono due aspetti opposti: la stabilizzazione dell’appaiamento tra estremità coesive, ottimale a basse temperature, e l’attività enzimatica della ligasi, massima a 37°C. Come compromesso si utilizza spesso una temp. di 16°C per 12 ore.

Fosfatasi alcalina
Per ottimizzare la reazione di ligazione, nel senso di evitare una elevata frequenza di ricircolarizzazione del vettore.
AATTCTTAAG -5’P3’OHP-5’
HO-3’
AATTCP-5’HO-3’
TTAAG -5’P3’OHvettore
vettore
inserto
Una strategia per superare questo problema consiste nell’utilizzo di una fosfatasi, come ad esempio la fosfatasi alcalina (BAP), un enzima che rimuove il gruppo fosfato al 5’impedendo così l’azione della ligasi. La defosforilazione del vettore con una fosfatasi impedisce la ricircolarizzazione del vettore, abbassando sensibilmente il background.
Poiché il vettore defosforilato non può essere ligato, inoltre, è possibile utilizzare un eccesso molare di vettore, minimizzando la possibilità di avere inserzioni multiple.

AATTCTTAAG -5’P3’OHP-5’
HO-3’AATTC
TTAAG-OH3’OHHO-
HO-3’vettore vettorefosfatasi
Sebbene un vettore defosforilato non possa essere circolarizzato dalla ligasi, può essereligato ad un inserto sfruttandone il suo fosfato al 5’. L’assenza di fosfati al 5’ del vetto-re produrrà due nicks, uno per filamento. Queste interruzioni a singolo filamento,tuttavia non influenzeranno negativamente la stabilità del costrutto (vettore + inserto) esaranno riparate dai sistemi di riparo di E.coli subito dopo la trasformazione.
AATTCTTAAG -5’P3’OHP-5’
HO-3’
AATTCHO-HO-3’
TTAAG-OH3’OHvettore
vettore
inserto
OH HOOH HOvettore vettoreinserto
+ ligasi

Quando frammenti di DNA sono blunt-ended,Come si procede?
Approccio che utilizza linkers:

Quando frammenti di DNA sono blunt-ended,Come si procede?
Approccio che utilizza adattatori:

Quando frammenti di DNA sono blunt-ended,Come si procede?
La Ligasi è incapace di formare legami fosfodiesterei tra 5’-OH e 3’-OH

Produzione di sticky ends attraverso una coda omopolimerica
Addizione di deossiguanosina attraverso un enzima deossinucleatidil transferasi che aggiunge nucleotidi sul 3’-OH terminale di una molecola di DNA double stranded.
La reazione in (c) non è necessaria nel test tube se l’omopolimero è lungo più di 20 nucleotidi perché è stabile abbastanza da essere introdotto nella cellula batterica ospite dove viene riparata da DNA polimerasi e ligasi

Plasmide di espressione della SOD
Sequenza N-terminale per far esprimere la proteina periplasmatica
Ampicillina

Preparazione del vettore di espressione
Digestione vettore H2O sterile 12.0 l
Buffer 2 (10) 2.5 lVettore pPHSOD1Iq (500 ng/l) 10.0 lHindIII (20 U/l) 0.5 l 25.0 l >1 h, 37 °C H2O sterile 19.0 l
Buffer x Sal (10) 5.0 lBsa (100 g/l)lVettore tagliato con HindIII 25.0 lSalI(20 U/l) 0.5 ll totale>1 h, 37 °C
Defosforilazione vettoreH2O sterile 39.0 l
Buffer 10 AP 10.0 lVettore tagliato 50.0 lAP (1 U/l) 1.0 l
100.0 l 30’, 37 °CPer disattivare l’enzima:15’, 65 °C
AP = Fosfatasi alcalina che rimuove fosfato al gruppo 5’ del DNA

Isolamento da gel del vettore
Isolamento da gel del vettore
Preparazione gel agarosio 1%Sciogliere 0.5 g di agarosio in 50 ml TBE 1× scaldando su piastra. Quando l’agarosio è fuso (diventa limpido) aggiungere 5.0 l di soluzione di EtBr 10mg/ml (già pronta, maneggiare con guanti). Versare la soluzione tiepida nella vaschetta da gel in cui è stato inserito il pettine con i denti uniti a due a due. Caricare su gel i seguenti campioni: 1) 6.0 l marker (1000bp) containing loading buffer 2) 100.0 l vettore defosforilato + 20.0 l loading buffer 3) 1.0 l vettore pPB10 non tagliato + 4.0 l H2O + 1.0 l loading buffer
Far correre a 100 V per 30-40’. Osservare la banda corrispondente al frammento di vettore di 5109 paia di basi.Tagliare con il bisturi il pezzetto di gel contenente la banda e trasferirlo in un eppendorf sterile (dopo aver determinato la tara di quest’ultimo).Pesare l’eppenderf con il DNA

Determinazione della concentrazione del DNA
La concentrazione di una campione di DNA puo’ essere fatta in 2 modi1) Valutazione dell’intensita’ della banda su gel di agarosio e confronto con standard
2) Per via spettrofotometrica.50 g/ml DNA a doppio filamento Abs260 = 140 g/ml DNA a singolo filamento Abs260 = 1Il rapporto tra Abs260/Abs280 fornisce una stima della purezzaCampione puro Abs260/Abs280 1.8
Quando DNA conc. è minore di circa 20 ng intensità della banda con EtBr è debole o non visibile
Purine assorbono < 260 nm; pirimidine = 260 nm

Quantificazione su gel del frammento e del vettore
Preparazione gel agarosio 2%Sciogliere 1.0 g di agarosio in 50 ml TBE 1× scaldando su piastra. Quando l’agarosio è fuso (diventa limpido) aggiungere 5.0 l di soluzione di EtBr 10 mg/ml (già pronta, maneggiare con guanti). Versare la soluzione tiepida nella vaschetta da gel in cui è stato inserito il pettine.
Caricare su gel i seguenti campioni: 1) 4.0 l frammento PCR + 6.0 l H2O + 2.0 l loading buffer
2) 4.0 l vettore + 6.0 l H2O + 2.0 l loading buffer
3) 1.0 l marker (100 bp) + 4.0 l H2O + 1.0 l loading buffer
4) 1.0 l marker (1 Kbp) + 4.0 l H2O + 1.0 l loading buffer
Confrontando l’intensità delle bande 1) e 2) con quelle presenti nel campione 3), a quantità nota, valutare la concentrazione del frammento PCR e del vettore.

Reazioni di ligasi tra frammento e vettore
Reazione di ligasi
1 (1:5) 2 (1:20) 3 (controllo)
H2O 8 – (X+Y1) 8-(X+Y2) 8-X Buffer x T4 ligasi 1.0 l 1.0 l 1.0 l Vettore (20 ng) X l X l X l Frammento Y1 l Y2 l -- T4 ligasi 400 U/l 1.0 l 1.0 l 1.0 l Totale 10.0 l 10.0 l 10.0 l 2h, 16°C
Si usano 20.0 ng di vettore. La reazione, catalizzata dall’enzima T4 DNA ligasi avviene a pH 7.5, in presenza di MgCl2e ATP
La quantità di frammento da clonare viene stimata tenendo conto che si useranno frammento e vettore nei rapporti molari 5:1 e 20:1.Vettore 5109 bpFrammento 473 bp 20 ng : X = 5109 : 473 X (ng di frammento) = (20 x 473)/5109 = 1.85 ng 5X = 9.2 ng20X = 37.0 ng
>