3 RNA proteine codice 2009 2010 · TRASCRIZIONE RNA (linguaggio chimico=nucleotidi) TRADUZIONE...
Transcript of 3 RNA proteine codice 2009 2010 · TRASCRIZIONE RNA (linguaggio chimico=nucleotidi) TRADUZIONE...
Il DNA funziona da stampo per la sintesi di molecole di RNA.
In questo modo l’informazione genetica
diventa direttamente utilizzabile per la cellula
Capacità di esprimersi dell’informazione genetica
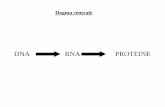
DOGMA CENTRALE
DNA
(linguaggio chimico=nucleotidi)
TRASCRIZIONE
RNA
(linguaggio chimico=nucleotidi)
TRADUZIONE
PROTEINE
(linguaggio chimico=aminoacidi)
mRNA:
porta l'informazione per la sintesi di una determinata proteina.
tRNA:
molecola adattatrice - ogni tRNA è specifico per il trasporto di un determinato aminoacido – converte sequenze di nucleotidi in sequenze di amminoacidi
Ribosoma:
officina che permette il corretto appaiamento tra mRNA e i diversi tRNA e partecipa attivamente alla formazione della catena proteica.
INTERAZIONE TRA mRNA, tRNA e RIBOSOMI
Nel piano: il filamento 5'→→→→3' assume una forma detta a quadrifoglio
-zone a bracci in cui le basi sono appaiate
-anse senza appaiamento per basi modificate post-trascrizionalmente
Iniziando dal 5‘:
- ANSA D: vi si lega l'enzima aminoacilsintetasi specifico per legare un determinato aminoacido al suo tRNA.
- ANSA dell'ANTICODONE: contiene una tripletta complementare e antiparallela al codone che sull'mRNA codifica per un determinato aminoacido.
- Un'ansa variabile
- ANSA del TϕϕϕϕC (ϕϕϕϕ=pseudouracile): necessaria per il legame tra il tRNA e rRNA 5S presente nella subunità grande del ribosoma.
Il tRNA termina al 3' con un codone CCA
(aggiunto postrascrizionalmente)
a cui viene legato l'aminoacido specifico di quel tRNA.
STRUTTURA MOLECOLARE DEL tRNA
Il tRNA nello spazio si ripiega in una struttura detta a L rovesciata dove rimangono bene accessibili l'ansa dell'anticodone e l'aminoacido legato.
Esempio di reazione di legame dell'aminoacido al tRNA
aminoacilsintetasi leu(specifico per leucina)
tRNA + LEUCINA+ ATP tRNALEU + AMP+P+P
(energia)
L'aminoacido con il suo gruppo carbossilico (-COOH) si lega all'-OH del C3 del ribosio del nucleotide (A) al 3' del tRNA.
Le aminoacil-sintetasi sono 20 una per ogni aminoacido.
Ognuna trasferisce un AA diverso sul rispettivo tRNA.
I tRNA sono più di 20 (circa 30-40 in procarioti e anche 50 in Eucarioti) per cui ad alcuni aminoacidi corrisponde più di un tRNA.
STRUTTURA MOLECOLARE DEL RIBOSOMA
Il ribosoma ha una struttura molecolare complessa formata da rRNA e proteine ribosomiali.
In eucarioti gli rRNA vengono prodotti nel nucleolo (tranne il 5S prodotto nel nucleo) dove entrano anche le proteine ribosomialiprodotte nel citoplasma che si assemblano con i rispettivi rRNA per dare i ribosomi.
In procarioti tutto ciò avviene nel citoplasma.
I ribosomi procariotici sono più piccoli dei ribosomi eucariotici.
Le differenze tra ribosomi procariotici ed eucariotici sono dovute al numero e alle grandezze diverse degli rRNA e al numero e alle grandezze diverse delle proteine.
I ribosomi sono formati da due subunità (piccola e grande) che si assemblano solo al momento della traduzione (le due subunitàribosomiali si assemblano e diventano funzionali solo in presenza di mRNA).
IN PROCARIOTI
Il filamento di mRNA inizia con una sequenza detta LEADER, che non viene tradotta in proteina, contenente la sequenza di Shine-Delgarno.
Tale sequenza si appaia durante la traduzione con l'rRNA 16S.
La sequenza codificante inizia sempre con la tripletta AUG e termina con una tripletta detta di STOP.
Segue un tratto detto TRAILER (rimorchio) non codificante.
I geni che codificano per proteine sono POLICISTRONICI (ad un unico promotore seguono tutti i geni di una determinata via metabolica per cui vengono trascritti tutti contemporaneamente).
IN EUCARIOTI
L'mRNA ha struttura simile a quella dei Procarioti.
In aggiunta:
-5’ CAP (7-metilguanosina): si legherà all'rRNA 18S durante la traduzione
-coda di poliA al 3'.
I geni che codificano per proteine sono MONOCISTRONICI (ciascun gene ha il suo proprio promotore).
STRUTTURA DEGLI mRNA
Perché avvenga la traduzione è necessario passare da un linguaggio fatto di NUCLEOTIDI ad un linguaggio di AMINOACIDI.
Gli aminoacidi presenti in tutte le proteine sono 20 mentre le basi sono solo 4.
Se ogni base specificasse per 1 amminoacido, avremmo solo 4 aminoacidi specificati 41=4.
Se la combinazione di due basi specificasse per 1 aminoacido, avremmo:42=16 aminoacidi specificati.
Se la combinazione di tre basi specificasse per 1 aminoacido, avremmo: 43=64 combinazioni per 20 aminoacidi.
Il codice pertanto è a triplette, cioè la combinazione di tre basi specifica per 1 aminoacido.
Ogni tripletta è detta CODONE.
Esistono 3 triplette non codificano per nessun aminoacido e sono dette di STOP (UAG; UAA; UGA).
IL CODICE GENETICO
Poiché ci sono 64-3 (codoni di stop)=61 triplette per 20 aminoacidi si dice che il codice è DEGENERATO o RIDONDANTE cioè ogni aminoacido può essere specificato da più di un codone.
Esempio: leucina 6 codoni, valina 4 codoni, triptofano e metionina 1 codone, etc…. (Si può osservare inoltre che i diversi codoni per un aminoacido sono diversi soprattutto nella 3 base).
CARATTERISTICHE DEL CODICE GENETICO
�Il codice genetico è UNIVERSALE cioè uguale per tutti gli esseri viventi tranne alcune eccezioni.
�Il codice genetico NON E' AMBIGUO cioè un determinato codonespecifica per un solo aminoacido.
�Il codice genetico è senza punteggiature cioè viene letto linearmente (di tre basi in tre basi) e non è sovrapponibile (es. la stessa base non può essere letta come l’ultima di un codone e la prima del successivo).
MUTAZIONI PUNTIFORMI IN ZONE CODIFICANTI
una base
-Transizione da purina a purina (A o G) o da pirimidina a pirimidina (C o T)
-Trasversione da purina a pirimidina o viceversa
Conseguenze:
1) nessuna se ad essere colpita è la terza base di un codone e il nuovo codone codifica per lo stesso aminoacido
2) cambio dell'aminoacido specificato dal codone ora mutato
3) proteina più corta se un codone per l'aminoacido si tramuta in un codone di STOPEs. UUA→→→→UAA
(Leu→→→→ stop)
4) proteina più lunga se ad esempio da un codone di stop per mutazione si passa ad un aminoacido qualsiasi
B) DELEZIONE o INSERZIONE di una base
Cambio della cornice di lettura. Da quel nucleotide (deleto o inserito) la proteina saràtutta diversa perché a codoni diversi corrisponderanno AA diversi
AUGCAACCCGGAUAAGCUUAA
(delezione)
AUGCAACCCGGUAAGCUUAA…
1) La lettura dell'mRNA procede in direzione 5'→3‘
2) La sintesi proteica procede dall'estremità N-terminale all'estremità C-terminale del polipeptide
3) La traduzione ha inizio sempre conFormil-metionina in ProcariotiMetionina in Eucarioti.
Il codone per Metionina è AUGL'anticodone è UAC.
TRADUZIONE
1) INIZIO:
L'mRNA si lega alla SUBUNITA' PICCOLA ribosomiale e al tRNAmet (codonecon anticodone) grazie a fattori di inizio (3 in procarioti, 7/8 in eucarioti) e ad 1 GTP→→→→GMP+P+P che dona energia necessaria per il legame.
L'mRNA in procarioti si lega con la sequenza SHINE-DALGARNO al 16S rRNAmentre in eucarioti l'mRNA si lega con il cappuccio (5-metilguanosina) al 18S rRNA.
Successivamente si assembla la SUBUNITÀ GRANDE contenente due siti detti A e P per accogliere i tRNA.
Solo il 1° tRNAmet si lega direttamente al sito P.
2) ALLUNGAMENTO
Il 2° tRNA con l‘AA corrispondente si lega all'mRNA (2°codone/anticodone) e alla subunità grande, nel sito A del ribosoma, grazie a fattori di allungamento (in Procarioti Ts e Tu, in Eucarioti Ef1-Ef1β) e ad 1 molecola di GTP che si scinde donando energia.
Il legame che si forma tra i due AA avviene tra il gruppo carbossilico (-COOH) della metionina e il gruppo amminico (-NH2) del 2° AA (legame peptidico).
Enzima responsabile è la Peptidil-transferasi.
Dopo la formazione del legame peptidico il 1° tRNA, rimasto scarico della metionina, viene allontanato dal sito P.
Avviene quindi la traslocazione:
l'mRNA scivola di un codone trascinandosi il 2° tRNA che passa dal sito A al sito P, lasciando il sito A libero per l'attacco del 3° tRNA.
Questa traslocazione è mediata da un fattore di traslocazione e dalla scissione di una molecola di GTP.
Il ciclo si ripete tante volte quanti sono gli AA che devono essere legati.
3) TERMINAZIONE
Quando sul sito A si trova un codone di STOP, a cui non corrisponde nessun tRNA, la sintesi si arresta.
Inoltre esistono fattori di rilascio che si legano al sito A impedendo comunque l'attacco dei tRNA.
La catena polipeptidica si stacca dall'ultimo tRNA grazie ad un enzima (idrolasi) con consumo di una molecola di GTP. Le due subunità ribosomiali si disassemblano.
IL VACILLAMENTO DELLE BASI
Il legame tra codone e anticodone è complementare e antiparallelo.
Esempio: 3'CAG5' (anticodone)
5'GUC3' (codone)
Il numero dei codoni (61) è più grande di quello degli anticodoni.
61 codoni (3 sono codoni di stop) mentre i tRNA sono circa 30-40 in procarioti e anche 50 in Eucarioti)
Questo implica che alcune molecole di tRNA con il loro anticodone sono capaci di accoppiarsi a più di un codone.
Alcuni tRNA hanno una struttura tale da richiedere un appaiamento accurato nelle prime due posizioni del codone (5') e da tollerare un appaiamento scorretto (oscillante) in terza posizione.
Questo appaiamento approssimativo rende possibile combinare 20 AA ai loro 61 codoni servendosi per esempio di solo 30/50 molecole di tRNA.
Frequentemente al 5' dell'anticodone (corrispondente alla terza base del codone) esiste la base modificata inosina capace di complementarsisia con A, C, U.
BILANCIO ENERGETICO
DELLA TRADUZIONE
Esempio proteina di 100 aa
1 GTP per il complesso di inizio
99 GTP per l'attacco dei 99 tRNA
99 GTP per la traslocazione dei 99 tRNA
1 GTP per il rilascio della proteina
Totale 200 GTP che corrisponde a 2 GTP per ogni aminoacido.
Riassumendo 1AA=2GTP
Le proteine sono dei polimeri di aminoacidi detti anche catene polipeptidiche.
Gli aminoacidi possono essere destrogiri (D-) e levogiri (L-).
Negli organismi viventi sono presenti solo L-aminoacidi. Gli aminoacidi sono 20.
Ciascun aminoacido è costituito da un Cαααα a cui sono legati un gruppo carbossilico (COOH), un gruppo amminico (NH2), un H, e un gruppo variabile detto radicale. I radicali conferiscono adogni aminoacido la propria specificità.
PROTEINE
-APOLARI (idrofobici) che contengono nel radicale un gruppo idrofobico ( es.-CH3)
-POLARI (idrofilici) che si suddividono in:
POLARI ACIDI che contengono nel radicale un gruppo acido (es.-COOH),
POLARI BASICI che contengono nel radicale un gruppo basico (es.-NH2),
POLARI NON CARICHI che contengono nel loro radicale sia un gruppo acido che un gruppo basico o comunque un gruppo idrofilico per cui il loro pH è neutro.
Gli aminoacidi tra loro sono legati con un legame peptidico che si forma tra il gruppo COOH di un aminoacido e il gruppo NH2 del successivo aminoacido con l'eliminazione di una molecola di acqua.
Questo legame è un legame covalente più forte perché più breve del singolo legame e quindi si avvicina ad avere le caratteristiche di un doppio legame.
Le proteine presentano tre livelli di struttura e alcune anche un quarto livello.
STRUTTURA PRIMARIA
E' rappresentata dalla sequenza lineare degli aminoacidi legati da legami peptidici determinata dal gene.
STRUTTURA SECONDARIA
Gran parte delle proteine, anche se in alcuni punti hanno una struttura irregolare, presentano lunghi tratti con una struttura regolare ad αααα-elica o a foglietto pieghettato ββββ.
L'αααα-elica e il foglietto pieghettato ββββ sono dati da legami ad H che si instaurano tra differenti gruppi peptidici.
Nell' αααα-elica l'ossigeno carbossilico di ciascun legame peptidico forma un legame ad idrogeno con l'idrogeno del gruppo amminico dell'aminoacido che si trova quattro residui più avanti nella sequenza lineare.
Nel foglietto pieghettato ββββ i legami ad H si instaurano tra gli atomi che formano i legami peptidici appartenenti a catene polipeptidiche diverse o a porzioni dello stesso polipeptide ripiegato su se stesso.
LE PROTEINE E LORO STRUTTURA
STRUTTURA TERZIARIA
La struttura terziaria è determinata da legami che si formano tra radicali di diversi aminoacidi che organizzati nelle strutture secondarie vengono a trovarsi vicino.Tali legami possono essere di tutti i tipi ( ionici, covalenti, ad idrogeno, idrofobici etc…) e determinano la struttura tridimensionale della catena polipeptidica (es. proteine globulari).
La struttura terziaria viene modificata inoltre dalle interazioni degli aminoacidi con l'ambiente in cui la catena polipeptidica si trova.
(es. proteine di membrana tenderanno ad esporre all'esterno gli aminoacidi idrofobici capaci di interagire con i lipidi ).
STRUTTURA QUATERNARIA
Alcune proteine specialmente quelle con funzione enzimatica, possono assumere una struttura quaternaria che deriva dall'associazione di più catene polipeptidiche.
Se le catene polipeptidiche sono uguali si parlerà di dimeri (2 catene polipeptidiche), tetrameri (4), etc…
Le catene che si associano possono essere differenti e ciascunaprende il nome di subunità (vedi RNA-polimerasi in procarioti).
Considerando la definizione di introni indicare il rapporto corretto in ogni gene tra il numero degli esoni e degli introni:
a) Esoni= introni +1
b) Esoni=introni
c) Esoni= introni –1
d) Esoni= 2 volte gli introni
La TATA box è una sequenza presente:
1. Al 5’ dei geni in procarioti
2. Al 5’ dei geni per mRNA in eucarioti
3. Sull’ mRNA nella sequenza leader
4. Al 5’ dei geni per rRNA in eucarioti
5. Al 5’ dei geni per tRNA in eucarioti
Descrivete le possibili conseguenze nella traduzione del seguente mRNAmutato rispetto a quello nativo
5’ UUCCCAAUCACAUAAGUAGCC 3’ RNA mutato
5’ UUCCCAAUCACAUACGUAGCC 3’ RNA nativo
(codoni di stop UAG, UAA, UGA)
La polimerasi I è la polimerasi che trascrive:
1. Tutti gli RNA in procarioti
2. tRNA e 5S ribosomiali in eucarioti
3. Gli snRNA e si trova nel nucleolo
4. Gli rRNA e si trova nel nucleolo
5. Gli hnRNA in eucarioti
RNA-POLIMERASI I
Si trova nel nucleolo
1) Trascrive un filamento precursore 45S da cui vengono poi ritagliati i tre rRNA: 28S, 18S, 5,8S.
2) Il DNA contenente i geni per gli rRNA si trova nel nucleolo.
3) La trascrizione avviene nel nucleolo
4) Le proteine ribosomiali vengono prodotte nel citoplasma e successivamente rientrano nelnucleo e quindi nel nucleolo dove si assemblano con i rispettivi rRNA per dare le due subunità ribosomiali.
1) La polimerasi I ha bisogno di 2 fattori trascrizionali generali (B ed S) per poter iniziare la trascrizione che si legano al promotore (da -100/-50 a +20).
RNA-POLIMERASI III
Si trova nel nucleo.
1) Trascrive i diversi tRNA e l' rRNA 5S (unico ribosomiale non prodotto nel nucleolo dove poi migrerà).
2) La polimerasi III ha bisogno di fattori di trascrizione generali per iniziare (A,B,C per rRNA 5S e B,C per i tRNA).
3) Le zone regolatrici di attacco dei fattori trascrizionali e RNA-polimerasi non sono a monte del trascritto ma a valle: Promotori Interni.
RNA-POLIMERASI II
Si trova nel nucleo.
1) Trascrive RNA primari (hnRNA) che successivamente verranno modificati (maturazione) per dare mRNA funzionanti.
2) Per iniziare ha bisogno di fattori trascrizionali generali (D, B, F, E, H).
3) Tali fattori si legano ad una sequenza del promotore detta TATA BOX (-40).
Esistono anche a monte della TATA BOX sequenze ricche di CG e CAAT BOX .