09 Visualizzazione molecolare -...
-
Upload
nguyenkhue -
Category
Documents
-
view
220 -
download
0
Transcript of 09 Visualizzazione molecolare -...

Visualizzazione
Laboratorio di Bioinformatica I
Visualizzazione Molecolare
Dott. Sergio Marin Vargas (2014 / 2015)

PDBsumhttp://www.ebi.ac.uk/pdbsum/
Ricerca per codice PDBcodice PDB
Ricerca testuale
Ricerca per sequenza
Ricerca per diversi databases

PDBsumhttp://www.ebi.ac.uk/pdbsum/
Molta informazione su struttura secondaria, ligandi e interazioni con altre proteine
PROCHECK:RamachandranPlot
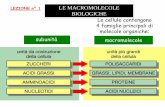
Proteine e catene all’interno della struttura risolta (file PDB)
Annotazione funzionale Gene Ontology (GO)
Ligandi

Ramachandran PlotCarbonio
Alfa
Angolo diedro Psi
Angolo diedro Phi
Angolo diedro
Angolo diedro Omega
Serve a verificare per ciascun aminoacido se la struttura risolta o modellata è concorde agli angoli Psi e Phi

Uppsala Ramachandran Serverhttp://eds.bmc.uu.se/ramachan.html
5
Codice PDB

Distanze di legame
Legame a idrogeno: 2.5-3.5 Å
Interazioni di van der Waals: 3-4 Å
Legame ionico (ponte salino): 2.8 Å
Interazioni di stacking: 4 Å
6
Interazioni di Stacking: circa 4 Å
Interazioni di stacking: 4 Å

Esercizio 1: UNIPROT-PDBsum
La DNA-glicosilasi umana (una particolare liasi), è unenzima che catalizza la rottura di svariati legami chimiciproducendo un nuovo doppio legame o una nuovastruttura aromatica. Ricercare in UNIPROT la DNA-
glicosilasi umana il cui gene è OGG1.
• Qual è il codice uniprot della proteina?• Qual è il codice uniprot della proteina?• Quanto è lunga la proteina?• Quante strutture esistono di questa proteina?• Qual è l’entry PDB a maggiore risoluzione?• Esistono strutture 3D della proteina intera?• Visualizzare in PDBsum la struttura di 2XHI (ci sono
link direttamente da UNIPROT?)• Scaricare il file PDB di 2XHI e analizzarlo (usare il link
diretto a PDB che c’è in PDBsum).• Ci sono atomi di idrogeno nella struttura? Perchè?

Esercizio 2: Ramachandran Plot
Visualizzare l’entry 1EBM in PDBsum• Questa proteina contiene α-eliche? β-sheets? Loops? Quali
elementi di ss sono più numerosi?• Quanti domini ci sono?
Cliccare sulla figura di Procheck per visualizzareCliccare sulla figura di Procheck per visualizzareil Ramachandran plot.• Da questo grafico se può intuire se la proteina contiene α-
eliche? β-sheets? Loops? E quali sono più numerosi?• I residui di Gly (triangoli blu) sono distribuiti diversamente
dagli altri aminoacidi (quadratini blu)? Perché?• Un solo residuo è mostrato in rosso. Quale? In che elemento
di ss è presente? Idee sul perchè?

Esercizio 2: Ramachandran Plot
GlicineAcido Aspartico 174posizionato nella regione gialla, una regione permessa ma non è quella comune per questo aminoacido!

Esercizio 3: PDB entry
Visualizzare l’entry 1EBM nel database PDB.- Quali macromolecole contiene la struttura?- Quante catene ha la struttura principale? Cosa
rappresenta la catena A?- La proteina è mutata? Se si, Quale mutazione?- La proteina è mutata? Se si, Quale mutazione?- E’ una proteina intera? Mancano residui?
Perchè?- Cliccare sul tab Sequence. Che informazioni
troviamo?

Visualizzare l’entry 1EBM nel database PDB edscaricare il file PDB che descrive la proteina.• Chi sono gli autori del lavoro strutturale?• Si tratta di cristallografia a raggi X o di NMR?• Qual è la risoluzione della struttura?• Cosa si trova al REMARK 200? Hanno usato
Esercizio 4: PDB file
• Cosa si trova al REMARK 200? Hanno usatoluce di sincrotrone per risolvere la struttura?
• Cosa si trova al REMARK 470?• Cosa si tova nel campo SEQRES?• Quante α-eliche e β-sheets ci sono?• Trovare le coordinate del carbonio alfa di
Asp174.

UCSF Chimerahttp://www.cgl.ucsf.edu/chimera/

PyMOLhttps://www.pymol.org/http://pymolwiki.org/index.php/Main_Page
Un programmautilità per lautilità per lavisualizzazione dimacro-molecole,il quale utilizzacome input un filein formato PDB.

PyMOL
Finestra menu
Menu della proteina: (A)ction, (S)how, (H)ide, (L)abel, (C)olor
Finestra principale

Esercizio 5: PyMOL introduzioneLanciamo PyMOL ed apriamo il file 1EBM.pdb precedentemente salvato.Prendiamo dimestichezza con il programma:• Mostriamo la sequenza: Display � sequence• Usiamo il mouse per ruotare la molecola (tenere premuto il
pulsante sx) e per lo zoom (tenere premuto il pulsante dx).Proviamo lo scroll: che fa?Proviamo lo scroll: che fa?
• Cosa sono le crocette rosse? Riusciamo a nasconderle?• Nascondiamo tutta la struttura cliccando a destra sul codice
PDB, poi selezionamo le varie modalità di rappresentazione in“S” (Show As): lines, sticks, ribbon, spheres, surface, perrasconderle in “H” (Hide). Che differenza c’è tra l’una el’altra?
• Provare “lines” e colour “Gray 60”, poi scegliere “by elements”.• Provare a colorare per catena e visualizzare come “cartoon”.

Esercizio 6: PyMOL selezione aaAprire il file 1EBM mediante PyMOL:• Nascondere tutto ciò che è visualizzato e scegliere “show
as cartoon” poi colorare “by secondary strucure”.• Quanti β-sheets ci sono? • Quanti filamenti β contiene ciascun foglietto?• Dal server Uppsala Ramachandran (http://eds.bmc.uu.se/ramachan.html)
generare il plot di Ramachandran di questa proteina. Quantigenerare il plot di Ramachandran di questa proteina. Quantioutlier ci sono?
• Visualizzare la sequenza della proteina in PyMOL.• Asp174 è un outlier nel plot di Ramachandran. Localizzare
il residuo nella sequenza di PyMOL e cliccarlo. Esso viene selezionato nel panel a destra (scrita “sele).
• Rinominare la selezione: Scegliere “Action�renameselection” poi digitare il nome: Asp174
• Visualizzare l’amino acido selezionato come “sfere” e colorarlo per elemento

Esercizio 7: PyMOL surface
Aprire il file 1EBM mediante PyMOL:• Rappresentare 1EBM come “surface”.• Il residuo Asp174 si trova sulla superficie?• Rappresentare 1EBM come “struttura secondaria” e
colorarlo di rosso.• Selezionare i residui di triptofano con il comando: “select
TRP, resn trpTRP, resn trp” rappresentarli in stick e colorarli di giallo.• I Trp sono prevalentemente esposti al solvente? Perchè?• Rappresentare nuovamente 1EBM come “surface” e
cambiare il setting della trasparenza dal menù in alto: Setting�Transparency�Surfare�50% e visualizzare nuovamente i triptofani.
• Il residuo Trp47 è esposto al solvente? Da cosa lo capisco? Cosa succede con il Trp313?
• Salvare l’image della proteina in formato PNG.

Esercizio 8: PyMOL interazioneScaricare e visualizzare la struttura corrispondente al PDB entry 1BRS.• Di che struttura si tratta?• Quante catene sono presenti? Si può dire che si tratta di
un esamero? Perchè?Creare due nuovi oggetti corrispondenti alle catene A e D, mediante il comando “select cat_A/D, chain A/D”, poi sulleselezioni generate fare “A�copy object” e poi rinomimare iselezioni generate fare “A�copy object” e poi rinomimare inuovi oggetti. Cancellare l’oggetto 1BRS “A�delete object”.• Quanti foglietti beta ci sono nella catena D?• Identificare una coppia di residui dell’interfaccia proteina-
proteina che fanno un legame di stacking?• Qual è la distanza (Wizard�Measurement) più corta tra
un qualsiasi atomo di His102 (cat.A) e un qualsiasi atomodi Tyr29 (cat. D)?

Esercizio 8: PyMOL interazione
H102 (Cat A) Y29 (Cat D)

VMDhttp://www.ks.uiuc.edu/Research/vmd/

VMDhttp://www.ks.uiuc.edu/Research/vmd/
Finestra MenuPrincipale
Caricare molecole
Rappresentazione
Finestra Display
Rappresentazione grafica

VMD tutorialhttp://www.ks.uiuc.edu/Training/Tutorials/vmd/tutorial-html/
R � modalità rotazioneT � modalità traslazioneS � modalità scalaC � Per centrare dove c’è il mouse

Esercizio 9: VMD introduzioneScaricare la struttura del complesso transferrin receptor 1 etransferrin legato al ferro (codice PDB 3S9L).• Quante catene ha la struttura? Quale catena appartiene a quale proteina?• In “Graphics�Representations”, cliccando sulla selezione “all”, mettere
“Drawing Method” a “NewCartoon” e “Coloring Method” a “Chain”.• Selezionare solo la catena “A” mediante “Create Rep” poi scrivere “chain
A” e con doppio click su “all” nascondere tutta la proteina. Quale colorecorrisponde a quale catena?
• Analizzare il file PDB, a quale numero di residuo “#” corrisponde lo ione• Analizzare il file PDB, a quale numero di residuo “#” corrisponde lo ione“Ca++” e a quale catene è legato?
• In VMD evidenziare il “Ca” mediante “Create Rep” poi scrivere “resid #”,poi “Drawing Method” a “VDW” (van der walls) e “Coloring Method” a“ColorID” e poi scegliere giallo.
• Mettere a trasparenza le catene.• Analizzando la struttura in PDBsum a quali aminoacidi è legato il Ca?• L’informazione riportata su PDBsum è veritiera? Cioè verificare nella
struttura 3D che gli aminoacidi indicati siano negli intorni del Ca(Suggerimento: “Drawing Method” � “Licorice” e “Coloring Method” �
“Element” poi etichettarli con “Label”).

Esercizio 9: VMD introduzione

Esercizio 10: VMD distanze
Con la stessa struttura del complesso transferrinreceptor 1 e transferrin legato al ferro (3S9L)dell’esercizio precedente.• Individuare il numero di residuo corrispondente allo
ione Fe++. A quale catene è legato?• Analizzare la struttura in PDBsum a quali catene è• Analizzare la struttura in PDBsum a quali catene è
legato il Fe? A quali amino acidi?.• Caricare la struttura in VMD, colorare e visualizzare
nel modo più conveniente, visualizzare solo unadelle catene che legano il Fe e poi evidenziare il Fe.
• Trovare la distanza tra il Fe e il carbonio alfa diciascuno degli aminoacidi con cui si lega.(Suggerimento: utilizzare Mouse�Label�Bonds)

Esercizio 11: VMD surfaceCaricare in VMD la struttura dell’emoglobina (PDB 2W6V).• Controllare quante catene ci sono e a cosa corrisponde ogni
catena, individuare il “resid” del gruppo EME (HEM).• In “Graphics�Representations” eliminare le acque con “not
water”.• Creare nuove rappresentazioni una per ogni catena e
colorare le catene A,B,C e D di Blu, Red, Gray e Orangecolorare le catene A,B,C e D di Blu, Red, Gray e Orangerispettivamente utilizzando la trasparenza ed impostare“Drawing method” a QuickSurf.
• Per una visione più chiara dei gruppi EME nasconderecompletamente la struttura “lines” di tutta la proteina (all).
• Creare nuove rappresentazioni per I gruppi “EME”, impostare“Drawing method” a Licorice, colorare per “elemento” emateriale “opaco”.
• Misurare la distanza tra il Fe++ legato al gruppo EME dellacatena B con quello della catena C. Quanti Å ci sono ?

Esercizio 12: VMD interazione
Caricare in VMD la stessa struttura utilizzatanell’esercizio 8 (PDB 1BRS).• Visualizzare solo le catene A e D, visualizzarle
come “New Cartoon” e colorarle come “strutturasecondaria”.
• Utilizzando l’estensione “Extensions � Analysis• Utilizzando l’estensione “Extensions � Analysis� Sequence Viewer” selezionare gli aminoacidiHis102 della catena A e Tyr29 della catena D.
• Qual è la distanza tra il carbonio E1 di His102e il carbonio E1 di Tyr29? Corrisponde alladistanza di un legame di stacking?

Esercizio 12: VMD interazione
H102 (Cat A)
Y29 (Cat A)
Y29 (Cat D)