sequenziamento biotecnologi
-
Upload
pattada3333 -
Category
Documents
-
view
69 -
download
1
description
Transcript of sequenziamento biotecnologi
-
Tecnica di Biologia Molecolare che consiste nel determinare tutte le basi che costituiscono un frammento di DNA.
Ha permesso enormi sviluppi nella Ricerca Scientifica: la decodifica di tutto il Genoma Umano ha chiarito lorigine molecolare di molte malattie ereditarie e diversi meccanismi oncogenici con cui si sviluppano i tumori.
In campo microbiologico e virale disporre della sequenza di virus e batteri ha messo a disposizione metodi di identificazione (PCR) e di tipizzazione per chiarire aspetti legati alla resistenza a farmaci, alla virulenza e alla determinazione di sottotipi.SEQUENZIAMENTO
-
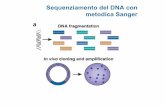
Il metodo Sanger (o metodo a terminazione di catena)
1) Il sequenziamento a terminazione di catena coinvolge la sintesi di nuovi filamenti di DNA, complementari ad uno stampo a singolo filamento Per la sintesi del DNA si utilizzano DNA polimerasi caratterizzate da
Elevata processivitBassa attivit esonucleasica 5-3Bassa attivit esonucleasica 3-5
(es. Klenow, Sequenasi)
-
Il metodo Sanger (o metodo a terminazione di catena)
Nel sequenziamento a terminazione di catena la reazione oltre ad
1) Uno stampo a singola elica
2) un innesco specifico (primer)
3) La marcatura con un dNTP* marcato, di solito con 35S
ha bisogno di:
4) una miscela di ddNTP, uno per ogni reazione di sequenza
-
Dideossi- e deossi- nucleotidi trifosfati
-
La sintesi del filamento complementare, tuttavia non prosegueindefinitivamente, perch la miscela di reazione contiene, in quattrodistinte reazioni, piccole quantit di specifici dideossinucleosiditrifosfati, ddATP, ddTTP, ddCTP e ddGTP, che bloccano lallungamento perch posseggono un solo atomo di idrogeno al posto del gruppo -OH in 3Terminazione della catena
-
Il risultato una serie di frammenti, ciascuno dei quali interrottiin corrispondenza di una delle dCTP presenti nel filamento
-
DNA stampo a singola elicaIbridazione conIl primer + [35S]dATP+dCTP,dGTP,dTT (dNTP) +SequenasiddATP, dNTP ddCTP, dNTP ddGTP, dNTP ddTTP, dNTP-CCG ddA-ddC-C ddC-CC ddG-CCGATT ddG-CCGA ddT-CCGAT ddTSequenza: 5-CCGATTGA C G T-CCGATT ddG-CCGAT ddT-CCGA ddT-CCG ddA-CC ddG-C ddC-ddCGTTA
GCCSchema di sequenziamento a terminazione di catena Direzione dilettura
-
Nuovi metodi di sequenziamento
Il sequenziamento a ciclo termico ( PCR asimettrica)
Il sequenziamento automatizzato con marcatori fluorescenti
Il pirosequenziamento
-
Il sequenziamento a ciclo termico ( PCR asimettrica)Vengono effettuate 4 reazioni di PCR, ognuna contiene un solo primer e uno dei 4 dideossinucleosidi trifosfati. In questo modo si ottiene soltanto il filamento complementare allo stampo. Si generano le consuete famiglie di filamenti a catena terminata che vengono risolti per elettroforesi su gel di poliacrilammide DNA stampoddATPPCR con un solo primerddAddAddAddAddAprimer
-
Il sequenziamento automatizzato con marcatori fluorescentiConiugando a ciascun ddNTPun diverso marcatore fluorescente, possibile effettuare le quattro reazioni di sequenziamento in un unico tubo da saggioe caricare il tutto in un solo pozzetto di gelddAddCddTddG
-
Le emissioni fluorescenti vengono captate da un rilevatore e leinformazioni vengono integrate e trasformate in picchi di colore diverso, con aree proporzionali allintensit di emissione.Si sta diffondendo luso di tubi capillari al posto dei tradizionali gele di procedure robotizzate in tutte le fasi. Nelle migliori condizioni possibile leggere 750.000 bp al giorno
-
DATI STORICI SUL PROGETTO GENOMA UMANO
-1983: i due pi importanti laboratori per lo sviluppo della genetica umana in relazione allo sviluppo delle armi nucleari, a Los Alamos e a Livermore, cominciarono a lavorare al Progetto Biblioteca Genetica. In particolare la DOE (US Department of Energy) era interessata agli effetti delle mutazioni nellambito dei programmi nucleari civili e militari .
-1988: lNIH (US National Institutes of Health) istituisce un organo competente per questa materia all'interno della sua organizzazione, l'Office of Human Genome Research (poi rinominato come National Center for Human Genome Research). Sempre nello stesso anno, il Congresso americano approva in via ufficiale il Progetto Genoma Umano della durata di 15 anni con inizio nel 1991. Inizialmente si stim che sarebbero serviti 15 miliardi di dollari.
-2000: su Science esce la sequenza completa del Genoma di Drosofila Melanogaster, da parte della Celera Genomics, in collaborazione con gli scienziati del Berkeley Drosofila Genoma Project (BDGP). Durante il progetto di sequenziamento hanno identificato 14.000 geni.
-2000: annuncio del completamento della sequenziamento del genoma umano da parte della Celera Genomics, progetto intrapreso da questa societ nel settembre 1999.
Il genoma umano si rivelato composto da 3,12 miliardi di coppie di basi nucleotidiche e 30.000 o 40.000 mila geni (inferiori agli 80.000 140.000 previsti). Il lavoro di assemblamento delle sequenze nucleotidiche stato effettuato grazie allutilizzo di computer per un totale di 20.000 ore.
Dopo il 2000 inizia la Proteomica: studio dellespressione dei geni legati alle proteine.
-
Quali sono gli obiettivi del Progetto?
identificare tutti gli oltre 80.000 geni del DNA umano
determinare la sequenza dei 3 miliardi di basi chimiche che formano il DNA umano
immagazzinare queste informazioni in un database, rendendoli accessibili per ulteriori studi biologici
sviluppare strumenti per le analisi e nuove tecnologie di sequenziamento
studiare i problemi etici, legali e sociali (ELSI) che possono sorgere dal progetto: per questo il DOE e il NIH hanno devoluto tra il 3% e il 5% dei loro budget del Progetto Genoma. Questo rappresenta il pi grande programma di bioetica mai realizzato al mondo, un modello per tutto il mondo scientifico.
-
Il progetto genoma umano (HGP) stato un progetto internazionale della durata di tredici anni, iniziato ufficialmente nell'ottobre del 1990 per scoprire tutti gli 80000 geni che compongono il nostro DNA e renderli accessibili per ulteriori studi biologici; originalmente il progetto doveva durare 15 anni divisi in tre quinquenni, ma il rapido progresso tecnologico lo ha accelerato e ne ha posto il termine prefissato nel 2003.
In parallelo a questo sforzo stato studiato il DNA di una serie di organismi per provvedere alle informazioni comparative necessarie per la comprensione del funzionamento del genoma umano.
Le informazioni generate dal progetto hanno portato benefici nel campo della medicina e ci aiutano a capire ed eventualmente a trattare molte delle oltre 4.000 malattie genetiche che affliggono il genere umano. IL PROGETTO GENOMA UMANO
-
Genomica: branca dellabiologia molecolareche si occupa dello studio del genomadegliorganismiviventi. In particolare si occupa della struttura, contenuto, funzione edevoluzionedel genoma.
-
Proteomica: consiste nell'identificazione sistematica diproteinee nella loro caratterizzazione rispetto astruttura, funzione,attivit, quantit einterazioni molecolari.
-
Il metodo Maxam-Gilbert(o metodo di sequenziamento a degradazione chimica)
Questo metodo, basato sulla degradazione chimica di frammenti di DNA a doppio filamento, ormai poco utilizzato, perch non adattoal sequenziamento automatico e perch utilizza composti chimici dannosi alla salute Si differenzia da tutti gli altri metodi perch non utilizza sintesi enzimatiche ma trattamenti chimici che agiscono a livello di singoli nucleotidi.I frammenti di DNA a doppio filamento possono essere generati per frammentazione o digestione enzimatica. In entrambi i casi le molecole a doppio filamento devono essere convertite in molecole a singolo filamento e marcate terminalmente
-
1 Marcatura terminale al 5 o al 3 del DNA a doppio filamento
2 Denaturazione e separazione dei due filamenti
3 Il DNA a singola elica viene suddiviso in quattro campioni, ognuno dei quali viene trattato con un reagente chimico che demolisce una o due delle 4 basi del DNA. G = DMS + piperidinaG + A = DMS + piperidina + acido formico C + T = idrazina + piperidinaC = idrazina + piperidina in NaCl 1,5 M Le reazioni sono controllate in modo da avere una frammentazione parziale: statisticamente tutti le possibili basi saranno degradate producendo una serie di frammenti la cui lunghezza dipender dalla distanza tra lestremit marcata e il sito di taglio
4 Separazione dei frammenti marcati mediante elettroforesi su gel denaturante e visualizzazione dei risultati mediante autoradiografia
-
************






![sequ.annot.genoma.hs.ppt [modalità compatibilità] · umano è stata la costruzione di una mappa fisica ad alta risoluzione ... Sequenziamento gerarchicoSequenziamento gerarchico](https://static.fdocumenti.com/doc/165x107/5c69c45809d3f2e4178b9590/sequannot-modalita-compatibilita-umano-e-stata-la-costruzione-di-una.jpg)