Predizioni Struttura Proteine
-
Upload
fabiothekingjames -
Category
Documents
-
view
11 -
download
2
description
Transcript of Predizioni Struttura Proteine
-
- 8 -
- Lo studio della struttura delle proteine
mediante metodi computazionali -
Lo scopo della Bioinformatica e della Biologia Computazionale quello di offrire
strumenti e metodologie capaci di gestire ed analizzare la grande quantit di
informazioni prodotte nel campo della ricerca biologica, determinata soprattutto
dallenorme produzione di sequenze di acidi nucleici e di proteine, che il risultato
degli studi relativi alle due discipline omiche, genomica e proteomica.
Lobiettivo dellera post-genomica quello di comprendere i meccanismi molecolari
che determinano lattivit biologica di tutte le proteine codificate da ciascun genoma
sequenziato.
Per poter raggiungere questo obiettivo, gli strumenti computazionali e bioinformatici
sono di grande aiuto, anche se essi devono essere considerati naturalmente
complementari e non alternativi alle normali tecniche sperimentali. Questi strumenti
sono, infatti, utili per comprendere come le proteine si ripiegano nelle strutture native
(protein folding), per predire la struttura tridimensionale di una proteina, in modo
veloce ed accurato, dalla sola conoscenza della sua sequenza amminoacidica, e per
formulare ipotesi sullattivit biologica della proteina in esame. Tuttavia, ovviamente
da ricordare che qualunque applicazione pratica di quanto ipotizzato pu essere
realizzata solo mediante ulteriori studi di tipo sperimentale.
-
- 9 -
1.1 Protein folding
Le proteine svolgono nelle cellule degli organismi viventi un gran numero di funzioni
che vanno dal semplice trasporto e immagazzinamento di piccole molecole e ioni a ruoli
pi complessi quali i processi enzimatici che sono necessari per la vita. Queste
macromolecole sono costituite da venti tipi diversi di amminoacidi, legati in
successione mediante il legame peptidico. Le diverse possibili sequenze di amminoacidi
determinano strutture diverse dal punto di vista sia della struttura covalente sia della
conformazione assunta nello spazio dalla proteina. Ed proprio la struttura 3D di una
proteina che determina la sua funzione.
In realt, non realistico ipotizzare una semplice assegnazione sequenza->struttura, dal
momento che sono conosciute molte proteine che, pur avendo un valore di omologia di
sequenza molto basso, hanno strutture tridimensionali molto simili. Il numero di
conformazioni strutturali (struttura tridimensionale di una proteina o fold), osservate
finora, minore di 700, anche perch queste derivano dalla combinazione di un piccolo
numero di elementi semplici quali i due elementi principali di struttura secondaria
presenti nelle proteine, eliche e foglietti .
Il meccanismo di avvolgimento della catena polipeptidica (folding), mediante il quale
una proteina assume in condizioni fisiologiche la sua struttura tridimensionale
funzionalmente attiva, rappresenta il passaggio conclusivo del trasferimento
dellinformazione genetica dal DNA al suo prodotto finale (proteina attiva). La
comprensione dei meccanismi, attraverso cui una catena polipeptidica giunge alla sua
struttura tridimensionale attiva, affascina gli studiosi da vari decenni. Nel 1931, quando
-
- 10 -
non era noto niente riguardo la sequenza e la struttura tridimensionale delle proteine,
Wu [Wu, 1931] ha analizzato il processo di denaturazione delle proteine ed il loro
ritorno allo stato nativo. Negli anni 30 sono stati pubblicati molti articoli, che mostrano
che il processo di unfolding delle proteine reversibile, sottolineando in questo modo
che il protein folding un processo spontaneo. Negli anni 50 le nuove conoscenze
termodinamiche hanno sottolineato limportanza delle interazioni non covalenti
riguardo la stabilit delle proteine; in particolare, Kauzmann ha suggerito che leffetto
idrofobico la forza guida, che dirige il processo del folding [Kauzmann, 1959]. La
determinazione della prima struttura 3D di una proteina (cio della mioglobina) nel
1958 ad opera di John Kendrew mediante il metodo di diffrazione ai Raggi X ha fornito
una nuova base per lanalisi della struttura delle proteine e per lo studio del processo di
folding. Anfinsen ha dimostrato che alcune proteine in vitro possono essere sottoposte,
introducendo agenti denaturanti quali la guanidina e lurea, ad un processo reversibile di
denaturazione, durante il quale perdono la loro struttura tridimensionale [Anfinsen et al.,
1961; Anfinsen et al., 1962; Anfinsen, 1973]. Rimuovendo questi agenti denaturanti si
riottiene la struttura tridimensionale attiva caratterizzata da una struttura tridimensionale
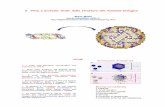
compatta [Figura 1.1]. Questa osservazione ha consentito ad Anfinsen di affermare che
linformazione necessaria per ottenere la conformazione nativa (N) di una proteina in
una data condizione fisiologica contenuta nella sua sequenza amminoacidica. Ci, da
un punto di vista termodinamico, si traduce nella possibilit di affermare che lo stato N
nelle condizioni fisiologiche costituisce un minimo dellEnergia Libera di Gibbs.
-
- 11 -
Figura 1.1 Schema relativo allesperimento di Anfinsen.
Le osservazioni di Anfinsen sono state ulteriormente ampliate e discusse da Levinthal,
che si pose il problema del tempo necessario affinch un sistema potesse raggiungere il
suo stato di equilibrio [Levinthal, 1968]. Infatti, supponendo che il numero di
conformazioni accessibili al singolo amminoacido sia uguale a due (elica e foglietto
beta), per una catena polipeptidica di 100 amminoacidi il numero totale di
conformazioni possibili 2100
, che corrisponde a pi di 1030
. Se noi assumiamo che il
tempo di interconversione da una conformazione alla sua alternativa pari a 10-11
Schema dellesperimento di Anfinsen
-
- 12 -
secondi, il tempo necessario per una ricerca casuale di tutte le conformazioni di 1011
anni. Dato che i tempi di folding spaziano da qualche secondo ad alcuni minuti
evidente che levoluzione ha trovato una soluzione efficace a questo procedimento. La
soluzione di Levinthal a questo paradosso, ampliata e portata avanti anche da altri
autori, stata che il meccanismo di folding sottoposto ad un controllo di tipo cinetico,
ovvero che esistono dei veri e propri percorsi definiti, che conducono dalla struttura
casuale e lineare (U) alla struttura nativa e funzionale (N). Da questo momento molti
autori cominciarono a valutare se gli stati parzialmente strutturati, evidenziati
sperimentalmente, rappresentassero degli intermedi produttivi (on pathway), cio delle
tappe fondamentali nel percorso del folding, o degli intermedi improduttivi (off
pathway). Agli inizi degli anni 80 stato evidenziato che gli intermedi on pathway
hanno una struttura secondaria in grado di formare un nucleo compatto, ma pi espanso
della proteina nativa a causa dellassenza di specifiche interazioni terziarie (molten
globule) [Figura 1.2].
Gli studi sono poi continuati negli anni fino ad arrivare ad una nuova visione del
protein folding (folding funnel). In questa nuova visione, il concetto di percorso di
folding, costituito da eventi sequenziali, stato sostituito dal concetto di imbuto (funnel)
di eventi paralleli rappresentato da diagrammi energetici [Figura 1.3]. In questi
diagrammi lasse verticale rappresenta lenergia libera interna di ogni specifica
conformazione mentre gli assi orizzontali rappresentano le coordinate conformazionali
necessarie per specificare ogni singola conformazione (ad esempio gli angoli diedri). La
forma ad imbuto descrive la progressiva riduzione dello spazio conformazionale
-
- 13 -
accessibile, a partire dai molti gradi di libert disponibili per le catene denaturate, fino
ad arrivare alla proteina nello stato nativo che caratterizzata, in prima
approssimazione, da un unico sottostato conformazionale. Nella Figura 1.3a
rappresentato limbuto che descrive il panorama energetico (energy landscape) pi
semplice cio quello di una reazione a due stati, in cui non c nessuno stato intermedio.
Se si prendono in considerazione la formazione di intermedi, di trappole cinetiche e la
presenza di barriere energetiche, il panorama diviene pi vario [Figura 1.3b]. Il
modello ad imbuto supera il paradosso di Levinthal, in quanto, pur ammettendo un gran
numero di diversi cammini, alcuni dei quali possono dare origine ad intermedi
inizialmente improduttivi, non consente un campionamento completamente casuale ma
spinge il sistema verso il suo minimo di energia favorendo alcuni riarrangiamenti che
portano verso lo stato nativo e rendendo estremamente improbabili i percorsi che
risalgono limbuto energetico.
La nuova visione sul folding ha fornito uno spunto per altri studi, che si propongono di
comprendere la relazione esistente tra le caratteristiche intrinseche di una proteina e la
sua velocit di folding allo scopo di identificare parametri utili a predire luna in base
alle altre. Qualche anno fa, da unanalisi di proteine appartenenti a famiglie non
omologhe, per le quali era disponibile una grande quantit di dati strutturali e cinetici,
emersa una correlazione tra la distanza media nella sequenza fra i residui che
interagiscono nello stato nativo e la velocit di folding.
-
- 14 -
Figura 1.2 Rappresentazione schematica del molten globule a confronto con la struttura
dello stato nativo.
Figura 1.3 Rappresentazione schematica attraverso diagrammi energetici a tre
dimensioni (folding funnels) del processo di folding a due stati (a) e multistato (b).
-
- 15 -
Questo parametro stato definito contact order. A questi lavori sperimentali si sono
affiancati una serie di lavori teorici di Dinamica Molecolare, con il fine di simulare le
propriet strutturali fondamentali degli intermedi, che si formano durante il folding e
che sono risultate essere correlate alle propriet topologiche dello stato nativo [Clementi
et al., 2000]. Inoltre, stato anche determinato il ruolo giocato dalla posizione
geometrica degli amminoacidi nel processo del folding in alcune proteine.
Il protein folding un argomento di grande interesse soprattutto dopo che stato
completato il sequenziamento del genoma di vari organismi (732 Batteri, 44 Archea e
786 Eucarioti). Infatti, il problema maggiore dellera post-genomica quello di
individuare i singoli geni e le proteine da essi codificate e, soprattutto, di avere
informazioni sulla loro struttura tridimensionale, perch essa, pi della sequenza, che
definisce la loro funzione biologica. La comprensione approfondita dei meccanismi
molecolari, che sono alla base del folding delle proteine, rappresenterebbe un traguardo
per tutta la comunit scientifica, in quanto essa fornirebbe la possibilit di curare molte
patologie associate ai processi di misfolding e di aggregazione di proteine, quali le
encefalopatie spongiformi, che si possono originare in seguito a disordini genetici
sporadici e/o infettivi, che coinvolgono il cambiamento conformazionale della proteina.
Ad esempio, quella del prione una proteina di cui non si conosce ancora la funzione.
Essa presente nella cellula nella sua conformazione normale PrPC, che costituita
principalmente da -eliche, e si converte nella forma patologica PrPSC in cui parte delle
-eliche si trasforma in foglietti . Il meccanismo dellazione infettiva del prione
sembra essere quello di agire come stampo per la conversione di altre proteine sane
-
- 16 -
nella forma patologica. In pratica, le proteine patologiche sono in grado di indurre in
proteine sane una cambiamento conformazionale che produce un riarrangiamento
conformazionale (misfolding). Come il prione si converte dalla forma normale a quella
patologica e cosa favorisce questo processo ancora sconosciuto.
La pubblicazione della sequenza completa del genoma di vari organismi, ha portato alla
scoperta delle sequenze di molte proteine, di cui non sono note n le strutture n le
funzioni. Al momento sono riportate nella banca dati GenBank 32 549 400 sequenze
nucleotidiche, in UniProt/Swiss-Prot 1 585 764 sequenze proteiche e in PDB 27 761
strutture proteiche. Come si vede da questi dati, il numero di proteine, di cui stata
determinata sperimentalmente la struttura 3D, molto pi basso di quello delle
sequenze note; ci dovuto certamente sia al fatto che i metodi sperimentali non sono
sempre applicabili sia al fatto che il loro utilizzo richiederebbe troppo tempo.
1.2 Metodi di predizione della struttura delle proteine
La struttura di una proteina pu essere ottenuta sperimentalmente mediante
Spettroscopia di Risonanza Magnetica (NMR) e mediante diffrazione ai Raggi X (RX).
LNMR permette di esaminare una proteina in soluzione e di generare anche un quadro
della sua dinamica ma applicabile solo a proteine che non superano i 250-300 residui.
La diffrazione ai Raggi X offre dati molto precisi ma le strutture costrette in cristalli non
sempre rappresentano immagini fedeli di proteine nella loro conformazione attiva.
Questi metodi sperimentali spesso non possono essere utilizzati dal momento che non
-
- 17 -
tutte le proteine sono cristallizzabili o solubili nelle quantit sufficienti per misure
NMR.
In alternativa ai metodi sperimentali, si sono sviluppati dei metodi computazionali
aventi lo scopo di predire la struttura tridimensionale di una proteina, in modo veloce ed
accurato, dalla sola conoscenza della sua sequenza amminoacidica e di comprendere
come le proteine si ripiegano nelle strutture native. Attualmente ci sono vari metodi di
predizione di struttura secondaria delle proteine e di struttura terziaria, tra i quali si
possono distinguere tre categorie: modellamento per omologia, riconoscimento di fold e
metodi ab-initio.
1.3 Metodi di predizione di struttura secondaria
Negli ultimi anni le tecniche di predizione di struttura secondaria sono arrivate ad
offrire un alto grado di affidabilit. In generale, si possono distinguere due tipi di
metodi: metodi statistici e metodi connessionistici. Essi si propongono di assegnare
gli elementi di struttura secondaria a sequenze proteiche partendo dalla conoscenza
della struttura di proteine, utilizzate come campioni esemplari, delle quali siano note sia
la sequenza sia la conformazione tridimensionale.
Una delle tecniche di predizione su base statistica pi usate quella elaborata da Chou
e Fasman, che va a valutare la propensit di ciascun amminoacido a trovarsi in una
particolare struttura secondaria (elica, -strand e coil) [Chou & Fasman, 1974]. Questo
metodo fornisce una tabella nella quale ciascun amminoacido viene classificato con un
-
- 18 -
coefficiente, che riflette la frequenza con la quale esso forma, interrompe o
indifferente alla formazione di ciascun tipo di struttura secondaria.
Un altro criterio statistico di predizione quello di Garnier, Osguthorpe e Robson.
Lidea basilare su cui tale metodo fondato che lo stato conformazionale di un dato
amminoacido determinato non solo dalla sua stessa natura ma anche da quella degli
altri amminoacidi ad esso adiacenti. Quindi un dato amminoacido R nella posizione j+m
esercita uninfluenza sullo stato confomazionale del residuo j-mo misurata come I(Sj,
Rj+m), dove Sj lo stato conformazionale del residuo j . Ci significa che se il residuo
nella posizione j-1 si trova in un dato elemento strutturale, probabile che anche il
residuo j faccia parte di esso. In pratica si considera significativa linfluenza di 8 residui
amminoacidici a sinistra ed a destra di quello considerato.
La probabilit che il residuo j adotti la conformazione S viene calcolata come
L(Sj) = I(Sj,Rj+m) dove m = -8, ., +8
e per il residuo j viene predetto lo stato conformazionale al quale corrisponde il
maggiore valore di probabilit.
Questi metodi statistici non raggiungono unaccuratezza maggiore del 65%.
Linformazione evolutiva presente nellallineamento multiplo di un insieme di proteine
omologhe pu consentire un incremento significativo dellaccuratezza della predizione
delle strutture secondarie. Infatti, il metodo connessionistico pi utilizzato PHDsec
(a Profile fed neural network system from Heidelberg for secondary structure
prediction). Esso utilizza linformazione evolutiva derivante dallallineamento multiplo
di un insieme di sequenze di proteine omologhe. In particolare, sottomessa una singola
-
- 19 -
sequenza, il programma cerca in modo automatico nella banche dati proteine omologhe
a quella di partenza, ne esegue lallineamento multiplo e procede allapplicazione
dellalgoritmo. Questo algoritmo di predizione utilizza una rete neurale a pi strati
tarata da una fase di apprendimento effettuata su una serie di proteine a struttura
tridimensionale nota. Questo metodo connessionistico raggiunge una accuratezza media
del 72%.
1.4 Modellamento per omologia
Il modellamento per omologia il metodo pi affidabile per ottenere una predizione
della struttura tridimensionale di una proteina ed applicabile quando la percentuale di
identit di sequenza tra la proteina da modellare e quella di riferimento compresa tra il
20-40%.
Infatti, due proteine omologhe, cio derivanti da uno stesso progenitore per un processo
evoluzionistico, hanno subito durante levoluzione solo mutazioni che non hanno
distrutto n la loro funzione biologica n la loro struttura 3D. Da ci consegue che,
quando due proteine hanno sequenze simili e la stessa funzione, avranno sicuramente
anche strutture 3D simili.
Si pu, inoltre, sottolineare che esiste una relazione non biunivoca tra la similarit di
due sequenze proteiche (numero di amminoacidi identici o simili) e la somiglianza tra le
rispettive strutture tridimensionali; infatti, sono anche note proteine che, pur non avendo
sequenze simili, hanno strutture simili.
-
- 20 -
Dal momento che il modello per omologia si basa sullosservazione empirica che la
similarit fra le sequenze di due proteine implica una similarit nella loro struttura, le
coordinate della catena principale degli amminoacidi della proteina presa come
riferimento (template) possono essere usate come unapprossimazione delle coordinate
delle regioni corrispondenti (secondo lallineamento) della proteina da modellare
(target).
Il modellamento per omologia si articola in vari stadi:
identificazione della proteina di struttura nota che si user come riferimento
(template);
identificazione delle regioni che ci si aspetta siano strutturalmente conservate tra
il template e la proteina target;
allineamento delle sequenze amminoacidiche di queste regioni;
costruzione del modello delle regioni conservate usando come coordinate quelle
della catena principale della proteina template secondo la corrispondenza dettata
dallallineamento delle sequenze;
costruzione del modello delle regioni strutturalmente variabili: regioni in cui ci
sono delezioni ed inserzioni;
modellamento delle catene laterali del modello;
rifinitura del modello.
ormai ben noto, che la similarit della catena principale nel core (nucleo
strutturalmente conservato tra proteine omologhe) di due proteine aumenta
allaumentare della somiglianza tra le loro sequenze. Qualche anno fa, Cyrus Chothia e
-
- 21 -
Arthur Lesk (1986) hanno analizzato una trentina di coppie di proteine omologhe di
struttura nota e sono andati a valutare la relazione tra lRMSD (deviazione quadratica
media) del core delle due strutture sovrapposte e la percentuale di identit tra le loro
sequenze. Questa analisi stata fatta nel 1986 ma i risultati ottenuti sono stati
successivamente confermati da vari autori che hanno utilizzato un numero maggiore di
strutture proteiche [Hilbert et al., 1993]. stato visto che lRMSD degli atomi della
catena principale del core tra due proteine con identit di sequenza maggiore del 50%
minore di 1.0 Angstrom ed il core comprende il 90% delle strutture. Inoltre per
coppie di proteine con identit di sequenza minore del 20%, la regione del core pu
comprendere non pi del 50% delle strutture con una RMSD della catena principale in
questa regione maggiore di 1.8 Angstrom; fuori dal core le deviazioni possono essere
significative. Infine, coppie di proteine con identit in sequenza tra il 20% ed il 50 %
hanno un grado di similarit intermedio [Figura 1.4].
Da ci consegue che per costruire un modello il migliore template quello che ha la
maggiore identit di sequenza con la proteina target. Quando esistono pi di una
proteina di struttura nota con la stessa percentuale di identit di sequenza con la proteina
target, consigliabile scegliere la migliore, in base alla completezza ed alla risoluzione.
Deciso quale o quali proteine possono essere utilizzate come riferimento, necessario
allineare le sequenze in modo da rendere massima la loro identit di sequenza (cio il
numero di amminoacidi identici in posizioni corrispondenti) o la loro similarit
(assegnando un punteggio che descriva in qualche modo la similarit di ciascuna
possibile coppia di amminoacidi).
-
- 22 -
Figura 1.4 Relazione tra la percentuale di identit di sequenza di coppie di proteine ed i
valori di RMSD relativi ai Carboni alfa (C), ottenuti dopo aver sovrapposto le loro strutture tridimensionali [Hilbert et al., 1993].
Gli algoritmi di allineamento di sequenze permettono di misurare ed ottimizzare
lidentit e la similarit fra sequenze in modo sufficientemente accurato. Questo per
non corrisponde alla migliore sovrapposizione strutturale fra proteine, che quello di
cui abbiamo bisogno per costruire un modello accurato. Pertanto dopo aver allineato le
sequenze in modo automatico, necessario controllare manualmente lallineamento
ottenuto sfruttando informazioni varie quali la predizione di struttura secondaria, le
sequenze di altre proteine della stessa famiglia della proteina target, la struttura
tridimensionale della proteina template ed informazioni sperimentali su una o tutte le
proteine. In particolare, le inserzioni e le delezioni, che sono le regioni pi difficili da
modellare, determinano variazioni strutturali locali. Pertanto tenendo presente la
struttura tridimensionale della proteina di riferimento, bisogna controllare che le
delezioni e le inserzioni non capitino in elementi di struttura secondaria, e aggiustare
manualmente lallineamento. Spesso utile allineare tutte le sequenze appartenenti alla
0
0,5
1
1,5
2
2,5
3
0 20 40 60 80 100
Identit [%]
RM
SD
(C
) [
]
-
- 23 -
famiglia della proteina di riferimento; ci permette di verificare quali regioni sono pi
conservate strutturalmente nella famiglia, anche perch queste saranno probabilmente
conservate anche nella proteina target. Infine, le informazioni sperimentali sono
importanti perch se la proteina target e quella di riferimento hanno la stessa funzione,
gli amminoacidi del sito attivo devono essere allineati. Ottenuto un buon allineamento
possibile modellare le regioni strutturalmente conservate (SCR) della proteina target ma
rimane il problema di come modellare i loop e le catene laterali.
I loop, definiti come regioni strutturalmente variabili, non possono essere costruiti per
omologia. Essi sono, di solito, regioni che connettono elementi di struttura secondaria,
sono esposti sulla superficie e meno regolari di -eliche e foglietti . Al momento per
modellare i loop vengono utilizzati o metodi di ricerca in banche dati o metodi ab-
initio.
Il metodo di ricerca in banca dati si basa sullosservazione che regioni di
conformazione simile si trovano in proteine sia omologhe sia non omologhe e, quindi,
costruite per omologia le strutture delle regioni che fiancheggiano il loop, il numero di
modi per unire tali strutture con un loop di lunghezza nota non pu essere infinito e si
possono ricercare nelle banche dati frammenti di proteine che si adattano a queste
regioni, che sono definite stem. In pratica, si va a ricercare nella banca dati di strutture
note delle regioni, che siano simili agli stem e che siano separate da un numero di
residui uguale a quello del loop, che si deve modellare. I metodi ab-initio per la
predizione dei loop si basano su simulazioni energetiche; quindi si generano le
coordinate tridimensionali di tutti i loop (o quasi) che potrebbero congiungere gli stem
-
- 24 -
andando a valutare lenergia dellintera proteina nei vari casi e scegliendo il loop, per il
quale lenergia totale assume il valore minimo.
Per quanto riguarda le catene laterali di ciascun amminoacido si andati a valutare la
frequenza, con cui ciascun amminoacido viene osservato in una certa conformazione
nelle proteine di struttura nota. Gli angoli corrispondenti a queste conformazioni sono
raccolti in librerie di rotameri, che possono essere utilizzate per assegnare la
conformazione agli amminoacidi della proteina target. Vari studi hanno confermato che
se esiste una relazione evoluzionistica fra la proteina di riferimento e quella target, ci si
pu aspettare che anche le catene laterali delle due proteine tendono ad assumere
conformazioni simili e quindi possono essere modellate le une sulle altre. Pertanto,
molti metodi copiano gli angoli della catena dellamminoacido del template fin dove la
lunghezza relativa delle catene laterali lo permette ed usano le librerie di rotameri per la
parte restante. Spesso si utilizzano anche calcoli energetici; infatti, assegnato a ciascun
amminoacido il suo rotamero pi frequente, lenergia totale della molecola viene
sottoposta ad un processo di minimizzazione per rifinire gli angoli. Laccuratezza di
questi metodi diminuisce allaumentare della deviazione della catena principale del
modello dalla struttura di riferimento; da ci si deduce che, se vengono migliorati i
metodi per costruire la catena principale, si riuscir ad ottenere anche una migliore
predizione delle catene laterali.
1.5 I metodi di riconoscimento di fold
-
- 25 -
noto che ci sono proteine che esibiscono lo stesso fold anche in assenza di una
rilevante similarit di sequenza e che il numero di fold, rappresentati in natura,
relativamente limitato (meno di 700)Thorntonet al., 1999. Proteine con lo stesso fold
ma con nessun similarit significativa di sequenza possono essersi evolute da un
ancestore comune ma essersi diversificate tanto che la loro origine comune non pi
facilmente deducibile dal confronto tra le loro sequenze, oppure anche possibile che la
similitudine sia dovuta al fatto che quella architettura favorita per ragioni chimico-
fisiche.
In presenza di una proteina, che non ha similarit di sequenza con nessuna delle
proteine note, il modellamento per omologia non pu essere utilizzato e, quindi, si va a
ricercare se la sua sequenza compatibile con uno dei fold gi noti, valutando la
probabilit con cui la sequenza target possa assumere una delle strutture presenti nella
banca dati, indipendentemente dalla loro similarit di sequenza (target/template).
Questo metodo viene chiamato riconoscimento di fold. I due approcci pi usati sono
quelli basati su profili e quelli cosiddetti di threading.
I metodi basati su profili si basano sulla possibilit di dedurre dallanalisi di proteine
di struttura nota alcune propriet caratteristiche per ciascun amminoacido, quali la
frequenza relativa con cui ciascun amminoacido osservato in uno dei tipi di struttura
secondaria (preferibilmente , preferibilmente e nessuna preferenza), la frequenza con
cui osservato sulla superficie di una proteina (alta, bassa ed intermedia) e la frequenza
con cui osservato in un ambiente idrofobico (alta, bassa). In questo modo possibile
-
- 26 -
associare a ciascun amminoacido una lettera, che rappresenta le modalit con cui esso
pi frequentemente osservato nelle strutture note [Tabella 1.1].
Tabella 1.1 Possibile codifica delle propensit degli amminoacidi
Pi spesso in ..
Frequenza
di presenza
in superficie
Altra
Bassa Pi frequentemente in
ambiente idrofobico (a)
Pi frequentemente in
ambiente idrofilico (d)
Pi frequentemente in
ambiente idrofobico (b)
Pi frequentemente in
ambiente idrofilico (e)
Pi frequentemente in
ambiente idrofobico (c)
Pi frequentemente in
ambiente idrofilico (f)
Alta Pi frequentemente in
ambiente idrofobico (g)
Pi frequentemente in
ambiente idrofilico (j)
Pi frequentemente in
ambiente idrofobico (h)
Pi frequentemente in
ambiente idrofilico (k)
Pi frequentemente in
ambiente idrofobico (i)
Pi frequentemente in
ambiente idrofilico (l)
Intermedia Pi frequentemente in
ambiente idrofobico (m)
Pi frequentemente in
ambiente idrofilico (p)
Pi frequentemente in
ambiente idrofobico (n)
Pi frequentemente in
ambiente idrofilico (q)
Pi frequentemente in
ambiente idrofobico (o)
Pi frequentemente in
ambiente idrofilico (r)
Ripetendo questo tipo di analisi per tutte le proteine di struttura nota, la banca dati di
struttura tridimensionale diventa una banca dati lineare come quella relativa alle
sequenze. Mediante i metodi classici di ricerca in banca dati, la sequenza delle
propensit della proteina target pu essere confrontata con la banca dati che rappresenta
le caratteristiche strutturali delle proteine note. In questo modo, le proteine, che
mostrano similarit significativamente pi alta con la proteina target, sono quelle che
possono essere utilizzate come riferimento.
-
- 27 -
Nei metodi di threading si costruiscono tanti possibili modelli della proteina usando
come riferimento (template) le proteine di struttura nota ed esplorando un gran numero
di possibili allineamenti che includono inserzioni e delezioni. Tra questi modelli
vengono scelti quelli che risultano migliori andando a fare per ciascuno di essi una
valutazione energetica a livello degli amminoacidi e non dei singoli atomi.
1.6 Folding ab-initio
I metodi descritti finora (modellamento per omologia e metodo basato sul
riconoscimento del fold) si basano sempre sullosservazione di proteine note ma non ci
permettono di capire come fa una proteina a raggiungere la sua struttura nativa in natura
dal momento che le proteine non consultano banche dati. Una proteina si struttura
nella sua conformazione nativa perch questa energeticamente pi favorevole di
qualunque altra possibile conformazione; pertanto, se si riuscissero a generare tutte le
possibili conformazioni di una proteina ed a valutare correttamente la loro energia,
basterebbe andare a scegliere la conformazione a energia pi bassa. Questo
procedimento, per, non applicabile poich richiederebbe troppo tempo.
I metodi ab-initio si basano sulla ricerca dei minimi di energia conformazionale e
necessitano di due requisiti fondamentali: la determinazione della funzione energia
che permetta di discriminare la conformazione nativa dalle altre ed un criterio affidabile
ed efficiente di ricerca dei minimi energetici nello spazio delle conformazioni. Per
valutare tutti i contributi energetici coinvolti nel calcolo dellenergia conformazionale si
deve tener conto sia di fattori intramolecolari (legami chimici, interazioni di van der
-
- 28 -
Waals, legami idrogeno, interazioni coulombiane, entropia conformazionale) sia
dellinterazione con il solvente (polarizzazione del mezzo, formazione di cavit,
interazioni soluto-solvente, variazioni di struttura del solvente).
Un modo per cercare la conformazione a energia minima di minimizzare la funzione
rispetto alla posizione degli atomi. Praticamente, partendo da una certa conformazione
si variano le posizioni degli atomi e si calcola lenergia della nuova conformazione. Se
questa minore della precedente, si ripete il procedimento effettuando unaltra piccola
variazione, altrimenti si ritorna indietro e si prova una variazione diversa. Mediante
questo procedimento di minimizzazione possibile trovare il minimo locale ma non
quello globale, cio quello pi vicino alla conformazione di partenza ma non il pi
basso possibile nel caso in cui ci sono delle barriere di potenziale tra questultimo e la
conformazione iniziale. Questo problema pu essere superato o esplorando in maniera
casuale lo spazio conformazionale senza preoccuparsi del fatto che esiste un modo
fisicamente permesso per andare da una conformazione allaltra (metodi stocastici) o
fornendo agli atomi unenergia cinetica che permetta a questi di superare la barriera di
potenziale (dinamica molecolare).
Molti studi sperimentali e teorici hanno dimostrato che il processo di folding
influenzato dalle propriet topologiche dello stato nativo. Baker et al. [Plaxco et al.,
1998] hanno indicato che esiste una correlazione tra le cinetiche del folding e la
complessit topologica dello stato nativo. Koga e Takada [Koga e Takata, 2001] hanno
studiato le relazioni tra la topologia di una proteina ed i folding pathways. Questi autori
sono riusciti a descrivere i folding pathways di piccole proteine a singolo dominio,
-
- 29 -
considerando solo i C della catena polipeptidica ed usando una funzione di energia
libera che tiene conto della connettivit della catena, delle interazioni e dellentropia.
Un interessante approccio topologico al problema del protein folding stato proposto
recentemente dal gruppo di Banavar e Maritan [Banavar et al., 2002, 2003a, 2003b].
Secondo questo approccio una proteina modellata come un tubo di spessore non nullo.
Mediante la procedura Metropolis Monte Carlo questi autori hanno simulato delle
strutture di tipo elica e strand simili a quelle presenti nelle proteine. I risultati ottenuti
sono incoraggianti e possono essere utilizzati in studi futuri [].
1.7 CASP
Una valutazione dellaffidabilit di questi metodi (modellamento per omologia, metodo
di riconoscimento di fold, metodi ab-initio) viene fatta ogni due anni dalla comunit
scientifica internazionale che ha istituito nel 1994 un esperimento chiamato CASP
(Critical Assessment of Methods for Protein Structure Prediction). Questo esperimento
valuta lefficacia di un metodo, confrontando la predizione con un risultato
sperimentale. In pratica, ogni due anni viene chiesto a cristallografi ed a spettroscopisti
NMR, che stanno per risolvere la struttura di una proteina, di rendere disponibile la sua
sequenza. Queste sequenze (target) vengono assegnate ad una serie di predittori che
devono depositare i loro modelli prima che la struttura sia resa pubblica. Un insieme di
valutatori (assessors) confronta i modelli e le strutture, appena queste ultime sono rese
disponibili, e cerca di valutare le predizioni e di trarre conclusioni generali. I risultati
-
- 30 -
vengono poi discussi in un convegno dove i valutatori ed i predittori si incontrano per
discutere dei risultati.
Dai risultati del CASP5 [Proteins 2003, 53 Suppl. 6, 333-585] si pu avere una
valutazione dellaccuratezza raggiunta dai tre metodi. Il modellamento comparativo
risultato ancora il metodo predittivo pi affidabile. Ottimi risultati sono stati ottenuti
soprattutto per le zone strutturalmente conservate (definite come core) della proteina
target. I limiti maggiori restano sempre quelli del modellamento delle catene laterali e
dei loop; infatti, molti metodi sono stati sviluppati ma i risultati non sono ancora
positivi. Sono stati ottenuti buoni risultati, nel caso di bassa percentuale di identit di
sequenza tra la proteina target e quella/e template, migliorando lallineamento mediante
i modelli di Markov ed i metodi basati sui profili.
Gli esperimenti del CASP prevedono anche una sezione di valutazione di server
automatici (CAFASP). Molti sono stati i server automatici di modellamento per
omologia, che hanno ottenuto risultati migliori della media dei predittori ma anche da
sottolineare che per lo stesso target si sono registrate sia predizioni di ottima qualit sia
predizioni completamente improbabili. Lo stesso si pu dire per il metodo del
riconoscimento di fold. Alcune volte i modelli ottenuti per riconoscimento di fold sono
risultati pi simili alla struttura sperimentale di qualunque delle strutture presenti nella
banca dati.
Inoltre, i predittori, che hanno ottenuto i migliori risultati, hanno combinato i loro
metodi ed hanno organizzato un paio di workshop per poter discutere dei risultati
ottenuti. Le proteine per cui si riusciti ad avere risultati migliori, sono state quelle su
-
- 31 -
cui uno dei partecipanti lavorava sperimentalmente. Ci ha fatto dedurre che un
qualsiasi metodo funziona meglio se abbinato ad una approfondita conoscenza delle
caratteristiche biologiche delle proteine.
Per quanto riguarda i metodi ab-initio, dai risultati del CASP5 si potuto dedurre che
nessuno dei metodi (minimizzazione, dinamica molecolare, Monte Carlo, algoritmi
genetici) in grado di trovare la conformazione a minima energia di una proteina. Ma la
combinazione di questi metodi pu dare buoni risultati per predire strutture di
frammenti proteici. Il metodo di maggior successo nella categoria dei metodi ab-initio
sia nel CASP4 sia nel CASP5 stato il metodo ROSETTA. In questo metodo, la
sequenza di una proteina target viene divisa in frammenti contigui di 3 e 9
amminoacidi. Tutti i frammenti di proteine di struttura nota che hanno sequenze uguali
o simili a queste regioni vengono combinati, utilizzando il Metodo di Monte Carlo, al
fine di predire la possibile conformazione della proteina target.
1.8 La Predizione delle interazioni proteina-proteina Quando nota la struttura di due proteine e si sa che esse interagiscono, predire la loro
orientazione relativa nel complesso rappresenta un problema non facile da risolvere. La
simulazione fatta in silico della formazione del complesso molecolare a partire dalle
strutture tridimensionali delle proteine, che lo compongono, viene definita con il
termine docking.
Il problema maggiore relativo alla predizione delle interazioni proteina-proteina che la
struttura delle proteine in un complesso abbastanza diversa da quella assunta dalle
-
- 32 -
stesse proteine nella loro forma libera soprattutto nelle regioni dellinterazione. Ci
certamente dovuto al fatto che le catene laterali dei residui delle proteine sono
relativamente mobili e talora seguono il formarsi del complesso con movimenti che
determinano una migliore complementarit tra i residui delle proteine interagenti.
Questi movimenti coinvolgono non solo le catene laterali dei residui ma talvolta
comportano anche spostamenti di interi segmenti di strutture secondarie.
Diversi metodi di docking sono stati sviluppati (DOCK, AUTODOCK, FlexX,
ESCHER) sia per la ricostruzione di complessi proteina-proteina sia per lanalisi di
complessi tra proteine e ligandi. Il docking molto utilizzato anche per la ricerca di
nuovi inibitori di una proteina data mediante lutilizzo di banche dati di possibili
ligandi. I metodi, finora sviluppati, si basano o su criteri geometrici o energetici. I
metodi energetici sfruttano il fatto che le proteine formano complessi poich questi
sono energeticamente favoriti mentre quelli geometrici si basano sulla considerazione
che le superfici di interazione delle due proteine, che formano il complesso, devono
essere complementari.
Una valutazione delle procedure di docking proteina-proteina, finora sviluppate, viene
fatta periodicamente mediante un esperimento, analogo a quello del CASP, denominato
CAPRI (Critical Assessment of PRedicted Interactions). Proprio come per il CASP, le
predizioni vengono fatte e confrontate con le strutture dei complessi, ottenute mediante
diffrazione ai Raggi X, prima che queste vengano rese pubbliche. In una delle ultime
edizioni (CAPRI round 3) emerso che molti metodi di docking trattano i componenti
molecolari come corpi rigidi, mentre altri fanno ci solo nei primi passaggi della
-
- 33 -
simulazione, in modo da eliminare le soluzioni pi improbabili, e poi modellano le
catene laterali e/o il backbone. Il maggiore limite di questi metodi nel fatto che essi,
quando tentano di predire strutture di complessi, raramente sono in grado di fornire una
sola soluzione. Infatti, la maggior parte delle volte forniscono una lista di possibili modi
di interazioni e scegliere la migliore tra queste non facile. Recentemente, analizzando
strutture di complessi note, si cercato di studiare quali possono essere i parametri
legati allinterfaccia proteina-proteina. Ma eccetto lampiezza dellinterfaccia, che, in
generale anche se non sempre, tende ad essere pi larga nei complessi biologicamente
attivi, altri parametri, come il numero di legami ad idrogeno per unit di superficie e le
propensit di contatto tra residui, non sono risultati discriminatori.
Per la predizione dellinterazione proteina-proteina sono stati ottenuti buoni risultati
combinando i metodi di docking con i due approcci classici, modellamento per
omologia e threading, i quali rappresentano una strategia integrata, capace di predire i
siti di interazione, i contatti tra i residui e, nei casi pi fortunati, anche un modello
dettagliato del complesso. Infatti, questi due metodi usano la struttura di un complesso
noto come riferimento (template) per costruire il modello del complesso target. Per il
limite di questo approccio certamente legato alla percentuale di somiglianza, che c
tra le proteine target e quelle template. Russell ed i suoi collaboratori hanno
recentemente dimostrato che proteine con una percentuale di omologia pari al 30-40%
interagiscono allo stesso modo mentre il modo di interagire raramente conservato per
proteine con percentuale di identit di sequenza pi bassa.