miv divisione batterica
-
Upload
antonio-nenna -
Category
Documents
-
view
1.027 -
download
5
description
Transcript of miv divisione batterica

Microbiologia DIVISIONE BATTERICA Antonio Nenna
DIVISIONE BATTERICA
Il nucleoide non è una struttura omogenea, ma simile a una spugna marina; la trascrizione quindi non può avvenire all’interno del nucleoide, ma sui prolungamenti (escrescenze) che si estendono in direzione radiale verso il citoplasma. INIZIO DELLA REPLICAZIONE NEI BATTERI La corretta trasmissione del materiale genetico richiede la precisa coordinazione e regolazione della replicazione cromosomica. Nel clessico modello del replicone, le proteine iniziatrici controllano l’inizio della replicazione mediante specifiche regioni cromosomiche, chiamate “origini di replicazione”. Nei batteri la regolazione maggiore è data dalla proteina iniziatrice DnaA (una ATPasi). Il cromosoma batterico tipicamente contiene una sola zona di origine della replicazione, chiamata OriC. Questo meccanismo è strutturalmente simile a quello presente negli archea. OriC in E. coli è formato da 250 bp, e contiene ripetizioni multiple di frammenti da 9 bp, chiamati DnaA-box, che sono specifici siti di legame per la proteina iniziatrice DnaA. DnaA si lega con massima affinità alla sequenza consenso 5’-TTATCCAC-3’ del DnaA-box. Inoltre, DnaA si può legare ad altre regioni con sequenze consenso differenti, chiamate siti-I, che si trovano in maniera dispersa intorno ad OriC. Adiacente a DnaA-box e siti-I è presente il DUE (AT-rich DNA-unwinding element), che contiene una terza categoria di sequenze che legano DnaA: gli ATP-DnaA-box. L’interazione tra DnaA e OriC permette di assemblare il complesso di inizio replicazione. DnaA rimane legato a tre siti DnaA-box, interagendo con altri siti durante il ciclo replicativo. Quando DnaA si localizza su OriC, forma polimeri (visibili al microscopio elettronico) per facilitare il legame di DUE (che divide il doppio filamento in due singoli filamenti), che permette di caricare l’elicasi e il macchinario di replicazione. Dopo lo srotolamento di DUE, un’altra importante funzione di DnaA è reclutare la DnaB elicasi; il caricatore dell’elicasi, DnaC, è necessario per questa operazione.

Microbiologia DIVISIONE BATTERICA Antonio Nenna
DnaA (della famiglia delle AAA, ATPasi associate ad attività cellulare) è formata da quattro domini: 1) N-term, media l’interazione con l’elicasi DnaB e facilita la polimerizzazione 2) connessione 3) attività ATP-asica (necessaria per formare il complesso della nucleoproteina) 4) C-term, sequenza di legame con il DNA (per OriC); sono necessari due elementi per il funzionamento DnaA-signature-sequence, permette di riconoscere il DnaA-box; si trova nella piega di una sequenza helix-turn-helix (HLH), all’interno del solco maggiore del DNA ansa basica, con un residuo di arginina per legarsi al DNA, all’interno del solco minore del DNA L’interazione ad alta affinità tra DnaA e DnaA-box localizza DnaA in OriC; l’interazione con ATP-DnaA-box, di bassa affinità, permette a DnaA di formare un polimero che permette l’interposizione di DUE.
Sia OriC che DnaA sono soggette a controllo regolatorio. Il processo di “sequestro dell’origine” assicura che le origini appena replicate siano temporaneamente impossibilitate a iniziare un altro ciclo di duplicazione; l’autoregolazione di DnaA limita sia la disponibilità che l’attività del fattore di inizio. Questo schema di controllo regola l’assemblaggio di DnaA su OriC, la durata del legame e la dissoluzione del complesso. OriC contiene 11 siti GATC all’interno dell’array di DUE. Queste sequenze sono normalmente tutte metilate in adenina dalla DAM-metiltrasferasi; dopo il passaggio della forca di replicazione, i nuovi siti GATC si trovano in maniera transiente in stato emimetilato. La proteina SeqA si lega alle sequenze emimetilate e permette il requestro per impedire la re-iniziazione della duplicazione. Sia SeqA che DnaA formano dimeri disposti a filamento, che aiuta il legame con il DNA. Per la DnaA, lo stato filamentoso aiuta a superare la differenze di struttura nell’origine di replicazione tra i differenti batteri. Per la SeqA, lo stato filamentoso permette il requestro rapido di nuovi OriC. Il controllo dell’attività della DnaA avviene mediante stimolo (autoregolazione) e inibizione (RIDA, situata sul caricatore della pinza, stimola l’idrolisi dell’ATP della DnaA dopo il legame con DUE; YabA in B. subtilis che interagisce con la DnaA).

Microbiologia DIVISIONE BATTERICA Antonio Nenna
INIZIO DELLA REPLICAZIONE La replicazione inizia ad OriC e procede bidirezionalmente, creando due forcelle di replicazione che entrano nel DNA dalla stessa origine. Le forcelle si muovono intorno al cromosoma circolare a 1000 bp/s e quindi dopo circa 40 minuti raggiungono il lato opposto di OriC.

Microbiologia DIVISIONE BATTERICA Antonio Nenna
TERMINAZIONE DELLA REPLICAZIONE Al lato opposto di OriC si trovano i siti di terminazione (Ter), che creano una trappola per la forcella di replicazione e permettono di entrare ma non di lasciare la sequenza di terminazione. L’interazione tra la proteina terminatrice della replicazione (Tus, termination utilization substance) e Ter genera l’arresto della forcella.
La sequenza Ter è formata da 23 bp con resisui conservati di GC, seguiti da una regione core di 13 bp con sostituzioni. La sequenza è asimmetrica, speculare all’asimmetria della forcella. La forcella di replicazione che arriva da sinistra è bloccata mentre quella che arriva da destra passa normalmente; la sequenza core è associata con il sito di bloccaggio della forca, preceduto da una regione ricca di AT. La proteina Tus p codificata da un gene situato 11 bp downstream a TerB. La sequenza di ShineDalgarno del promotore si sovrappone a TerB: la proteina Tus regola l’espressione del proprio gene legandosi alla sequenza TerB e bloccando l’inizio della trascrizione.
Il complesso Tus-Ter blocca l’azione della DNA elicasi (DnaB). Nel normale processo di replicazione, DnaB è di fronte al replisoma; è un enzima omoesamerico che srotola il DNA a doppio filamento di fronte alla DNApolIII, la replicasi che sintetizza contemporaneamente entrambi i filamenti; DnaB è associata fisicamente al replisoma tramite la subunità γ. In presenza del complesso Tus-Ter, la sintesi del DNA è alterata fino a 4 bp prima della sequenza ricca in GC nel sito TerB; il filamento guida viene escluso dal canale della DnaB.

Microbiologia DIVISIONE BATTERICA Antonio Nenna

Microbiologia DIVISIONE BATTERICA Antonio Nenna
CITOCHINESI BATTERICA E. coli: processo di costrizione cellulare; il peptidoglicano, funzionando da esoscheletro della cellula batterica, deve essere sintetizzato per il processo di divisione (azione degli antibiotici). B. subtilis: crescita del setto di divisione. Molte proteine sono implicate nella divisione cellulare, molte delle quali sono della famiglia Fts, che sono ancorate alla membrana citoplasmatica e il loro dominio principale aggetta nel citoplasma o nel periplasma. L’assemblaggio del divisoma inizial con il posizionamento dell’anello di FtsZ al centro della cellula, stabilizzato da FtsA e ZipA; poi, altre proteine Fts vengono reclutate, formando diversi subassemblaggi.
FtsZ è una GTPasi simile alla tubulina mentre FtsI è la PBP3. I componenti della sintesti del peptidoglicano quindi non importanti per la divisione cellulare. La sintesi del PG specifica per la divisione inizia allo stesso tempo del posizionamento di FtsZ intorno al centro della cellula, suggerendo una connessione tra la polimerizzazione citoplasmatica di FtsZ e la sintesi periplasmatica di peptidoglicano. Il meccanismo contrattile basato sul complesso di actina-miosina sembra improbabile nei batteri, poichè non sono state trovate analoghe proteine motrici. Visto che il diametro del divisoma di E. coli diminuisce durante la divisione, deve avvenire la depolimerizzazione di FtsZ; ciò è dovuto alla proteina EzrA, che controlla la depolimerizzazione. La formazione dell’anello FtsZ coincide con il termine della replicazione del cromosoma, e la segregazione dei cromosomi segue la depolimerizzazione di FtsZ. La localizzazione del piano di divisione è direttamente correlata alla posizione intracellulare del cromosoma batterico. Le proteine Min, se mutate, producono minicellule prive di DNA. E. coli contiene tre potenziali siti di divisione, uno al centro e due ai poli. La combinazione di MinC e MinD inibisce la polimerizzazione di FtsZ al potenziale sito di divisione, a meno che MinE sia presente (la polimerizzazione di FtsZ è inibita da MinCD, e l’inibizione è rimossa da MinE). Quindi MinE impedisce l’inibizione della polimerizzazione di FtsZ al centro della cellula, infatti un anello di MinE è stato osservato al centro della cellula indipendentemente dall’anello FtsZ. L’assemblaggio di FtsZ è quindi soggetto a una doppia regolazione negativa.

Microbiologia DIVISIONE BATTERICA Antonio Nenna
La replicazione del cromosoma batterico circolare inizia dall’origine di replicazione e procede in maniera bidirezionale, fino a raggiugnere il punto opposto, chiamato terminazione. Il caricamento della DnaA su OriC causa uno srotolamento localizzato, e questo processo viene continuato dalla DNAelicasi. Lo srotolamento dell’origine di replicazione permette l’assemblaggio del complesso di replicazione del DNA, il replisoma. L’effetto “bobina” del filamento nascente fornisce la forza motrice per la segregazione del DNA. Questa azione di spinta è coadiuvata dalla azione di tiraggio dovuta alla condensazione del DNA neoformato ad opera delle proteine SMC. Altri fattori, come il sistema Par, sono importanti per la direzionalità di movimento del cromosoma, cioè spingere i filamenti verso i poli della cellula. Quindi i fattori che contribuiscono al movimento dei cromosomi sono differenti: il replisoma (macchinario per la replicazione del DNA) è localizzato al centro della cellula (R); le regioni OriC appena duplicate (O) migrano rapidamente verso i poli opposti della cellula; la forza motrice per il movimento è la polimerizzazione del DNA ad opera del replisoma stazionario. Altre proteine, come MukB o SMC, sono importanti per il partizionamento dei cromosomi: queste proteine disassemblano il DNA per permettere il passaggio della forcella di replicazione e riassemblano le regioni appena duplicate del genoma. Questa attività genera condensazione, e in collaborazione con il movimento di OriC, causano lo spostamento del DNA parentale e neosintetizzato verso i poli opposti della cellula. Il completamento della replicazione vede la presenza della regione terminale al centro della cellula, e i dimeri di DNA sono scissi da XerCD tramite una reazione stimolata da FtsK; quindi la divisione cellulare e la segregazione cromosomica sono appaiati. Anche il ruolo delle proteine Par è importante nel partizionamento del DNA, poichè anomalie impediscono la formazione dell’anello di FtsZ. Se il rapporto ParA / ParB aumenta, viene inibita la divisione cellulare.

Microbiologia DIVISIONE BATTERICA Antonio Nenna
ASSEMBLAGGIO DEL DIVISOMA In E. coli, i componenti del divisoma sono reclutati al centro della cellula in base a una precisa gerarchia che predice la modalità di assemblaggio. Negli anni ’60, alcuni batteri mutanti non riuscivano a duplicarsi poichè producevano cellule filamentose ad alte temperature; queste mutazioni permisero l’identificazione di geni fts (filamentation thermosensitive), i cui prodotti sono essenziali per la divisione cellulare. Questi mutanti si replicano e segregano i cromosomi normalmente, ma non riescono a dividersi e quindi acquisiscono un fenotipo filamentoso. FtsZ La proteina FtsZ è la più abbondante della famiglia, e si localizza al centro della cellula. E’ una GTPasi che forma una struttura a forma di anello, chiamato anelloZ. Le 15000 copie di FtsZ per cellula sono sufficienti a formare un anello composto da più filamenti. L’anelloZ è utilizzato come zattera per l’assemblaggio degli altri componenti del divisoma. E’ conservato tra tutti i batteri, ad eccezione di Chlamidia. Analogamente alla tubulina, FtsZ polimerizza utilizzanto GTP, e l’anelloZ è una struttura estremamente dinamica, poichè i monomeri passano dal citoplasma all’anello circa ogni 10 secondi. FtsZ è soggetta a rapido assemblaggio e disassemblaggio, permettendo alla cellula di regolare rapidamente la formazione dell’anelloZ. Due proteine servono a stabilizzare FtsZ: FtsA e ZipA. Se entrambe sono mutate, non si forma l’anelloZ. Queste proteine riconoscono FtsZ mediante il dominio C-term e servono a legarlo alla membrana. MinCDE Tre proteine, codificate tutte dall’operone MinCDE, cooperano per formare un sistema oscillatorio autogestito. MinD è una ATPasi e interagisce con MinC per formare un inibitore di FtsZ associato alla membrana. L’attività di MinE restringe il complesso MinCD ai poli della cellula, quindi permettendo che la divisione cellulare avvenga solo al centro della cellula. MinCD oscilla tra i poli della cellula circa ogni minuto: si assembla da un lato, e poi questa struttura a cappuccio perde subunità fino a sparire; allo stesso tempo, si assembla dall’altro lato e il ciclo ricomincia. MinE forma una struttura circolare che causa la retrazione del complesso MinCD. In assenza di MinE, si perde il comportamento oscillatorio del sistema, e MinCD si forma su tutta la membrana, inibendo la divisione cellulare. Il sistema è differente per i diversi batteri. MinCDE è capace di stabilire un’oscillazione sull’asse maggiore della cellula, riconoscendo anche piccole asimmetrie nella iniziale cellula sferica generata dalla divisione cellulare dei bastoncelli; quindi è capace di riconoscere l’asse maggiore della cellula, definendo il piano di divisione cellulare. MipZ Esistono organismi che non possiedono il sistema MinCDE ma riescono a dividersi al centro della cellula, e questo suggerisce l’esistenza di altri meccanismi di regolazione. MipZ è una ATPasi che interagisce con la proteina ParB e si lega a un sito (ParS) vicino OriC. Questo complesso si posiziona al polo “vecchio” della cellula in replicazione. L’inizio della replicazione genera due segmenti ParS, che vengono subito raggiunti da ParB-MipZ. Nella seguente segregazione, uno di questi segmenti rimane nella posizione originale, mentre l’altra copia si muove attraverso la cellula verso il polo opposto. MipZ agisce come un inibitore della polimerizzazione di FtsZ. MreB MreB è una proteina del citoscheletro, equivalente procariotico dell’actina. Il gene fu identificato in E. coli come quel gene che permette di mantenere la forma a bastoncello della cellula, infatti MreB e omologhi sono presenti nei bastoncelli ma mancano nei cocchi. Inoltre è importante per la segregazione dei cromosomi (poichè lega le sequenze vicino OriC) e aiuta il targeting di proteine dirette ai poli cellulari (con l’eccezione delle proteine MinCDE)

Microbiologia DIVISIONE BATTERICA Antonio Nenna
SELEZIONE DEL SITO DI FORMAZIONE DEL DIVISOMA Prima di dividersi, è importante per la cellula scegliere il sito dove avverrà la divisione, determinando la posizione dell’anelloZ: ciò è garantito da due processi. 1) “nucleoid occlusion”: tendenza dell’anelloZ di formarsi in regioni prive di DNA [inibizione della formazione di FtsZ dovuta alla presenza del nucleoide, mediata dalle proteine Noc e SlmA] 2) “Min proteins”: inibizione dell’anelloZ in regioni prive di DNA ai poli della cellula *la delezione di queste proteine causa la formazione dell’anelloZ nei poli e la produzione di minicellule prive di DNA+ Quindi queste due strategie garantiscono che l’anelloZ si formi solo in regioni prive di DNA al centro della cellula dopo la segregazione dei cromosomi. Inoltre il sistema SOS viene attivato in caso di danni al DNA, e recluta una proteina inibente la settazione (SulA) che impedisce la formazione dell’anelloZ e quindi impedisce la formazione del setto di divisione, inibendo la divisione cellulare. Alcuni batteri non si dividono al centro della cellula, come gli sporigeni (il setto di divisione si trova al polo cellulare). Inoltre si producono più spore per ogni batterio, con la formazione di setti multipli interni di divisione. Il primo evento è comunque la formazione dell’anelloZ ad entrambi i poli della cellula, ma solo uno di questi verrà utilizzato per formare il setto di divisione per le spore. Due fattori sono necessari per indurre la formazione del duplice anelloZ: un aumento della concentrazione di FtsZ (mediato dall’aumento della trascizione del fene Fts all’inizio della sporulazione) e la proteina Spo2E (una fosfatasi che interagisce con FtsZ e serve alla compartimentazione dell’espressione genica durante la sporulazione).

Microbiologia DIVISIONE BATTERICA Antonio Nenna

Microbiologia DIVISIONE BATTERICA Antonio Nenna
CITOCHINESI Quando la cellula si divide, l’anelloZ si costringe generando direttamente la forza per la citochinesi, in modo analogo all’interazione tra actina e miosina negli eucarioti. La citochinesi avviene in due momenti, e FtsZ è necessaria per entrambi (se il gene Fts è spostato su temperature non permissive, l’anelloZ scompare e tutte le altre proteine coinvolte nella citochinesi vengono degradate, e il setto smette di invaginarsi): 1) formazione di una piccola invaginazione nel sito del setto di divisione 2) ispessimento del setto di divisione
I cocchi come S. aureus sintetizzano nuovo cell wall all’equatore. La graduale costrizione di quest’anello genera i due nuovi poli delle cellule figlie. Quindi la sintesi del cell wall al centro della cellula è sufficiente a guidare la divisione dei cocchi. I bastoncelli, al contrario, utilizzano due differenti modi di divisione. Nella fase di allungamento, il nuovo cell wall è inserito in maniera diffusa sulle pareti laterali; ad un momento specifico, il cell wall viene allungato solo sul setto di divisione. Qui avviene il bivio tra le due modalità di divisione: i bastoncelli gram-positivi (B. subtilis) sintetizzano il setto tra le due cellule figlie e si liberano perchè delle autolisine erodono il rivestimento di mureina; i bastoncelli gram-negativi (E. coli) si dividono per costrizione degli strati del cell wall (analogamente ai cocchi).

Microbiologia DIVISIONE BATTERICA Antonio Nenna
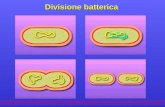
riassunto degli eventi maggiori nella divisione di una cellula batterica