Nel 1955, Arthur Kornberg e collabo- La scoperta della...
Transcript of Nel 1955, Arthur Kornberg e collabo- La scoperta della...
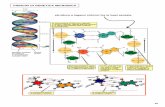
La molecola di DNA è composta da due filamenti di nucleotidi legatida ponti di idrogeno: desossiadenosina-5'-monofosfato, desossiti-midina-5' -monofosfato, desossiguanosina-5' -monofosfato e desos-sicitidina-5'-monofosfato di cui sono indicate le basi: A, adenina;
T, timina; G, guanina; C, citosina. I nucleotidi di un filamento sonocomplementari a quelli dell'altro, cioè A è sempre appaiata con Te G con C. Ciascun filamento ha un'estremità 3' e una 5' e orien-tazione opposta all'altro; i filamenti sono quindi antiparalleli.
T
alvolta le buone idee vengonoproprio quando non le si cerca.Così, un venerdì sera dell'aprile
1983, mentre al volante della mia au-tomobile procedevo lungo una serpeg-giante strada di montagna illuminatadalla luna, nella regione ricca di bo-schi di sequoie della California setten-trionale, per un'improbabile combina-zione di coincidenze, di ingenuità e dierrori fortunati ebbi una specie di ri-velazione: escogitai un processo che po-teva produrre un numero illimitato dicopie di geni. Questo processo vienechiamato oggi reazione a catena dellapolimerasi (PCR).
A partire da un'unica molecola diDNA, la reazione a catena della poli-merasi può generare 100 miliardi di mo-lecole simili in un solo pomeriggio. E,per di più, la reazione può essere esegui-ta facilmente: bastano una provetta, al-cuni semplici reagenti e una fonte di ca-lore. Il campione di DNA di cui si vo-gliono ottenere copie può essere purooppure può far parte di un miscuglioestremamente complesso di materialibiologici. Il DNA può provenire da unprelievo bioptico effettuato in ospeda-le, da un capello umano, da una gocciadi sangue trovata sul luogo di un delit-to, dal tessuto cerebrale di una mummiao dal corpo di un mammut risalentea 40 000 anni fa, ritrovato congelatonel terreno.
Nei sette anni trascorsi da quella serale applicazioni della reazione a catenadella polimerasi si sono diffuse in ognicampo della biologia e sono stati pubbli-cati più di 1000 articoli sull'argomento.Considerando gli effetti profondi avutidal metodo sulla ricerca biologica e lasua semplicità concettuale, molti osser-vatori si stupiscono del fatto che esso siastato trascurato per oltre 15 anni, benchéfossero disponibili tutti gli elementi perpoterlo sfruttare.
I a reazione a catena della polimerasiJ--d rende molto più agevole il compitodegli studiosi di biologia molecolare, da-to che fornisce la quantità voluta di unparticolare DNA. Talvolta, quando siparla superficialmente di molecole diDNA, si ha l'impressione che esse sianoottenibili senza difficoltà. La verità èche, in pratica, non è facile ottenere unamolecola ben definita di DNA naturaleda un organismo qualsiasi, a meno chenon si tratti di virus molto semplici.
La difficoltà risiede nella natura dellamolecola. Il DNA è una catena fragile,costituita da quattro desossiribonucleo-tidi: il dAMP (desossiadenosina-5'-mo-nofosfato), il dTMP (desossitimidina-5'--monofosfato), il dGMP (desossiguano-sina-5'-monofosfato) e il dCMP (desos-sicitidina-5'-monofosfato). La sequenzadi questi desossiribonucleotidi (o dellerispettive basi A, T, G e C) specifical'informazione genetica. Raramente sitrova un singolo filamento di DNA; disolito coppie di filamenti con sequenzecomplementari formano una doppia eli-ca in cui le basi A di un filamento silegano con le T dell'altro filamento e leG con le C (si veda l'illustrazione nellapagina a fronte). All'interno della cellu-la, questa molecola di DNA è circondatae ulteriormente avvolta a elica da varieproteine. Quando i biologi cercano diisolare un filamento di DNA «nudo»,questo è così lungo e sottile che ancheforze modeste lo spezzano casualmentein vari punti. Di conseguenza, rimuo-vendo il DNA da 1000 cellule identichevi sarebbero 1000 copie di un dato gene,ma ogni copia si troverebbe su un fram-mento di DNA di differente lunghezza.
Per anni, questo problema rese arduolo studio dei geni. Poi, negli anni settan-ta, furono scoperte le endonucleasi direstrizione, enzimi che scindono i fila-menti di DNA in punti specifici. Le en-donucleasi hanno permesso di dividere il
DNA in segmenti più piccoli, più resi-stenti e meglio identificabili, rendendocosì più facile l'isolamento dei segmentiche contenevano il gene in esame.
Pertanto, già alla fine degli anni set-tanta, gli esperti di biologia molecolarestudiavano il DNA servendosi delle en-donucleasi e di altre molecole chiamate«sonde oligonucleotidiche». Un oligo-nucleotide è una corta catena di basi nu-cleotidiche disposte in un ordine specifi-co. Nelle condizioni opportune, un oli-gonucleotide si lega in modo specifico auna sequenza complementare di nucleo-tidi presenti in un filamento singolo diDNA. Pertanto oligonucleotidi sintetici,marcati con isotopi radioattivi, possonofungere da sonde per determinare se uncampione di DNA contiene una data se-quenza nucleotidica, ossia uno specificogene. Nel 1979, la Cetus Corporation diEmeryville, in California, mi affidò l'in-carico di sintetizzare queste sonde.
Nel 1983 l'aspetto affascinante dellasintesi di oligonucleotidi cominciò a de-clinare e la maggior parte di coloro chelavoravano in questo settore fu lieta diessere testimone di un simile declino. Imetodi laboriosi, ma attraenti con cuivenivano sintetizzati manualmente glioligonucleotidi e ai quali ci eravamo co-modamente abituati erano stati sostituitida una tecnica automatizzata, molto me-no entusiasmante ma affidabile. Fu unimmenso passo in avanti.
A causa di questa piccola rivoluzioneindustriale noi chimici esperti in nucleo-tidi ci trovammo felicemente sottoccu-pati. In laboratorio le macchine, che noicaricavamo e sorvegliavamo, produce-vano una quantità di oligonucleotidi ad-dirittura maggiore di quella che poteva-mo sistemare nelle celle frigorifere e cer-tamente maggiore di quella che gli stu-diosi di biologia molecolare - che sem-bravano operare in modo ancora più len-to e noioso di quanto avessimo pensato
in un primo tempo - potevano utilizzarenei loro esperimenti. Quindi nel mio la-boratorio alla Cetus disponevo di parec-chio tempo libero; fu così che mi trovaia pasticciare con gli oligonucleotidi.
Mi rendevo conto che sarebbe statoutile disporre di una tecnica per de-
terminare facilmente l'identità di un nu-cleotide in una data posizione su unamolecola di DNA, specialmente se que-sta tecnica avesse funzionato con unDNA complesso (come quello umano) edisponibile solo in quantità limitate.Non vedevo alcuna ragione per non uti-lizzare la DNA-polimerasi e una varian-te di una tecnica chiamata «sequenzia-mento didesossi»; così, per verificarequesta ipotesi di lavoro, progettai unsemplice esperimento.
Per capire il procedimento che avevoin mente, vale la pena di richiamare al-cune nozioni sul DNA. Un filamento diDNA ha un'estremità che, per conven-zione chimica, è chiamata estremità 3' euna che è denominata estremità 5'. Inuna doppia elica di DNA, si dice che ifilamenti complementari sono antiparal-leli nel senso che l'estremità 3' di un fi-lamento si appaia con l'estremità 5' del-l'altro filamento e viceversa.
Nel 1955, Arthur Kornberg e collabo-ratori della Stanford University scopri-rono una DNA-polimerasi, un enzimacellulare che in natura assolve diversefunzioni, tra cui la riparazione e la du-plicazione del DNA. La DNA-polime-rasi può allungare un breve oligonucleo-tide che funge da «innesco» attaccandoun ulteriore nucleotide all'estremità 3';questo però accade solamente se l'inne-sco è ibridato, o legato, con un filamen-to «stampo» complementare. La solu-zione circostante deve anche contene-re molecole di nucleosiditrifosfati (unnucleoside è formato da una base e dauno zucchero; un nucleoside fosforilatoè un nucleotide) che fungono da unitàcostruttive.
Il nucleotide che la polimerasi attac-ca alla catena è complementare a quel-lo che si trova nella posizione corri-spondente sullo stampo. Per esempio, seil nucleotide adiacente sullo stampo èdAMP, la polimerasi attacca un nucleo-tide dTMP; se il nucleotide dello stampoè dGMP, la polimerasi attacca un nu-cleotide dCMP. Ripetendo questa ope-razione, l'enzima riesce ad allungare l'e-stremità 3' dell'innesco fino a raggiunge-re l'estremità 5' dello stampo (si vedal'illustrazione a pagina 35). In una dop-
pia elica di DNA ogni filamento fungeda stampo per l'altro filamento nel corsodella duplicazione e della riparazione.
Passiamo ora al sequenziamento dide-sossi, comunemente noto come tecnicadi Sanger, dal nome di uno dei suoi sco-pritori, Frederick Sanger del BritishMedical Research Council Laboratoryof Molecular Biology. Per determina-re le sequenze di DNA, la tecnica ricor-re a una DNA-polimerasi, a filamentistampo, a inneschi, a nucleosiditrifosfatie a speciali didesossinucleosiditrifosfati(ddNTP). Come i normali nucleotidi, iddNTP possono essere attaccati dallepolimerasi alle catene degli inneschi invia di allungamento, ma formano un«cappuccio» all'estremità 3' che impedi-sce l'aggiunta di ulteriori nucleotidi. Latecnica di Sanger produce inneschi allun-gati in varia misura e quindi «incappuc-ciati» da un ddNTP. Ordinando questisegmenti in funzione della lunghezza esapendo quali ddNTP sono stati aggiun-ti, un ricercatore può determinare la se-quenza di basi nel filamento che fungeda stampo. Per esempio, se in una deter-minata posizione viene aggiunta dide-sossiadenina (ddA), la base complemen-tare corrispondente nello stampo deveessere T; l'aggiunta di una didesossigua-
La scoperta della reazionea catena della polimerasi
Un modo sorprendentemente semplice per produrre in numero illimitatocopie di segmenti di DNA è stato escogitato in circostanze inconsuete,una sera in auto, al chiaro di luna, tra le montagne della California
di Kary B. Mullis
32 LE SCIENZE n. 262, giugno 1990 LE SCIENZE n. 262, giugno 1990 33
32 COPIE 64 COPIE
1~1•1111111~, MIIMInv-~~-~11~
411M
La reazione a catena della polimerasi consente di copiare un seg-mento di DNA in laboratorio utilizzando reagenti di facile reperibi-
lità. Dato che il numero di copie aumenta esponenzialmente, in po-che ore si possono produrre anche 100 miliardi di molecole di DNA.
,
4 COPIE
2 COPIE
MI111111n11111111
~1111n1111,~1n1•1/,~11111"Mi
011111111111111111111111~
111111•11111~2~1511111111111,
\ \111111~111111"..
itges~~1~1"
1111•1111MMIP
milimume
wwwwwwwwer
1111111111~1~1el•MMIII•1n11
8 COPIE
16 COPIE
1 COPIA
111111111~11.
CA T A
T
T
v
TA C T A
A T c5,
POLIMERASI
c5,
nina (ddG) sottintende la presenza di Cnello stampo.
Nella versione modificata di questatecnica alla quale mi stavo interessando,avrei usato soltanto polimerasi, stampi,ddNTP e inneschi, omettendo cioè dallamiscela di reazione i nucleosiditrifo-sfati normali. Pertanto l'allungamentodegli inneschi si sarebbe concluso im-mediatamente dopo l'aggiunta alla cate-
na di un nucleotide proveniente da unddNTP. Sapendo quale ddNTP era statoaggiunto agli inneschi, avrei anche co-nosciuto l'identità del nucleotide corri-spondente nel filamento stampo. In que-sto modo sarei stato in grado di de-durre l'identità di un nucleotide delfilamento stampo situato in posizioneadiacente al sito in cui si era legatol'innesco.
IIfatto di cui non mi resi conto al mo-mento era che, per molti validi moti-
vi, la mia idea di sequenziamento nonpoteva funzionare. Il problema era chegli oligonucleotidi si ibridano talvoltacon sequenze di DNA diverse da quellevolute: questi appaiamenti inevitabili miavrebbero dato risultati ambigui. Anchenelle mani di ricercatori abilissimi nel-l'arte dell'ibridazione, era impossibile
legare oligonucleotidi all'intero DNAumano con specificità sufficiente a otte-nere un risultato almeno lontanamentesignificativo.
Proprio per questa limitazione i ricer-catori erano ricorsi a procedimenti piùcomplessi per analizzare il DNA umano.Per esempio, per scindere il campione diDNA in vari frammenti che potevanopoi essere separati l'uno dall'altro me-diante elettroforesi, venivano utilizzatigli enzimi di restrizione; in questo modo,prima dell'ibridazione con le sonde oli-gonucleotidiche , il campione poteva es-sere in qualche misura «purificato», eli-minando tutto il DNA tranne il fram-mento che interessava. Questo procedi-mento riduceva le ibridazioni errate inmisura appena sufficiente a fornire risul-tati significativi, ma era laborioso e nonpoteva essere applicato a campioni diDNA degradati o denaturati.
Un'altra tecnica troppo laboriosa perun'analisi di routine del DNA compor-tava la donazione. Una sequenza inte-ressante di DNA umano poteva essereclonata, o copiata, in un plasmide, ossiaun piccolo anello di DNA. Copie di que-sto plasmide e della sequenza in esamepotevano quindi essere prodotte in bat-teri e l'informazione sulla sequenza po-teva essere ricavata mediante ibridazio-ne con sonde oligonucleotidiche e se-quenziamento didesossi. Agli inizi deglianni ottanta questo procedimento prati-cato su DNA clonato servì a ottenere lamaggior parte dell'informazione oggi di-sponibile sul DNA umano.
Nel proporre il mio semplicistico espe-rimento, davo implicitamente per scon-tato che la donazione o altre fasi preli-minari non sarebbero state necessarieper identificare sequenze specifiche nelDNA umano mediante una singola ibri-dazione con sonde oligonucleotidiche.Per difendere le mie elucubrazioni possofar notare che, nel laboratorio accanto almio, un gruppo diretto da uno dei piùautorevoli scienziati della Cetus, HenryA. Erlich, stava provando un altro me-todo basato sull'ibridazione di un singo-lo oligonucleotide con il DNA umanocercato (quello che chiamavamo DNAbersaglio). Nessuno avrebbe osato ride-re di Henry ed entrambi venivamo pa-gati regolarmente. In realtà, eravamopagati sufficientemente bene da indurrealcuni di noi a credere, forse un po' au-dacemente, che dovevamo essere pros-simi a una grande svolta nella tecnolo-gia del DNA.
Un venerdì sera, sul finire della pri-mavera, stavo recandomi in auto
nella Mendocino County assieme a unachimica, mia amica. La strada statale101 non richiedeva particolare attenzio-ne e la mia compagna di viaggio dormi-va. Mi piace guidare di sera e ogni finesettimana avevo preso l'abitudine di re-carmi al nord per passare un paio di gior-ni nella mia casa di campagna. Me nestavo al volante per tre ore, con le mani
occupate e la mente sgombra. Quella se-ra, in particolare, pensavo al mio espe-rimento di sequenziamento del DNA.
Il mio progetto era semplicissimo. Inprimo luogo avrei separato il segmentodi DNA prescelto nei singoli filamentimediante riscaldamento. Quindi avreiibridato un oligonucleotide con una se-quenza complementare presente su unodei due filamenti. Avrei poi messo unpo' di questo miscuglio di DNA in quat-
3'
POLIMERASI
POLIMERASI
tro differenti provette. Ogni provettaavrebbe contenuto tutti e quattro i tipidi ddNTP, ma in ciascuna di esse unddNTP diverso sarebbe stato marcatocon un isotopo radioattivo. Quindi avreiaggiunto la DNA-polimerasi, che avreb-be allungato di un solo ddNTP gli oligo-nucleotidi ibridati in ogni provetta. Me-diante elettroforesi avrei poi potuto se-parare gli oligonucleotidi allungati daiddNTP residui e, identificando quale
3'
A
T
d A
A
T
5'
5'
5'
5
AT
T
k.M."
A
T
A
v
5'
L'enzima DNA-polimerasi può far allungare un corto filamento di DNA - l'innesco oligo-nucleotidico - se il filamento stesso è legato a un filamento più lungo di DNA che funge dastampo. L'enzima aggiunge l'appropriato nucleotide complementare all'estremità 3' del-l'innesco legato. Se però si aggiunge un didesossinucleosidetrifosfato (ddNTP), per esem-pio il didesossiadenosintrifosfato (ddATP), non si ha alcun allungamento, perché l'estre-mità 3', dove è presente la base di didesossiadenina (ddA), non si lega ad altri nucleotidi.
34 LE SCIENZE n. 262, giugno 1990
LE SCIENZE n. 262, giugno 1990 35
3'A C
C
T
A
T
AT
A
T
COPPIA DI BASIIN ESAME
I FILAMENTI SEPARATI MEDIANTE FUSIONESI ATTACCANO AGLI INNESCHI
v3'
A
C T
T
A
3'
C
T
A
AC
/N
T
T
T
A
N./
A
C _J
A C
/N
ddC
A
A A
C
T
T
C
A
N/
/N
T
A
A
T
3
ddG1 '
T
A
A
T
L'IDENTITÀ DEI ddNTP AGGIUNTI CONSENTEDI RICONOSCERE LE BASI DELLA COPPIA IN ESAME
A
T
A
T
A
T
Per determinare l'identità di una coppia di basi in un segmento diDNA l'autore sperava di utilizzare una variante di una tecnicachiamata «sequenziamento didesossi». Dapprima due inneschi ven-gono legati ai filamenti opposti della molecola di DNA in punti adia-centi alla coppia di basi prescelta. Poi si aggiungono alla miscela direazione DNA-polimerasi e didesossinucleosiditrifosfati (ddNTP),
per far sì che ogni innesco si allunghi di un solo nucleotide. Le ba-si dei ddNTP aggiunti indicano quali sono le basi complementarinel DNA bersaglio. La tecnica potrebbe funzionare anche con unsolo innesco, ma utilizzandone due si dispone di un controllo perverificare i risultati. La progettazione di questo esperimento haportato l'autore a scoprire la reazione a catena della polimerasi.
ddNTP marcato era stato incorporatonell'oligonucleotide , avrei determinatonel filamento stampo la corrispondentebase complementare. Facile!
Presso Cloverdale, dove la strada sta-tale 128 si dirama dalla 101 verso nordo-vest e sale serpeggiando attraverso la ca-tena costiera, decisi che la determinazio-ne della sequenza sarebbe stata più pre-cisa se, al posto di un solo oligonucleo-tide , ne avessi utilizzati due. I due inne-schi avrebbero «chiuso tra parentesi» lacoppia di basi che speravo di identificaree, preparando oligonucleotidi di diffe-renti dimensioni, sarei stato in grado didistinguerli l'uno dall'altro. Se avessi di-retto un oligonucleotide verso ciascun fi-lamento del campione di DNA avrei po-tuto ottenere per entrambi i filamenti in-formazioni complementari sulle sequen-ze. Pertanto, l'esperimento avrebbe po-tuto disporre di un controllo interno sen-za alcun inconveniente aggiuntivo (si ve-da l'illustrazione nella pagina a fronte).
Sebbene al momento non me ne ren-dessi conto - con quei due oligonucleo-tidi che si libravano nella mia mente, leestremità 3' dirette l'una contro l'altrasui filamenti opposti del gene prescelto -ero arrivato a una svolta: ero sul puntodi scoprire la reazione a catena della po-limerasi . Ma in realtà le svolte a cui inquel momento badavo di più erano quel-le della strada di montagna che stavopercorrendo.
uella sera l'aria era satura di umiditàe del profumo dei fiori. Pensavo ai
nuovi laghetti che stavo scavando nellamia proprietà e, nello stesso tempo, ri-muginavo su tutte quelle cose che avreb-bero potuto non funzionare nell'esperi-mento di sequenziamento delle basi.
Dai miei studi di specializzazione svol-ti nel laboratorio di Wolfgang Sadee al-l'Università della California a San Fran-cisco, dove John Maybaum stava met-tendo a punto prove cliniche per i nu-cleotidi, ricordai che i miei campioni diDNA potevano contenere tracce inci-dentali di nucleosiditrifosfati. L'inter-pretazione del gel sarebbe stata più dif-ficile, mi dicevo, se queste impurezze in-trodotte con il campione si fossero addi-zionate all'estremità 3' degli inneschiprima dell'aggiunta dei ddNTP marcati.
Un'idea che mi passò per la mente eraquella di distruggere tutti i nucleosiditri-fosfati nel campione con un enzima bat-terico, la fosfatasi alcalina, che era ingrado di staccare i gruppi fosfato reattivida una molecola di nucleosidetrifosfato,rendendo così il composto incapace diprendere parte alla reazione a catenadella polimerasi. A questo punto dovevoperò eliminare in qualche modo la fosfa-tasi dal campione per evitare che distrug-gesse anche i ddNTP, quando li avessiaggiunti alla miscela di reazione. Nor-malmente è possibile inattivare enzimiindesiderati sottoponendoli a riscalda-mento in modo da alterarne la confor-mazione di base; pensavo, però, che la
fosfatasi alcalina batterica potesse riav-volgersi assumendo di nuovo la confor-mazione originale. Pertanto la scartaicome soluzione del problema.
In realtà mi sbagliavo. Molto tempodopo appresi che la fosfatasi alcalina puòessere irreversibilmente denaturata me-diante riscaldamento se non è presentezinco nella soluzione. Ma il mio era statoun errore straordinariamente fortunato:se ne avessi sputo di più, avrei cessatodi cercare alternative.
A ogni chilometro una possibile solu-zione si affacciava alla mia mente, manessuna appariva adeguata. Poi, quandocominciai a scendere nella AndersonValley, fui colpito da un'idea che soddi-sfaceva il mio senso estetico, ma appari-va anche conveniente: avrei utilizzato lostesso enzima, la DNA-polimerasi, duevolte, prima per eliminare i nucleosidi-trifosfati estranei dal campione e poi perincorporare i ddNTP marcati.
Pensavo che, se nel campione vi fossestata una quantità di nucleotidi sufficien-te a disturbare l'esperimento, questaquantità sarebbe bastata anche a indurrela DNA-polimerasi ad agire. Sottopo-nendo il campione a una specie di rea-zione preliminare simulata, con inneschioligonucleotidici e polimerasi ma senzaddNTP, avrei potuto facilmente elimi-nare dalla miscela tutti i nucleotidi pre-senti incorporandoli negli oligonucleoti-di in fase di allungamento. Quindi, au-mentando la temperatura del campione,avrei potuto separare gli oligonucleotidiallungati dalle molecole bersaglio diDNA. Certamente gli oligonucleotidi al-lungati sarebbero ancora stati presentinel campione: ma poiché nella misceladi reazione le molecole innesco non al-lungate sarebbero state di gran lunga piùnumerose di quelle allungate, i bersaglidi DNA si sarebbero probabilmenteibridati con gli inneschi non allungatinon appena la miscela si fosse raffredda-ta. A questo punto avrei potuto aggiun-gere i ddNTP e ulteriore polimerasi pereseguire il sequenziamento.
Alcuni problemi, però, continuavanoa tormentarmi. Gli oligonucleotidi al-lungati nel corso della reazione simu-lata avrebbero interferito con le suc-cessive reazioni? E che cosa sarebbe suc-cesso se i nucleotidi aggiunti fosserostati parecchi anziché uno o due soltan-to? Quali sarebbero state le conseguenzese l'allungamento fosse stato tale dadar luogo a una sequenza che includesseun sito di legame per l'altra molecolainnesco? Certamente vi sarebbero statedifficoltà...
No, niente di tutto questo! Venni qua-si folgorato dall'improvvisa intuizione: ifilamenti del DNA bersaglio e gli oligo-nucleotidi allungati avrebbero avuto lestesse sequenze di basi. In effetti, la rea-zione simulata avrebbe raddoppiato ilnumero di copie di DNA nel campione!
All'improvviso, mi parve che l'inten-sità del profumo dei fiori diminuisseesponenzialmente.
In altre circostanze, avrei potuto nonriconoscere così rapidamente l'impor-
tanza di questa duplicazione. L'idea diripetere più e più volte un procedimentoavrebbe potuto sembrare insopportabi-le; tuttavia avevo trascorso molto tempoa scrivere programmi per calcolatore emi ero familiarizzato con i cicli iterativi,procedure in cui un'operazione matema-tica viene applicata ripetutamente aiprodotti di precedenti iterazioni. Avevocosì appreso quali fossero le potenzialitàdei processi iterativi di crescita esponen-ziale. La tecnica di duplicazione delDNA che avevo immaginato sarebbestata proprio un processo di questo tipo.
In preda all'eccitazione, cominciai acalcolare mentalmente le potenze didue: 2, 4, 8, 16, 32... Ricordavo vaga-mente che due alla decima potenza eracirca 1000 e che, quindi, due alla vente-sima potenza doveva essere circa un mi-lione. Fermai l'automobile in uno spiaz-zo che guardava sulla Anderson Valley.Dal cassetto portaoggetti estrassi carta ematita: avevo bisogno di controllare imiei calcoli. La mia assonnata compa-gna fu contrariata per la sosta e per laluce improvvisa, ma io le dissi che avevoscoperto qualcosa di fantastico. Perples-sa, preferì immergersi di nuovo nel son-no. Ebbi la conferma che due alla ven-tesima potenza era realmente più di unmilione e mi rimisi in moto.
Dopo aver percorso un altro chilome-tro, mi resi conto che c'era dell'altro aproposito dei prodotti di reazione. Dopoalcuni cicli di allungamento degli inne-schi, di dissociazione dei prodotti allun-gati, di reibridazione di nuovi inneschi edi allungamento di questi ibridi, la lun-ghezza dei filamenti di DNA che si ac-cumulavano esponenzialmente sarebberimasta fissa, perché le estremità di que-sti filamenti sarebbero state nettamen-te definite dalle estremità 5' degli inne-schi oligonucleotidici. Potevo duplicarei frammenti più lunghi del campione ori-ginale di DNA preparando inneschi ingrado di ibridarsi con esso in posizionipiù distanziate l'una dall'altra. I fram-menti sarebbero stati sempre entità bendistinte di lunghezza specifica.
Fermai di nuovo l'automobile e co-minciai a disegnare file di molecole diDNA che si ibridavano e si allungavano,i prodotti di un ciclo che diventavano glistampi per il ciclo successivo in una rea-zione a catena... . Soddisfatto procedettifino a casa, senza ulteriori soste. Nonriuscii quasi a dormire quella notte, conle bombe «desossiribonucleari» che miesplodevano nel cervello.
Ma al mattino ero davvero troppostanco per non pensare che qual-
cuno, in qualche parte del mondo, do-veva già aver sperimentato quest'idea.Migliaia di ricercatori avevano allunga-to, per varie ragioni, singoli oligonucleo-tidi ricorrendo alle polimerasi: sicura-mente qualcuno doveva aver notato lapossibilità di una reazione a catena cata-
lizzata da questo enzima. Se la cosa aves-se funzionato, però, ne avrei certamentesentito parlare: i ricercatori se ne sareb-bero serviti senza dubbio per amplifica-re, o moltiplicare, frammenti di DNA.
Di ritorno alla Cetus il lunedì succes-sivo, chiesi a uno dei bibliotecari. Geor-ge McGregor, di farmi una ricerca bi-bliografica sulla DNA-polimerasi. Nonemerse nulla che avesse attinenza conl'amplificazione. Nelle settimane succes-sive esposi la mia idea a chiunque fossedisposto ad ascoltarmi. Nessuno avevasentito dire che ci fossero stati almenodei tentativi in questo senso; nessuno ve-deva buone ragioni per cui non avrebbedovuto funzionare; nessuno, però, di-
mostrò un particolare entusiasmo per lamia idea. In passato, i colleghi pensava-no generalmente che le mie idee sulDNA fossero campate per aria e talvol-ta, dopo alcuni giorni, finivo per trovar-mi d'accordo anch'io. Ma questa voltasapevo di avere in mano qualcosa.
Anni addietro, prima che nascesserole biotecnologie, il nostro edificio allaCetus era appartenuto alla Shell Deve-lopment Company. E il nostro laborato-rio, in particolare, dalle cui finestre po-steriori si gode di un'ampia vista sullecolline di Berkeley, aveva prodotto la«striscia insetticida». Non potei fare ameno di immaginare che anche la rea-zione a catena della polimerasi, un gior-
no. avrebbe potuto fare altrettanta stra-da di quella striscia di plastica gialla dal-l'odore caratteristico.
Passarono mesi prima che fossi prontoper il primo esperimento in cui avrei do-vuto verificare se la reazione a catenadella polimerasi funzionava. Non potevofare altro che basarmi sull'intuito perstabilire quali soluzioni tampone dovevoutilizzare e quali dovevano essere le con-centrazioni relative e assolute dei rea-genti, quanto dovevo riscaldare e raf-freddare le miscele di reazione, quantoa lungo bisognava far procedere le rea-zioni e così via. Alcuni dei primi articolidi Kornberg sulla DNA-polimerasi mifurono di grande aiuto. Per eseguire l'e-
5
5
5,
5'A A A
T T T
3'
5'
A A A C
5'
A A A
/N
T C T T
3'
5'
AGGIUNTA DI POLIMERASIE DI ddNTP MARCATI
v
36 LE SCIENZE n. 262, giugno 1990
LE SCIENZE n. 262, giugno 1990 37
L'apparecchio che esegue la reazione a catena della polimerasi viene caricato con i campio-ni di DNA. Questi apparecchi stanno diffondendosi rapidamente in numerosi laboratori.
"-n
ALL'INFINITO
La reazione a catena della polimerasi è un processo ciclico nel qualeil numero di copie del DNA bersaglio raddoppia a ciascun ciclo. Inognuno dei segmenti in esame di DNA a doppia elica i due filamen-ti vengono separati mediante riscaldamento e in seguito raffreddati
per consentire agli inneschi di legarsi a essi. Nella fase successiva leDNA-polimerasi provocano l'allungamento degli inneschi, aggiun-gendo nucleotidi alle catene. In questo modo si possono produrrenumerosi duplicati dei filamenti della molecola di DNA bersaglio.
SEPARAZIONE DEI FILAMENTI DEL DNA BERSAGLIOE ATTACCO DEGLI INNESCHI
ALLUNGAMENTO DEGLI INNESCHIPER OTTENERE COPIE DEL DNA BERSAGLIO
=1-21112:110
v111111111
CICLO 2 \
Ov—ICC= c"—=1.1.1i11:====1 I-
III 1111
o11111111101
crTIMMI
11.111.1.illIr=1:=1EEE1110----
crTTT111
91111111111111111111111111111111110
lll IlfllIIIIIIIlIIlIIIII1IIlIIIII!L
o
O
O
o
CICLO 3
o37771771CICLO i
sperimento scelsi un frammento di pla-smide costituito da 25 coppie di basi e,come inneschi, due oligonucleotidi lun-ghi rispettivamente 11 e 13 nucleotidi.
Quando tutto fu pronto, effettuai ilmio tipo di esperimento preferito, espe-rimento che si eseguiva con una sola pro-vetta e dava una risposta univoca: sì ono. La reazione a catena della polimerasiavrebbe amplificato la sequenza di DNA
che avevo scelto? La risposta fu sì.Uscendo dal laboratorio la sera tardi,
notai che Albert Halluin, il legale che sioccupava di brevetti per la Cetus, eraancora nel suo ufficio. Gli riferii che ave-vo inventato qualcosa e gli descrissi lareazione a catena della polimerasi. Fu ilprimo, delle forse cento persone a cuil'avevo spiegata, a concordare sul fattoche si trattava di qualcosa di significa-
tivo. Volle vedere subito l'autoradio-gramma ancora umido che mostrava idati sperimentali.
Alcune persone non sono favorevol-mente colpite da esperimenti eseguiti inuna sola provetta, ma Al non mostrò al-cuno scetticismo. In fin dei conti, chi sioccupa di brevetti ha un legittimo inte-resse per le invenzioni. Al aveva seguitola mia spiegazione del processo nel suo
ufficio ed era d'accordo che si trattassedi qualcosa di significativo. In laborato-rio, mostrò addirittura una certa eccita-zione e mi suggerì di cominciare a lavo-rare sull'esperimento e di stenderne unadescrizione per ottenere il brevetto. Nellasciarmi, si congratulò con me.
Mei mesi successivi continuai a studia-1 3 re e a perfezionare la reazione a ca-tena della polimerasi con l'aiuto di FredA. Faloona, un giovane mago della ma-tematica che avevo conosciuto tramitemia figlia. Fred mi aveva aiutato nel mioprimo esperimento di reazione a catenasottoponendo la miscela di DNA a ciclidi reazioni: era stata la sua prima espe-rienza nel campo della biochimica e per-tanto la sera celebrammo insieme, conalcune birre, il successo ottenuto.
In seguito confermammo che la rea-zione a catena della polimerasi potevaoperare su frammenti sempre più grandidi DNA ottenuto dai plasmidi. Alla fineottenemmo un poco di DNA umano dallaboratorio di Erlich e dimostrammo cheun frammento di un gene in copia singo-la poteva venire amplificato.
Oggi molte delle iniziali difficoltà oinefficienze della reazione a catena dellapolimerasi sono state superate e sono en-trati nell'uso diversi protocolli legger-mente differenti l'uno dall'altro. Gene-ralmente raccomando che i campioni diDNA siano sottoposti a cicli di reazio-ni a temperature comprese tra 98 gradiCelsius (appena al di sotto del punto diebollizione dell'acqua) e 60 gradi Cel-sius circa. I cicli possono durare anchesolo uno o due minuti e in ognuno di essiil numero delle molecole bersaglio diDNA raddoppia. Gli inneschi hanno ingenerale una lunghezza compresa tra 20e 30 nucleotidi. Uno dei miglioramentipiù importanti del processo è stato l'im-piego di una particolare DNA-polime-rasi, originariamente estratta dal bat-terio Thermus aquaticus che vive nellesorgenti calde. La polimerasi che aveva-mo usato nei primi esperimenti venivafacilmente distrutta dal calore; di con-seguenza in ogni ciclo di reazione occor-reva aggiungerne di nuova. La DNA-po-limerasi di Thermus aquaticus, inve-ce, è stabile e attiva a temperature ele-vate, il che significa che basta introdur-la all'inizio della reazione. Questa poli-merasi resistente alle alte temperatureviene oggi prodotta in modo convenien-te dai batteri mediante manipolazionegenetica.
L'amplificazione virtualmente illimi-tata del DNA per mezzo della reazionea catena della polimerasi era una no-vità di portata troppo grande per es-sere accettata facilmente. Nessuno erapreparato di fronte a un processo cheoffriva tutto il DNA che si poteva desi-derare. La reazione sembrava ovvia aFred e a me perché era il nostro giocat-tolo, ma la maggior parte dei nostri col-leghi ebbe bisogno di tempo per abituar-si all'idea.
Nella primavera del 1984, mentre sta-vo lavorando sul brevetto, presen-
tai all'incontro scientifico annuale orga-nizzato dalla Cetus un poster che descri-veva la reazione a catena della polime-rasi . Questi incontri erano sempre inte-ressanti perché la Cetus aveva consulentiscientifici di prim'ordine e io speravodi riuscire a parlare con loro della miainvenzione.
Tuttavia nessuno sembrava interes-sarsi al poster, il che mi rendeva semprepiù ansioso; i partecipanti lo guardavanoe passavano oltre. Alla fine, notai lì vi-cino Joshua Lederberg, rettore dellaRockefeller University, e lo costrinsi aguardare i miei risultati. Osservò il po-ster con attenzione, poi girò l'enormetesta, una vera testa da premio Nobel,quella testa che, nel lontano 1946, avevadedotto che i batteri potevano avere rap-porti sessuali, e chiese: «Funziona?».Sembrava divertito.
Con piacere confermai che era pro-prio così e parlammo a lungo. A un certopunto ricordò che una ventina di anniprima, dopo che Kornberg aveva sco-perto la DNA-polimerasi, entrambi ave-vano preso in considerazione l'idea chel'enzima potesse in qualche modo essereutilizzato per produrre grandi quantitàdi DNA. Tuttavia non erano riusciti aescogitare un procedimento realizzabile.Gli ricordai che gli oligonucleotidi nonerano facilmente disponibili a quell'e-poca e che, inoltre, mancavano quasi to-talmente le informazioni sulle sequenzedel DNA.
Guardò di nuovo il poster con un'e-spressione che ormai mi è diventata fa-miliare. Penso che Josh, dopo aver com-preso l'assoluta semplicità della reazionea catena della polimerasi, sia stato forseil primo a provare quella che ora è la
reazione quasi universale degli studiosidi biologia molecolare e degli altri ricer-catori che lavorano sul DNA: «Perchénon l'ho pensata io?». E nessuno sa ve-ramente perché; io sicuramente non loso. Mi sono semplicemente imbattuto inessa, una sera.
BIBLIOGRAFIA
MULLIS KARY, FALOONA FRED,SCHARF STEPHEN e altri, SpeaficEnzymatic Amphfication of DNA In Vi-tro: The Polymerase Chain Reaction in«Cold Spring Harbor Symposia onQuantitative Biology», 51, n. 1, 1986.
MULLIS KARY B. e FALOONA FRED A.,Specific Synthesis of DNA In Vitro via APolymerase-Catalyzed Chain Reactionin «Methods in Enzymology», 155, par-te F, pp. 335-350, 1987.
JEFFREYS ALEC J., WILSON VICTORIA,NEUMANN RITA e KEYTE JOHN, Amplifi-cation of Human Minisatellites by thePolymerase Chain Reaction: TowardsDNA Fingerprinting of Single Cells in«Nucleic Acids Research» 16,pp. 10953-10971, 1988.
INNIS M. A., MYAMBO K. B., GELFAND D.H. e BROW MARY ANN D., DNA Sequen-cing with Thermus aquaticus DNAPolymerase and Direct Sequencing ofPolymerase Chain Reaction-AmplifiedDNA in «Proceedings of the NationalAcademy of Sciences», 85, n. 24, dicem-bre 1988.
WHITE T. J., ARNHEIM NORMAN ed ER-LICH H. A., The Polymerase Chain Reac-tion in «Trends in Genetics», 5, n. 6, giu-gno 1989.
38 LE SCIENZE n. 262, giugno 1990 LE SCIENZE n. 262, giugno 1990 39






















![Corso di Microbiologia 7 virus.ppt [Read-Only]users.unimi.it/dmora/materiali/BM2012/Virus-Fortina-2015.pdf · meccanismi della cellula ospite per produrre i propri componenti (mRNA](https://static.fdocumenti.com/doc/165x107/5c69a64309d3f2e4258d2e68/corso-di-microbiologia-7-virusppt-read-onlyusersunimiitdmoramaterialibm2012virus-fortina-2015pdf.jpg)