La cellula procariota come modello di studio e...
Transcript of La cellula procariota come modello di studio e...

La cellula procariota come modello di studio e applicazione
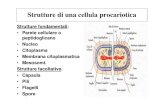
La cellula procariota ha una struttura semplice, non contiene organelli specializzati, ma
una membrana citoplasmatica dove risiedono molte funzioni (trasporto dei metaboliti e nutrienti, è sede della respirazione per i batteri aerobi, è sede della fotosintesi per batteri fotosintetici). Non
possiede un nucleo, ma una regione nucleare, in cui è compattato il genoma cellulare. Il genoma, a differenza della cellula eucariota, è costituito da una unica molecola di DNA cromosomale ( la cellula è dunque aploide). La presenza di una sola molecola, a differenza di 2n cromosomi presenti nelle altre
cellule, non deve trarre però in inganno: in quest’unica molecola sono contenute tutte le informazioni genetiche della cellula, che sono tantissime, infatti la molecola contiene più di 1 milione di basi nucleotidiche ( i costituenti del DNA), ed è talmente lunga che può essere contenuta all’interno della
cellula solo in una forma chiusa, circolare e superavvolta, a formare un gomitolo compatto. Il riavvolgimento e disavvolgimento del DNA è una operazione complessa che deve essere regolata in modo specifico da numerosi enzimi specializzati; solo attraverso un avvolgimento/disavvolgimento
guidato è possibile infatti lo svolgimento delle funzioni vitali della cellula. Vedremo poi come molti batteri contengano del DNA extracromosomale, acquisito in particolari condizioni, che è costituito da una o più molecole di DNA circolare covalentemente chiuso di piccole
dimensioni, che ha la caratteristica di replicarsi ed esprimersi in modo indipendente dal DNA cromosomale della cellula ospitante; tali molecole possono codificare per specifiche proprietà
(resistenza ad antibiotici, enzimi detossificanti, endodesossiribonucleasi di restrizione, enzimi in grado di degradare composto organici particolari, quali toluene, benzene, idrocarburi) che possono essere sfruttate dalla cellula ospitante in particolari condizioni.
La semplicità strutturale di una cellula procariota non deve quindi trarci in inganno, anzi, proprio in ragione della sua semplicità tale cellula nel tempo (in termini filogenetici) ha saputo adattarsi agli habitat più diversi (ricordiamo gli archeobatteri estremofili) e questo spiega perché dei 3 domini della
vita, 2 siano rappresentati da organismi a cellula procariota (eubatteri e archeobatteri).
Ma quali sono le funzioni vitali di una cellula? In primo luogo adattarsi e sopravvivere in un dato habitat: e questo comporta esprimere quelle specifiche caratteristiche fenotipiche che consentono alla cellula di utilizzare un dato substrato, o adattarsi ad un cambiamento ambientale. In secondo luogo riprodursi,
affinchè la propria progenie sia in grado di colonizzare un dato habitat: e questo comporta la costituzione di nuove cellule figlie dotate dello stesso patrimonio genetico della cellula madre. In terzo luogo evolversi, là dove le condizioni ambientali sono tali da ridurre drasticamente le possibilità di
sopravvivenza: e questo comporta in particolari condizioni, la capacità di compiere dei processi di ricombinazione genetica, allo scopo di acquisire qualche caratteristica fenotipica utile (ricordiamo che i batteri non sono in grado di compiere una riproduzione sessuale).
Tutto questo comporta lo svolgimento di tre fondamentali processi: la REPLICAZIONE del DNA, la sua
TRASCRIZIONE in RNA messaggero e la successiva TRADUZIONE in fenotipo, a livello dei ribosomi cellulari, con il coinvolgimento dell’RNA ribosomale (contenuto nei ribosomi) e dell’RNA transfer.

La struttura dell’acido desossiribonucleico (lo scheletro, le coppie di basi, la stabilizzazione, l’avvolgimento)
Come è noto, il DNA nella sua forma nativa, è composto da due filamenti avvolti a doppia elica: in ciascun filamento un gruppo fosfato lega la posizione 5’ di una molecola di zucchero alla posizione 3’
della molecola successiva; ad ogni molecola di zucchero è poi legata una diversa base azotata.
Il DNA possiede 4 basi azotate diverse, 2 pirimidine, la timina T e la citosina C e 2 purine, la guanina G e la adenina A. Poiché le purine sono più grandi delle pirimidine, in una doppia elica regolare, la presenza di una pirimidina in un filamento è sempre accoppiata alla presenza di una purina nell’altro. Inoltre, la
regolarità della struttura a doppia elica richiede la formazione di specifici legami idrogeno fra le basi, in modo tale che esse possano incastrarsi correttamente, perciò una A si contrappone sempre ad una T ( con 2 legami idrogeno) ed una G sempre ad una C ( con 3 legami idrogeno). Ci si riferisce a questi
accoppiamenti fra basi con il termine di complementarietà, e quindi ad un filamento come il complementare dell’altro
I due filamenti inoltre sono orientati in direzione opposta, uno 5’ 3’, l’altro 3’ 5’. Dal momento che i due filamenti sono
complementari, l’informazione contenuta in un filamento può essere dedotta dal filamento complementare, quindi per convenzione una sequenza di DNA a doppio filamento si
rappresenta solo scrivendo la sequenza di uno dei due filamenti in direzione 5’ 3’. La struttura del DNA possiede quindi una carica netta negativa a causa dei gruppi fosfato, che causa una
repulsione elettrostatica che tende a far allontanare i due filamenti: la stabilità della molecola è conferita proprio dai legami idrogeno tra le basi complementari dei due filamenti a da
interazioni idrofobiche tra le basi stesse.
La sintesi del DNA avviene mediante allungamento dalla estremità 3’‐OH con la formazione di un legame fosfodiesterico tra il 3’‐OH e un nuovo dNTP (desossiribonucleotide trifosfato = base
nucleica= zucchero‐fosfato‐base azotata).
Il DNA a doppio filamento nel suo stato rilassato è presente normalmente come doppia elica destrogira con 10 coppie di basi per giro dell’elica, nota come forma B del DNA. Le interazioni idrofobiche tra le basi consecutive sullo stesso filamento contribuiscono all’avvolgimento dell’elica, in quanto si
determina, per l’avvicinamento delle basi, una più efficace esclusione delle molecole di acqua dall’interazione con le basi idrofobiche. Possono esistere altre forme di doppia elica e conformazioni di ordine superiore. La doppia elica è a sua volta ripiegata su stesa, fenomeno noto come
superavvolgimento o supercoling, e l’avvolgimento della doppia elica e il superavvolgimento sono correlati: una variazione dell’avvolgimento determina un cambiamento nel livello di superavvolgimento e viceversa.
La replicazione del DNA. I due filamenti del DNA fungono entrambi da stampo per la sintesi di
nuovi filamenti. E’ quindi una replicazione semiconservativa, dove i due duplex generati possiedono un filamento parentale e un filamento neosintetizzato. Affinchè ciò avvenga è necessario che il doppio
filamento nativo superavvolto si apra in modo da permettere l’azione di specifici enzimi in grado di

iniziare il processo di sintesi. Come è ovvio immaginare, questa “apertura” dei due filamenti, o
denaturazione, non può avvenire lungo tutta la molecola contemporaneamente. Allora, in un punto specifico del cromosoma, noto come origine di replicazione, si ha la formazione di una forcella, cioè una piccola parte del DNA si apre permettendo che inizi il processo. Tale separazione dei filamenti avviene
impiegando energia e l’azione di specifici enzimi. In primo luogo agiscono delle DNA elicasi che catalizzano la separazione dei filamenti, poi delle proteine che legano il DNA a singolo filamento (SSB= single strand binding) impedendo il riappaiamento. Inoltre, poiché non appena il DNA si svolge alla forca
di replicazione, si ha in qualche altra regione un avvolgimento compensatorio che crea tensione alla struttura in quanto chiusa, devono agire altre enzimi specifici, le DNA girasi, della classe delle topoisomerasi, che introducono rotture a singolo o a doppio filamento sul DNA, permettendo un
cambiamento di forma o topologia. A questo punto inizia il lavoro della DNA polimerasi, l’enzima chiave della sintesi del DNA. La cellula possiede almeno tre differenti tipi di DNA polimerasi, e una di queste, nota come Pol III sembra
direttamente interessata al processo. Come tutte le polimerasi, essa possiede oltre che attività polimerasica anche attività esonucleasica in grado di rimuovere nucleotidi non corretti non appena vengono incorporati permettendo la correzione delle bozze. E’ stato calcolato che grazie all’azione
specifica di tale enzima, la forca di replicazione in E. coli, ad esempio, si muove alla velocità di 1000 nucleotidi /secondo. Ma perché ciò avvenga è necessario che l’enzima agisca in direzione 5’ 3’ e che inizi la sintesi a partire da DNA a doppio filamento. Per questo motivo,prima dell’inizio del processo di
replicazione, delle primasi, sintetizzano dei brevi frammenti di RNA complementari alle sequenze del DNA nella forcella della replicazione, noti come inneschi, a partire dai quali inizia l’azione della polimerasi.
Sul filamento guida in direzione 5’ 3’ la sintesi a partire dall’innesco avviene in modo continuo, ma l’altro filamento complementare (il filamento lento o copia) dovrà essere sintetizzato in modo discontinuo, per rispettare la direzione 5’ 3’ di attività dell’enzima. E’ il noto modello della replicazione
semidiscontinua di Okasaki, da cui prendono il nome i frammenti di DNA di nuova sintesi sul filamento copia. Al termine del processo, una DNA ligasi, congiungerà i diversi frammenti.
Poiché la replicazione è bidirezionale, dall’origine di replicazione nascono due forche che si muovono in direzioni opposte intorno al DNA circolare finchè non si incontrano nella parte opposta che corrisponde
alla terminazione della replicazione. A questo punto è necessario dividere le due molecole neo sintetizzate, prima che avvenga la divisione cellulare. Esse infatti sono concatenate e è necessario l’intervento di una topoisomerasi in grado di creare una rottura di un doppio filamento.
La struttura dell’acido ribonucleico (lo scheletro, le basi, i tipi di RNA e la loro conformazione)
Chimicamente l’RNA è molto simile al DNA: l’unica differenza è che il suo scheletro contiene il ribosio anziché il desossiribosio. Questa differenza, che sembra piccola, in realtà ha un notevole effetto sulla
stabilità: infatti mentre il DNA in condizioni di pH elevato si denatura ma in modo reversibile, l’RNA viene degradato in modo irreversibile. Un ‘altra differenza è la presenza di uracile, che sostituisce la timina. Infine l’RNA si presenta sempre a singolo filamento, anche se in grado di formare strutture a
doppio filamento.
L’RNA è presente in tre differenti tipi,ognuno con un compito specifico:

l’RNA messaggero, è un filamento lineare di sequenze nucleotidiche che sono state copiate, attraverso
il processo della trascrizione, dal DNA. Il compito di tale molecola è dunque di portare il messaggio del’informazione genica dal DNA cromosomale a livello dei ribosomi, dove verrà tradotto in proteine. La sua lunghezza varia a seconda delle necessità di sintesi della cellula e dalla organizzazione delle
informazioni genetiche lungo il DNA. Nella cellula possono formarsi contemporaneamente più mRNA differenti.
L'RNA ribosomale è la tipologia più abbondante di RNA presente nella cellula. E’ il componente principale (circa i
due terzi) dei ribosomi. Il ribosoma è una struttura che si auto‐assembla in due subunità ripiegate (la subunità maggiore e la subunità minore) costituite dall'RNA
ribosomiale, in presenza di 70‐80 proteine ribosomiali, che si trovano all'esterno oppure nelle cavità presenti all'interno dell'rRNA ripiegato.La funzione dell'rRNA è
fornire un meccanismo per decodificare l'mRNA in amminoacidi durante la sintesi proteica, come vedremo in seguito.
Alcune caratteristiche dell'RNA ribosomiale sono importanti per la medicina e per lo studio dell'evoluzione: • L'rRNA è il bersaglio di molti antibiotici: cloramfenicolo, eritromicina, micrococcina, streptomicina;
• L'rRNA è il componente più conservato (che ha subito meno variazioni nel corso dell'evoluzione) in tutte le cellule. Per questa ragione, i geni che codificano per l'rRNA vengono sequenziati per identificare il gruppo tassonomico di un organismo, per riconoscere i gruppi correlati e stimare il
tasso di divergenza tra le varie specie. Nei Bacteria, negli Archaea, nei mitocondri e nei cloroplasti, la subunità minore contiene l'rRNA 16S (16 Svedberg = coefficiente di sedimentazione), mentre la subunità maggiore ne contiene due tipi: il 5S ed il
23S. Il ribosoma completo attivo ha un coefficiente di 70S (50S + 30S). Al contrario negli Eukarya la subunità ribosomiale minore contiene l'rRNA 18S, mentre la subunità maggiore contiene il 5S, il 5,8S ed il 28S; il coefficiente di sedimentazione del ribosoma di una cellula eucariota è 80S (60S + 40S).
L'RNA transfer (o RNA di trasporto), abbreviato in tRNA, è una corta catena nucleotidica (di 74‐93 nucleotidi) che lega e trasferisce un amminoacido specifico ad
una catena polipeptidica in crescita a livello dei ribosomi durante il processo della traduzione del messaggio genetico in proteine. Il tRNA ha un sito di attacco per l'amminoacido ed
una regione con tre basi (nucleotidi), chiamata anticodone, che riconosce il corrispondente codone a tre basi dell'mRNA attraverso l'appaiamento di basi complementari. Il tRNA ha
una struttura primaria (l'ordine dei nucleotidi da 5' a 3'), una struttura secondaria (chiamata comunemente struttura a trifoglio), ed una struttura terziaria

Il flusso dell’informazione genica (lo schema di lettura del codice genetico, la
degenerazione del codice, la definizione di gene ).
Il genoma è scritto in un linguaggio composto da parole di tre lettere, le triplette o codoni, ognuna
indica un amminoacido. I conti però non tornano: con 4 diverse lettere si possono formare ben 64 parole diverse, mentre gli aminoacidi sono solo venti. Proprio come nella nostra lingua, anche il
genoma ha dei sinonimi: alcune triplette indicano lo stesso amminoacido. Osservando la tabella possiamo fare alcune
importanti considerazioni:
• Tutti i 64 possibili codoni hanno una
funzione specifica: 61 di essi, detti codoni di senso, specificano i diversi amminoacidi, i 3 rimanenti, detti codoni
non senso, o codoni di stop, sono impiegati come segnali di arresto della traduzione
• I 20 amminoacidi possono essere codificati da 61 diversi codoni. Questa
caratteristica è detta degenerazione del codice genetico. Nella maggior parte dei casi i codoni sinonimi differiscono solo per la terza base all’estremità 3’, che può variare senza modificare il significato del codone
• Poiché i tRNA sono gli adattatori dei codoni, dovremmo aspettarci 61 diversi tRNA: in realtà il numero è inferiore e sebbene variabile da specie a specie, non raggiunge mai il numero dei
codoni possibili. Questo comportamento è noto con il termine vacillamento della terza base: in realtà l’appaiamento del codone con l’anticodone è tale da consentire la presenza di una base diversa in terza posizione
• In termini evolutivi, la degenerazione del codice consente alla cellula di far fronte a possibili modificazioni/mutazioni ch potrebbero cambiare gli amminoacidi fondamentali di una proteina,
oppure generare codoni di stop.
• Il codone di inizio nei procarioti è solitamente ATG (più raramente GTG o TTG) a cui corrisponde
un tRNA che trasporta una N‐formil‐metionina che è il primo amminoacido incorporato in una proteina e che viene frequentemente rimosso dalla proteina durante la sua maturazione. Quando i codoni ATG, GTG o TTG si ritrovano all’interno di una sequenza nucleotidica codificano
normalmente per una metionina, valina e leucina rispettivamente.
Possiamo ora definire un GENE come una sequenza nucleotidica continua, presente in una data regione del cromosoma, dotata di un codone di inizio, un certo numero di codoni interni a cui si aggiunge il
codone di stop. Questa sequenza limitata da un inizio ed una fine, se codifica per un prodotto del fenotipo, viene chiamata GENE. Un tempo era noto il concetto “un gene, una proteina”. Con il progredire degli studi genetici e biochimici si scoprì che molte proteine sono costituite da diversi

polipeptidi che possono essere codificati da distinte regioni nucleotidiche. Di conseguenza la definizione
di gene si è estesa per identificare una regione i DNA che contiene l’informazione per la sintesi di un polipeptide. Tuttavia anche questa definizione è limitata perché, come vedremo, ci sono sul cromosoma regioni di DNA che sebbene non codifichino per proteine o polipeptidi, sono importanti per la vitalità
cellulare o per la regolazione genica. In termini molecolari si utilizza anche il termine ORF (open reading frame) per indicare una regione codificante, della quale ancora non si conosce la funzione a livello del fenotipo. Quanto può essere lungo un gene, in termini di numero di basi nucleotidiche? Possiamo
assumere che il peso molecolare (PM) medio di una proteina sia di circa 40.000 Dalton; se consideriamo che un amminoacido in media pesa 110 D, allora la proteina è costituita da circa 364 amminoacidi. Poiché ciascun amminoacido richiede una tripletta di basi nucleotidiche, possiamo dedurre che una
proteina sia codificata da un gene di circa 1092 coppie di basi (bp= base pair)
La trascrizione. Quando la cellula ha bisogno di un determinato prodotto, codificato da uno
specifico gene, inizia il processo della trascrizione del gene in mRNA. Tale evento è catalizzato
dall’enzima RNA polimerasi. L’enzima riconosce una regione specifica a monte del gene da
trascrivere, denominata sito promotore, si lega ad esso in modo specifico e causa una denaturazione localizzata della doppia elica del DNA, che consente poi all’enzima di iniziare la copiatura di uno dei
filamenti del DNA, aggiungendo in direzione 5’ 3’ i ribonucleotidi trifosfato presenti nel pool cellulare. Facendo ciò si muove lungo il filamento stampo del DNA estendendo la denaturazione, mentre a monte il DNA si riavvolge a doppia elica. Il segnale che il gene è stato trascritto deriva dalla presenza, a valle del
gene in questione, di una altra regione nucleotidica specifica, nota come sito terminatore, il cui compito è di indebolire l’associazione tra l’RNA sintetizzato e lo stampo di DNA. In questo modo l’RNA polimerasi si dissocia dal DNA e dall’mRNA e ha termine la trascrizione.
La RNA polimerasi è costituito da diverse subunità (β’, β, σ, σ2, ω); tra queste la subunità σ è deputata al riconoscimento del sito di legame per l’RNA polimerasi sul DNA, nella regione che abbiamo chiamato
sito promotore: esso consta di una sequenza nucleotidica di circa 6 basi localizzata a circa 10 bp a monte dell’inizio della trascrizione, nota come Pribnow (dal nome dello scopritore) box, o più semplicemente box ‐10, e di una altra sequenza a circa ‐35 bp dal sito di inizio della trascrizione, nota come box‐35.
Dall’esame di numerose sequenze del promotore, è emerso che in molti casi esiste una sequenza tipica per ciascun box, la cosiddetta sequenza consensus:
box10 TAtAaT (chiamata anche tata box‐le lettere maiuscole indicano basi più conservate,
mentre quelle minuscole hanno meno probabilità di comparire in quella data
posizione)
box35 TTGACa
Quando andremo ad analizzare le diverse sequenze nucleotidiche ci accorgeremo che raramente troveremo sequenze promotrici che corrispondano alle sequenze consensus, comunque sequenze simili tendono ad essere conservate e a ricorrere in promotori forti, cioè regioni che consentono una
trascrizione attiva e veloce. Infatti mutazioni che cambiano tali sequenze danno luogo ad una riduzione della trascrizione dei geni adiacenti (promotori deboli). Anche la distanza dei siti promotori dall’inizio della trascrizione può variare e influenzare così il processo della trascrizione. Sono questi alcuni

meccanismi con i quali la cellula regola, insieme ad altri processi che vedremo in seguito , l’espressione
genica in funzione delle necessità del momento. La terminazione avviene ad opera di una sequenza specifica, posta a valle del gene, che è una sequenza
ripetuta e invertita, che consente la formazione, nell’RNA trascritto, di una ansa, nota come struttura a forcina, che destabilizza l’ibrido RNA‐DNA.
La traduzione. La traduzione è il processo attraverso il quale i ribosomi leggono il messaggio
genetico contenuto nell’RNA messaggero e producono un prodotto proteico in base alle istruzioni del
messaggero. Prima che questo abbia inizio, la cellula prepara una scorta di amminoacil‐tRNA (tRNA con i corrispondenti amminoacidi legati) (processo di caricamento del tRNA), grazie all’azione di una amminoacil‐tRNA sintetasi. Esistono 20 sintetasi nella cellula, ognuna specifica per ciascun
amminoacido.
L’inizio della traduzione è preceduto anche dalla dissociazione delle due subunità dei ribosomi: la subunità 30S legherà l’mRNA e l’anticodone del tRNA, la subunità 50 S legherà l’estremità del tRNA caricata con l’amminoacido; essa possiede inoltre una attività peptidil trasferasica che consente la
formazione del legame peptidico tra gli amminoacidi che via via vengono legati. Come già accennato, il codone di inizio nei procarioti è solitamente AUG che codifica per una metionina
formilata. Essa possiede un suo tRNA, tRNAMetf (diverso da quello per la metionina interna alla catena
polipeptidica, tRNAMetm) che consente la formilazione della metionina dopo il suo legame. Subito a
monte del codone di inizio esiste una breve sequenza, chiamata sequenza Shine‐Dalgarno (dal nome dei
suoi scopritori) che trova complementarietà con una sequenza posta all’estremità 3’ del 16S rRNA. Questa complementarietà dà luogo al legame tra subunità 30S ed il giusto sito di inizio sull’mRNA. La sequenza di Shine‐Dalgardo è ricca in A e G ( AGGAGG‐ GGAGG) e viene chiamata anche Ribosomal
Binding Site (RBS); la sua distanza dal codone di inizio può comunque variare, condizionando l’efficienza della traduzione. Ma come avviene l’allungamento? Sul ribosoma si trova un sito di legame peptidico (sito P) e un sito
amminoacilico (sitoA). Il tRNA per la formil‐metionina è legato al sito P, il secondo codone è nel sito A, a questo si lega il secondo amminoacil‐tRNA, ad opera di un fattore di allungamento (EF‐tu) e una molecola di GTP. L’attività peptidil transferasica della subunità 50S consente la formazione del legame
peptidoco tra fMet‐tRNA e il secondo amminoacil‐tRNA e il trasferimento del dipetidil‐tRNA nel sito A . Nel passaggio successivo, chiamato traslocazione (che prevede la presenza di un fattore di allungamento EF‐G e una molecola di GTP), l’ mRNA con il peptidil‐tRNA formato si muove di un codone in avanti.
Questo comporta il distacco del tRNA deacilato dal sito P, mentre il dipeptidil si sposta dal sito A al sito P; questo fa sì che il codone successivo si trovi ora al sito A, pronto a interagire con il corrispondente amminoacil‐tRNA. Il ciclo di allungamento prosegue fino a quando il ribosoma incontra il codone di stop:
esso viene riconosciuto da specifiche proteine, i fattori di rilascio (RF) che determinano la terminazione della traduzione.

L’organizzazione del DNA e la sua regolazione. Il genoma di un batterio può
contenere più di 3000 geni. I batteri, infatti, hanno una grande versatilità dal punto di vista metabolico e in particolare nell’utilizzazione degli zuccheri come fonte di carbonio. Se la cellula batterica dovesse
sintetizzare simultaneamente tutti gli enzimi coinvolti in tali processi, probabilmente consumerebbe più energia di quanto riuscirebbe a ricavarne. La cellula batterica ha quindi sviluppato dei meccanismi per reprimere tutti i geni che non sono necessari, attivandoli solo nel momento in cui servono.
Alcuni geni sono sempre attivi (accesi) perché i loro prodotti sono sempre richiesti dalla cellula. Altri
invece sono inattivi (spenti) e vengono attivati solo quando richiesto, in particolari situazioni. Per esempio, i geni codificanti gli enzimi della via glicolitica sono sempre attivi (può variare eventualmente il livello di espressione), mentre quelli relativi agli enzimi in grado di utilizzare l’arabinosio sono quasi
sempre spenti e attivati solo nel momento in cui questa insolita fonte di carbonio rappresenta per la cellula l’unica fonte disponibile per la crescita. Questo controllo è indispensabile per la cellula, perché l’espressione genica è un processo molto
dispendioso in termini energetici e la cellula ha bisogno di fare economia per poter continuare a crescere e competere con altri organismi più efficienti. Nel tempo quindi la cellula ha attuato questa strategia evolutiva per organizzare al meglio le informazioni genetiche e per controllare l’espressione
genica. Oltre a regolare l’espressione genica i batteri hanno anche attuato un’altra strategia, raggruppando geni correlati tra loro funzionalmente, in modo da essere regolati insieme. Un gruppo di geni contigui e
controllati in maniera coordinata è definito operone.
Il primo operone ad essere scoperto è l’operone lac di E.coli: esso contiene 3 geni che codificano per gli enzimi che
consentono alla cellula di utilizzare il lattosio: una lattosio permeasi, una β galattosidasi e una galattoside
transacetilasi. I 3 geni sono raggruppati nell’ordine: lacZ (β
galattosidasi), lacY (lattosio permeasi) e lacA (galattoside transacetilasi) e vengono trascritti
contemporaneamente per produrre un unico RNA messaggero (chiamato policistronico‐ cistrone è un sinonimo di gene), a partire da un unico promotore che si trova a monte del primo gene dell’operone. Consideriamo una crescita diauxica, vale a dire in un terreno contenente sia glucosio che lattosio. Le
cellule utilizzano subito il glucosio, per il quale sono già disponibili nel pool citoplasmatico gli enzimi (costitutivi), e solo quando il glucosio è esaurito, attiveranno l’operone lac per produrre gli enzimi necessari alla metabolizzazione del lattosio (inducibili). Come avviene la regolazione ed il controllo
dell’espressione genica? Come molti altri operoni, l’operone lac è soggetto a due tipi di controllo. Il primo è un controllo negativo, che mantiene spento l’operone in assenza di lattosio. Questo avviene
ad opera di un repressore specifico, il repressore lac in questo caso, il prodotto di un gene regolatore lacI, che si lega in un sito specifico, noto come sito operatore, che sta appena a valle del sito promotore, impedendo il giusto legame tra sito promotore ed RNA polimerasi, inibendo così la trascrizione.

In realtà le cellule riescono a mantenere l’operone in uno stato relativamente inattivo, con la
produzione di poche molecole di enzimi per il lattosio. Il repressore è una proteina allosterica: in presenza di un giusto induttore che si lega ad esso si determina una conversione della struttura e il passaggio a una conformazione che ne favorisce la dissociazione dall’operatore, inducendo così
l’operone. In questo caso la presenza di lattosio consente la derepressione dell’operone. Il livello basale di enzimi per il lattosio fa sì che questa fonte di carbonio possa essere in parte metabolizzata ad allo lattosio (legame β 1‐6 anziché β1‐4) che rappresenta l’induttore in grado di legarsi al repressore e
dereprimere l’operone lac.
Nel caso della crescita diauxica si osserva un tipo di controllo positivo dell’operone. Abbiamo detto che le cellule inattivano i geni per il lattosio fino a quando è presente il glucosio: è necessaria la presenza di
una molecola in grado di “sentire” la mancanza di glucosio e di rispondere attivando il promotore lac (controllo positivo). Tale molecola è l’AMP ciclico, la cui concentrazione aumenta al dimunuire dei livelli di glucosio . Per ottenere l’attivazione dell’operone è però necessaria la presenza anche di un’altra
proteina, chiamata proteina attivatrice del catabolita o CAP (detta anche proteina recettrice di AMP ciclico, CRP). Il sito di legame per il complesso CAP‐cAMP (sito di legame per l’attivatore) si trova subito a monte del promotore ed è stato dimostrato che il legame del complesso a tale sito stimola il legame
dell’RNA polimerasi al sito promotore e quindi la trascrizione. Tale processo è anche noto come repressione da catabolita in quanto è stata per lungo tempo attribuita all’influenza di un qualche prodotto derivante dalla metabolizzazione del glucosio, o catabolita.
Un altro operone, di importanza vitale per le cellule è l’operone ribosomale. Esso è caratterizzato dalla
presenza dei geni strutturali necessari per la sintesi dell’rRNA 16S , 23S e 5S, indispensabili per la formazione dei ribosomi attivi. Come vedremo più avanti è uno degli operoni più studiati per valutare in termini filogenetici, le correlazioni esistenti tra i vari microrganismi. In particolare i geni 16S rDNA ed i
geni 23S rDNA sono considerati, in termini evolutivi, i più significativi orologi molecolari: essi sono presenti in tutte le cellule, dove svolgono la stessa funzione ed hanno una sequenza nucleotidica che si è conservata nel tempo.
Altre caratteristiche di questo operone lo rendono particolarmente utile per studiare il grado di
biodiversità microbica inter ed intraspecie, in particolare la presenza, tra il gene 16SrDNA ed il gene 23S

rDNA di una regione nucleotidica che può presentare maggiori variazioni di sequenza e di lunghezza
rispetto ai geni strutturali. Tale regione è nota come Regione Spaziatrice (RS) o Internal Transcribed Spacer (ITS). I vari microrganismi infine, possono possedere, lungo il genoma, più di un operone ribosomale (da 1 a 11 copie): la presenza di multicopie di questo operone è stato correlato alla necessità
cellulare di disporre di più ribosomi attivi (ad esempio cellule con metabolismo energetico respiratorio), per una attiva sintesi proteica). E’ stato osservato che anche all’interno di uno specifico genoma, le varie copie dell’operone ribosomale possono variare leggermente in sequenza e soprattutto per la RS, in
lunghezza.
Ricordiamo infine, che in funzione del tipo di microrganismo gli operoni possono presentare una maggiore complessità rispetto a quelli presi come esempio, sia per quanta riguarda il tipo di regolazione che il numero di geni strutturali in essi contenuti; al riguardo è possibile osservare in operoni complessi
come la cellula possa aver riunito anche geni strutturali che possono in situazioni metaboliche differenti essere accesi singolarmente anche per altre vie metaboliche. In tal caso c’è da aspettarsi che all’interno dell’operone e a monte del gene in questione sia presente un promotore, solitamente più debole del
promotore dell’operone, che consenta la regolazione e l’espressione del singolo gene.

Il DNA plasmidico. In numerose cellule batteriche si possono trovare repliconi autonomi dal
cromosoma, i plasmidi (un replicone è una porzione di DNA provvista della funzione genica di iniziare e completare autonomamente la propria replicazione). Tali plasmidi possono dare crossing‐over sia con il
cromosoma della cellula ospite che tra di loro; nel primo caso si integrano nel cromosoma e sono indicati come episomi, nel secondo formano plasmidi ricombinanti.
Nell'ospite batterico i plasmidi si presentano come molecole circolari superavvolte CCC, che, durante le
manipolazioni sperimentali, possono rilassarsi (forma OC)o linea rizzarsi (forma L) in seguito a rotture a singolo o a doppio filamento. Il loro peso molecolare può variare da poche kb (1‐10) fino a raggiungere dimensioni di 200‐400 kb.
La replicazione dei plasmidi può essere strettamente coordinata con quella del cromosoma (stringente)
ed allora per ogni cellula si avrà un solo plasmide accanto ad un solo cromosoma oppure essa può essere non coordinata con quella del cromosoma (rilassata) ed allora potremo avere per ogni cellula un numero variabile e talvolta molto elevato di molecole plasmidiche sempre in presenza di un solo
cromosoma.
La replicazione autonoma è conferita loro dalla presenza di una origine di replicazione, chiamata ori. Alcuni plasmidi sono caratterizzati da una forma di replicazione bidirezionale, mentre altri presentano
una replicazione monodirezionale che si compie in media in 1/10 del tempo necessario alla replicazione del DNA cromosomico. Molti plasmidi ritrovati in batteri gram positivi si replicano con un meccanismo denominato "rolling circle mechanism". Oltre ad essere essenziale per la replicazione, l’origine di
replicazione controlla il numero di copie, la specificità d’ospite e i gruppi di incompatibilità. Meccanismi molecolari precisi mantengono un numero stabile di copie del plasmide nella cellula ospite e assicurano la loro ripartizione tra le cellule figlie (regione par). La mancanza di questa regione può portare ad una
incorretta ripartizione durante la divisione cellulare fino a perdita del plasmide in condizioni di stress. In seguito a trattamenti diversi un plasmide può essere perso dalla cellula ospite. Questo processo (curing) è dovuto all’inibizione della replicazione del plasmide senza la contemporanea inibizione della
replicazione del cromosoma, per cui in seguito a divisione cellulare il plasmide non sarà ereditato da tutte le cellule. Due plasmidi che non possono coesistere nella stessa cellula –in assenza di pressione selettiva‐ si dicono
incompatibili (appartengono allo stesso gruppo di incompatibilità). Ciò accade quando i due plasmidi contengono la stessa regione par e utilizzano lo stesso meccanismo di replicazione.
Che tipo di informazione è portata a livello plasmidico? Solitamente informazioni non essenziali per la vita della cellula, ma informazioni addizionali che possono aiutare la cellula che li ospita in particolari situazioni: geni codificanti per enzimi coinvolti nella utilizzazione di mono e polisaccaridi, per particolari
endodesossiribonucleasi, per la produzione di sostanze antibatteriche e sostanze tossiche per altre specie batteriche. In base alle informazioni che portano i plasmidi vengono classificati in: plasmidi R: hanno geni codificanti per resitenza ad antibiotici
plasmidi Col: codificano per colicine (sostanze antibatteriche) plasmidi di virulenza: hanno geni codificanti per tossine, adesine, citolisine plasmidi degradativi: permettono di metabolizzare sostanze insolite (geni per la degradazione di
naftalene, benzene, toluene). Esistono tuttavia ancora numerosi plasmidi per i quali non sono note le informazioni geniche: tali
plasmidi vengono definiti criptici.

Alcuni plasmidi denominati plasmidi coniugativi possiedono un set di geni (geni tra, da trasferimento)
che sono in grado di promuovere il loro trasferimento in cellule diverse (trasmissione orizzontale) attraverso un ponte citoplasmatico. Per questo motivo i plasmidi vengono considerati Elementi Genetici Mobili. La presenza di questa regione in un plasmide può avere un’altra importante conseguenza nel
caso in cui il plasmide si integri nel cromosoma. In questo caso il plasmide può mobilizzare il trasferimento di DNA cromosomale da una cellula ad un'altra. Ceppi batterici in grado di trasferire una grande quantità di DNA cromosomale durante il processo di coniugazione, sono detti Hfr (high
frequency of ricombination).

La variabilità genetica. Una caratteristica dei geni è che essi accumulano cambiamenti, o
mutazioni. Il modo più semplice è sostituire una base con un’altra. Questo può portare a convertire un codone in un altro codone e ad ottenere una proteina con un amminoacido diverso. Questo potrebbe
essere l’unica variazione su centinaia di amminoacidi che costituiscono quella proteina, e tale variazione potrebbe essere irrilevante per l’attività della proteina, ma anche scatenare effetti importanti. I geni possono poi andare incontro anche a cambiamenti più significativi, quali delezioni o inserzioni di grandi
tratti di DNA; regioni di DNA possono anche spostarsi da un locus ad un altro, ed in questo caso i cambiamenti saranno più drastici e maggiore sarà la probabilità ch uno o più geni coinvolti sino inattivati.
LE MUTAZIONI ( vedi un testo di Microbiologia)
LA RICOMBINAZIONE GENETICA (vedi un testo di Microbiologia)
LA TRASPOSIZIONE. Nei batteri avviene ad opera di due elementi genetici mobili, le sequenze di
inserzione ed i trasposoni.
Si definiscono sequenze di inserzione (denominate anche sequenze IS, elementi IS o, semplicemente, IS) delle sequenze di DNA capaci di spostarsi autonomamente da un punto all'altro del genoma. Le IS possiedono solo i geni necessari per la trasposizione.
Una sequenza di inserzione è un filamento di DNA a
doppia elica costituito generalmente da circa 8‐900 coppie di basi. Sono costituite da una porzione centrale che contiene dei geni funzionali, che
codificano per gli enzimi necessari alla trasposizione, fiancheggiata da due corte sequenze ripetute invertite, ovvero delle sequenze di basi che non
codificano per nessun enzima, disposte specularmente alle due estremità. Quando una IS traspone rimane sempre una copia nella posizione originaria mentre una nuova copia si inserisce in una nuova posizione del genoma; in altre parole, la trasposizione è sempre replicativa.
Le sequenze IS, quando traspongono, si inseriscono in luoghi casuali del genoma; il sito in cui avviene
l'inserzione di una IS viene chiamato sito bersaglio. Perché sia possibile l'integrazione dell'IS, il sito bersaglio viene rotto (tagliato) dagli enzimi prodotti dalla IS, e successivamente questa si integra nella sequenza. Poiché l'integrazione di una IS è casuale, può avvenire anche all'interno di un gene
funzionante; in questo caso, la corretta espressione del gene può venire alterata o interrotta, causando così una mutazione genetica.
I trasposoni procariotici contengono tutti i geni necessari alla integrazione e alla escissione dal genoma. In più, normalmente contengono anche dei geni aggiuntivi, quali quelli relativi alla resistenza agli
antibiotici, o alla capacità di sintetizzare una particolare molecola. I trasposoni complessi sono costituiti da una parte centrale, che contiene i geni, e da due parti laterali costituite da sequenze di inserzione che presentano sequenze ripetute ed invertite di coppie di basi alle estremità. La trasposizione resa possibile
proprio da tali elementi. Essi infatti producono gli enzimi trasposasi, necessari per lo spostamento. Le

trasposasi inoltre hanno bisogno di riconoscere le ripetizioni invertite degli elementi IS alle estremità del
trasposone, per iniziare il processo.

Gli enzimi di restrizione. Tra gli strumenti più utili alla cellula per riparare danno al DNA e
soprattutto per difendersi da DNA estraneo entrato nella cellula, si annoverano le
endodesossiribonucleasi di restrizione (enzimi di restrizione, endonucleasi, ER). Esse prendono nome dal fatto che impediscono l’invasione da parte di DNA esogeno, tagliandolo in frammenti. Di conseguenza “restringono” lo spettro di possibili ospiti indesiderati. Inoltre questi enzimi tagliano in siti presenti all’interno del DNA esogeno, e non a partire dalle estremità, per questo si chiamano “endo”.
Gli enzimi di restrizione prendono le prime 3 lettere del loro nome dal nome latino del microrganismo
che li produce (esempio Eco, dove E è l’iniziale del genere Escherichia e co l’iniziale della specie coli) e poi viene aggiunta la sigla del particolare ceppo da cui l’ER è stata estratta (esempio EcoR1). Si riconoscono così.
EcoRI da Escherichia coli HindIII da Haemophilus influenzae BamHI da Bacillus amyloliquefaciens Dalla fine degli anni ’60, quando si è scoperta la prima ER in E.coli, sono state ritrovate in numerosi altri
batteri nuove ER o isoschizomeri di ER già note. Vedremo come la possibilità di impiego delle ER sia condizione indispensabile nella biologia molecolare. Infatti, tali enzimi sono capaci di riconoscere sul DNA brevi sequenze bersaglio, di solito palindromiche tagliandole in posizioni specifiche. Una
palindrome è una parola che si legge allo stesso modo sia da destra che da sinistra, e un sito di riconoscimento palindromico è una sequenza in cui il filamento superiore e inferiore, letti in direzione 5'‐3', sono uguali, per es. la sequenza:
5'‐GAATTC‐3‘ 3'‐CTTAAG‐5'
Esistono tre classi di enzimi di restrizione, ma solo gli enzimi di classe II sono caratterizzati da una
elevata specificità di taglio. E la straordinaria importanza degli enzimi restrizione risiede proprio nella loro specificità. Ogni particolare enzima di restrizione, infatti, riconosce una sequenza specifica di basi all’interno di una catena polinucleotidica. La maggior parte degli enzimi più comuni riconoscono da 4 a
6 basi. Il numero di basi riconosciute è di importanza pratica perché determina la frequenza media di taglio e la dimensione media dei frammenti generati. E’ ovvio che un enzima che riconosce una
sequenza di 4 basi taglierà più frequentemente di uno che ne riconosce 6. Un’altra particolarità è la modalità di taglio. Si conoscono ER che tagliano in tre modi diversi, e anche questo fatto, come vedremo in seguito, rappresenta un grande vantaggio in termini applicativi.
Generando estremità piatte (blunt ends) Es. SmaI
5'‐CCC/GGG‐3'
3'‐GGG/CCC‐5'
• Generando estremità coesive (sticky) sporgenti al 5' (5' protuding)
Es. EcoRI
5'‐G/AATTC‐3'

3'‐CTTAA/G‐5'
• Generando estremità coesive (sticky) sporgenti al 3' (3' protuding)
Es. PstI 5'‐CTGCA/G‐3'
3'‐G/ACGTC‐5'
Gli enzimi di restrizione fanno parte dei sistemi di restrizione/modificazione cellulari, capaci di metilare specifiche basi e, contemporaneamente, di tagliare le stesse basi quando non metilate. Con questo sistema la cellula produttrice di ER è capace di degradare DNA esogeno tagliandolo in specifici siti di
riconoscimento. Gli stessi siti presenti sul DNA endogeno, tuttavia, non sono tagliati perché preventivamente metilati dal sistema di restrizione‐metilazione.
Nomenclatura
La nomenclatura degli enzimi di restrizione si basa sul genere e sulla specie del batterio dal quale è stato isolato l’enzima di restrizione: per es., EcoRI da Escherichia coli, HindIII da Haemophilus influentiae etc.

L’estrazione del DNA. Il primo passaggio della maggior parte delle procedure analitiche consiste
nell’estrazione del DNA totale dalle cellule batteriche in studio e nella sua purificazione mediante separazione dagli altri componenti cellulari Esistono numerosi protocolli messi a punto per la lisi
cellulare dei diversi tipi di batteri, per cui analizzeremo solo le fasi generali:
‐raccolta e lavaggio delle cellule cresciute in idoneo terreno colturale (se possibile un terreno non molto ricco in modo da evitare la formazione di strati extraparietali di natura polisaccaridica che interferiscono direttamente sulla resa di lisi e in un secondo tempo con la purificazione del DNA)
‐lisi della parete cellulare mediante enzimi litici appropriati (lisozima solitamente), in combinazione con
EDTA e un surfactante, come il sodio dodici solfato o SDS. L’EDTA complessando i cationi bivalenti, destabilizza la membrana esterna dei Gram negativi, inibisce le DNAsi che altrimenti degraderebbero il DNA, mentre l’SDS aiuta a solubilizzare i lipidi di membrana. Se si lavora in condizioni isotoniche
(mediante aggiunta di saccarosio o glucosio al tampone di lisi) si evita lo scoppio della cellula, indotto dalla rottura dei legami β 1‐4 del peptidoglicano ad opera del lisozima; in questo modo si riduce il danno alla struttura del DNA nativo.
‐l’estratto che si ottiene contiene una miscela complessa di DNA, RNA, proteine, lipidi, carboidrati e
residui parietali. Per purificare il DNA è necessario allontanare l’RNA, attraverso l’azione di una ribonucleasi (RNasi) e le proteine, attraverso l’azione di una proteinasi e di successive estrazioni con una miscela di fenolo/alcool isoamilico o cloroformio. Quando la miscela è agitata vigorosamente,le proteine
vengono denaturate e precipitano all’interfase; quindi gli acidi nucleici possono essere recuperati dalla fase acquosa sovrastante (importante è usare fenolo equilibrato con tampone neutro o alcalino per trattenere il DNA nella fase acquosa)
‐A questo punto si può procedere alla precipitazione selettiva del DNA, con doppio volume di etanolo o
ugual volume di alcool isopropilico, per ottenere la concentrazione del DNA e una ulteriore purificazione da residui di solventi. Il precipitato di DNA, raccolto sul fondo della provetta dopo centrifugazione, può poi essere ridi sciolto in tampone fino ad ottenere una idonea concentrazione.
N.B. Il DNA in soluzione rappresenta il DNA totale della cellula, quindi si estrae sia il DNA cromosomale
che plasmidico. La lisi enzimatica delle cellule è un metodo relativamente blando e questo consente, rispetto ad altri
metodi di estrazione, quali sonicazione o rottura meccanica, di ottenere DNA ad alto peso molecolare; non è possibile però in molti casi mantenere la conformazione nativa delle molecole di DNA: nel caso di DNA plasmidico, solitamente parte delle molecole nella loro conformazione nativa, quindi CCC, si
trasformano in OC, mentre per il DNA cromosomale si ha la conversione e frammentazione in molecole lineari, il cui numero e peso molecolare rispecchiano le precauzioni adottate durante l'estrazione (più delicata è la procedura, meno frammentato risulterà il DNA cromosomale).

Le caratteristiche quantificabili del DNA
Determinazione quantitativa. Il DNA assorbe nello spettro della luce ultravioletta, con un massimo di
assorbanza a 260 nm. E' così possibile quantificare il DNA estratto attraverso una lettura spettrofotometrica alla lunghezza d'onda di 260 nm, ricordandosi la correlazione: 1OD = 50 µg/ml, vale a dire che ad un valore di assorbanza (o densità ottica OD) pari a 1
corrisponde una concentrazione di DNA in soluzione di 50 µg/ml. La lettura spettrofotometrica viene inoltre impiegata per valutare il grado di purificazione della soluzione di DNA, nei confronti di proteine residue (assorbanza a 280 nm) e di RNA e tracce di solvente
(assorbanza a 230nm). Un buon grado di purificazione risulta dal valore del rapporto: OD 260 nm/OD 230nm = 1,8‐2 OD 260 nm/ OD 280 nm = 1,8‐2
Curva di denaturazione termica. Il DNA possiede una caratteristica di estrema importanza per la messa a punto di tanti esperimenti di biologia molecolare: la capacità di riassociare o dissociare, in condizioni
controllate, processo che risulta REVERSIBILE. Tale processo, mediato dalla temperatura, viene seguito attraverso la costruzione di una curva, nota come curva di denaturazione termica del DNA. Come si può osservare il valore di assorbanza aumenta all'aumentare della temperatura: a parità di concentrazione
iniziale, l'aumento di OD è da correlare al fenomeno noto come IPERCROMOCITA' DELLE BASI NUCLEOTIDICHE, vale a dire, quando le basi nucleotidiche non sono legate a formare il doppio filamento, si ha un aumento di OD perchè le singole basi assorbono maggiormente. Questo implica che
quando, per effetto della temperatura, si rompono i legami idrogeno che tengono uniti i due filamenti di DNA, si ha un inizio di dentaurazione (dissociazione) che porta ad un conseguente aumento di OD. Come si può osservare dal grafico, ad una data temperatura si ha la completa dissociazione del DNA, con il
raggiungimento di un valore massimo che poi resta costante, di OD. Da tale curva è possibile estrapolare un valore di temperatura, noto come Tm (melting temperature o temperatura media di denaturazione termica) che rappresenta la temperatura alla quale il 50% del DNA in esame si trova nella sua forma
dissociata. Al di sotto di tale valore il DNA tenderà a rimanere nella sua forma nativa, cioè riassociata, al di sopra del valore di Tm il DNA tenderà a dissociarsi. Il valore di Tm è caratteristico per ciascun DNA e correlato alla quantità di G e C presenti lungo il DNA: infatti più il DNA è ricco di G e C più energia
termica dovrà essere fornita per dissociarlo in quanto più legami idrogeno dovranno essere rotti (ricordiamo i 3 legami H rispetto ai 2 di A e T). Non è però un valore costante nell'ambito dello stesso DNA: può variare infatti in funzione della forza ionica del mezzo in cui il DNA è disciolto. Più alta è la
forza ionica, più il DNA tenderà a riassociarsi per effetto dei controioni che vanno in parte a ridurre la carica negativa netta dei due filamenti, e di conseguenza più alto risulterà il valore di Tm (cioè della temperatura richiesta per ottenre il 50% di dissociazione). Lavorare invece a bassa forza ionica significa
ridurre il valore di Tm e favorire il processo della dissociazione. Visualizzazione del DNA. Il DNA, carico negativamente, e caratterizzato da un peso molecolare elevato, rispetto alle proteine, può venir separato e visualizzato attraverso una gel elettroforesi orizzontale in gel
di agarosio. Il gel viene preparato a concentrazioni variabili da 0,5 al 3%, in funzione del range di PM delle molecole che si vogliono separare e visualizzare (più grandi sono le molecole più bassa deve essere
la concentrazione del gel di agarosio). Sciolto in idoneo tampone, dopo solidificazione in idonei vassoio, nei quali si sono create delle tasche di caricamento, il gel viene posto all'interno della vaschetta di elettroforesi e sommerso dal tampone di corsa. La preparazione di DNA da sottoporre ad analisi viene

addizionata di un indicatore di corsa (solitamente BBF= blu di bromo fenolo addizionato di saccarosio) in
ragione di 1/6 del volume finale e caricata nelle tasche del gel. L'indicatore di corsa ha lo scopo di permettere di visualizzare l'andamento della corsa elettroforetica, il saccarosio in esso contenuto, di aumentare la densità del campione e consentirne l'entrata e l'impaccamento sul fondo della taschina
del gel. La corsa viene fatta avvenire applicando una differenza di potenziale, e procede dal polo negativo a quello positivo. Al termine della corsa elettroforetica, il DNA viene visualizzato con l'ausilio di un transilluminatore, dopo aver messo a contatto il gel con una soluzione di bromuro di etidio, un
agente mutageno ( e quindi tossico anche per l'operatore) che riesce a intercalarsi tra le basi del DNA e quando viene esposto a rdaiazioni UV (come quelle emessa dalla lampada del transilluminatore – 254 nm) emette una fluorescenza di colore rosso, che permette di visualizzare le molecole di DNA come
bande fluorescenti. Il transilluminatore è poi di solito collegato ad un apparecchio fotografico che acquisisce l'immagine e ne permette la stampa.
Con queste poche conoscenze sulle caratteristiche del DNA possiamo già allestire saggi genetico‐molecolari in grado di fornirci importanti indicazioni, come stabilire l'appartenenza di un ceppo ad una data specie microbica, cioè ottenerne la corretta collocazione tassonomica e valutare la presenza di DNA
plasmidico nei ceppi in esame, procedendo ad una loro preliminare caratterizzazione.