Bioluminescenza...dell’organismo stesso (lucciole, batteri o protozoi ecc.) attraverso un...
Transcript of Bioluminescenza...dell’organismo stesso (lucciole, batteri o protozoi ecc.) attraverso un...

1
Bioluminescenza
Prof. Giorgio Sartor
Copyright © 2001-2009 by Giorgio Sartor.
All rights reserved.
Versione 0.2 – may 2009
gs © 2001-2009 ver 0.2 Bioluminescenza - 2 -
Luminescenza
• Emissione della luce in seguito al passaggio dallo stato eccitato allo stato fondamentale di un elettrone;
• Lo stato eccitato viene raggiunto a seguito di una somministrazione di energia.

2
gs © 2001-2009 ver 0.2 Bioluminescenza - 3 -
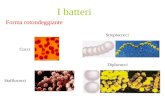
Bioluminescenza• Il fenomeno della bioluminescenza èproprio di alcuni organismi marini:
Microrganismi
Celenterati Teleostei
gs © 2001-2009 ver 0.2 Bioluminescenza - 4 -
Bioluminescenza• … e terrestri:
InsettiAnellidi
Funghi

3
gs © 2001-2009 ver 0.2 Bioluminescenza - 5 -
gs © 2001-2009 ver 0.2 Bioluminescenza - 6 -
Bioluminescenza
• Gli scopi della bioluminescenza sono molteplici:–Comunicazione–Difesa–Attacco– Illuminazione –Mimetismo–…

4
gs © 2001-2009 ver 0.2 Bioluminescenza - 7 -
Bioluminescenza
• L’emissione della luce da parte di un organismo può essere dovuta alla bioluminescenza propria dell’organismo stesso (lucciole, batteri o protozoi ecc.) attraverso un meccanismo molecolare che coinvolge delle reazioni redox o a causa della presenza di batteri simbionti.
gs © 2001-2009 ver 0.2 Bioluminescenza - 8 -
Batteri e protozoi
DINOFLAGELLATI
BATTERI
Luciferina (un pigmento biliare) Luciferasi O2
Luciferina (aldeide) Luciferasi FMNH2 e O2
FATTORI ESSENZIALI
470
478-505
λ (nm)
Vibrio fischeri e V.harveyi; P. phophoreum
Gonyaulux e
Pyrocystis fusiformis
Organismo

5
gs © 2001-2009 ver 0.2 Bioluminescenza - 9 -
Batteri e protozoi
DINOFLAGELLATI
BATTERI
Luciferina (un pigmento biliare) Luciferasi O2
Luciferina (aldeide) Luciferasi FMNH2 e O2
FATTORI ESSENZIALI
470
478-505
λ (nm)
Vibrio fischeri e V.harveyi; P. phophoreum
Gonyaulux e
Pyrocystis fusiformis
Organismo
gs © 2001-2009 ver 0.2 Bioluminescenza - 10 -
Celenterati
Antozoi
509Luciferina (celenterazina) e O2
Renilla, Ptilosarcus
485Fotoproteina e Ca2+Mnemiopsis e Beroe
Ctenofori
Idrozoi Meduse, Idroidi
Fotoproteine Ca2+proteina fluorescente verde
FATTORI ESSENZIALI
508
λ (nm)
Aequorea, Obelia
Organismo

6
gs © 2001-2009 ver 0.2 Bioluminescenza - 11 -
Celenterati
Antozoi
509Luciferina (celenterazina) e O2
Renilla, Ptilosarcus
485Fotoproteina e Ca2+Mnemiopsis e Beroe
Ctenofori
Idrozoi Meduse, Idroidi
Fotoproteine Ca2+proteina fluorescente verde
FATTORI ESSENZIALI
508
λ (nm)
Aequorea, Obelia
Organismo
gs © 2001-2009 ver 0.2 Bioluminescenza - 12 -
Celenterati
Antozoi
509Luciferina (celenterazina) e O2
Renilla, Ptilosarcus
485Fotoproteina e Ca2+Mnemiopsis e Beroe
Ctenofori
Idrozoi Meduse, Idroidi
Fotoproteine Ca2+proteina fluorescente verde
FATTORI ESSENZIALI
508
λ (nm)
Aequorea, Obelia
Organismo

7
gs © 2001-2009 ver 0.2 Bioluminescenza - 13 -
Celenterati
Antozoi
509Luciferina (celenterazina) e O2
Renilla, Ptilosarcus
485Fotoproteina e Ca2+Mnemiopsis e Beroe
Ctenofori
Idrozoi Meduse, Idroidi
Fotoproteine Ca2+proteina fluorescente verde
FATTORI ESSENZIALI
508
λ (nm)
Aequorea, Obelia
Organismo
gs © 2001-2009 ver 0.2 Bioluminescenza - 14 -
Molluschi
Cefalopodi
Bivalvi
Gasteropodi
Luciferina (celenterina) Luciferasi e O2
Encleoteuthis, Chiroteuthis e Sepiolina
490Luciferina (una proteina) luciferasi(o Fe2+ e O2)
Pholas
Luciferina Luciferasi proteina porpora e O2
FATTORI ESSENZIALI
535
λ (nm)
Latia
Organismo

8
gs © 2001-2009 ver 0.2 Bioluminescenza - 15 -
gs © 2001-2009 ver 0.2 Bioluminescenza - 16 -
Molluschi
Cefalopodi
Bivalvi
Gasteropodi
Luciferina (celenterina) Luciferasi e O2
Encleoteuthis, Chiroteuthis e Sepiolina
490Luciferina (una proteina) luciferasi(o Fe2+ e O2)
Pholas
Luciferina Luciferasi proteina porpora e O2
FATTORI ESSENZIALI
535
l (nm)
Latia
Organismo

9
gs © 2001-2009 ver 0.2 Bioluminescenza - 17 -
Molluschi
Cefalopodi
Bivalvi
Gasteropodi
Luciferina (celenterina) Luciferasi e O2
Encleoteuthis, Chiroteuthis e Sepiolina
490Luciferina (una proteina) luciferasi(o Fe2+ e O2)
Pholas
Luciferina Luciferasi proteina porpora e O2
FATTORI ESSENZIALI
535
l (nm)
Latia
Organismo
gs © 2001-2009 ver 0.2 Bioluminescenza - 18 -
Molluschi
Cefalopodi
Bivalvi
Gasteropodi
Luciferina (celenterina) Luciferasi e O2
Encleoteuthis, Chiroteuthis e Sepiolina
490Luciferina (una proteina) luciferasi(o Fe2+ e O2)
Pholas
Luciferina Luciferasi proteina porpora e O2
FATTORI ESSENZIALI
535
λ (nm)
Latia
Organismo

10
gs © 2001-2009 ver 0.2 Bioluminescenza - 19 -
Crostacei
Gamberetti eufausidi
Gamberetti decapodi
Ostracodi
476Fotoproteinasostanza fluorescente
Eufhausiae Meganyctiphanis
462Luciferina (celenterazina) Luciferasi e O2
OplophorusSergestes
Luciferina (cipridinaluciferina) e O2
FATTORI ESSENZIALI
465
λ (nm)
Cypridina
Organismo
gs © 2001-2009 ver 0.2 Bioluminescenza - 20 -
Crostacei
Gamberetti Eufausidi
Gamberetti decapodi
Ostracodi
476Fotoproteinasostanza fluorescente
Eufhausiae Meganyctiphanis
462Luciferina (celenterazina) Luciferasi e O2
OplophorusSergestes
Luciferina (cipridinaluciferina) e O2
FATTORI ESSENZIALI
465
λ (nm)
Cypridina
Organismo

11
gs © 2001-2009 ver 0.2 Bioluminescenza - 21 -
Crostacei
Gamberetti Eufausidi
Gamberetti decapodi
Ostracodi
476Fotoproteinasostanza fluorescente
Eufhausiae Meganyctiphanis
462Luciferina (celenterazina) Luciferasi e O2
OplophorusSergestes
Luciferina (cipridinaluciferina) e O2
FATTORI ESSENZIALI
465
λ (nm)
Cypridina
Organismo
gs © 2001-2009 ver 0.2 Bioluminescenza - 22 -
Crostacei
Gamberetti Eufausidi
Gamberetti decapodi
Ostracodi
476Fotoproteinasostanza fluorescente
Eufhausiae Meganyctiphanis
462Luciferina (celenterazina) Luciferasi e O2
OplophorusSergestes
Luciferina (cipridinaluciferina) e O2
FATTORI ESSENZIALI
465
λ (nm)
Cypridina
Organismo

12
gs © 2001-2009 ver 0.2 Bioluminescenza - 23 -
Teleostei
459ApogonApogonidi
459Luciferina (cypridinaluciferina) Luciferasi e O2
PorichthysBatracoidi
Pemperidi
Mictofidi
Stomiatoidei
459Parapriacanthus
Diaphus
Luciferina (celenterina) Luciferasi e O2
FATTORI ESSENZIALI
459
λ (nm)
Argyropelecus e Yarrella
Organismo
gs © 2001-2009 ver 0.2 Bioluminescenza - 24 -
Teleostei
459ApogonApogonidi
459Luciferina (cypridinaluciferina) Luciferasi e O2
PorichthysBatracoidi
Pemperidi
Mictofidi
Stomiatoidei
459Parapriacanthus
Diaphus
Luciferina (celenterina) Luciferasi e O2
FATTORI ESSENZIALI
459
λ (nm)
Argyropelecus e Yarrella
Organismo

13
gs © 2001-2009 ver 0.2 Bioluminescenza - 25 -
Teleostei
459ApogonApogonidi
459Luciferina (cypridinaluciferina) Luciferasi e O2
PorichthysBatracoidi
Pemperidi
Mictofidi
Stomiatoidei
459Parapriacanthus
Diaphus
Luciferina (celenterina) Luciferasi e O2
FATTORI ESSENZIALI
459
λ (nm)
Argyropelecus e Yarrella
Organismo
gs © 2001-2009 ver 0.2 Bioluminescenza - 26 -
Teleostei
459ApogonApogonidi
459Luciferina (cypridinaluciferina) Luciferasi e O2
PorichthysBatracoidi
Pemperidi
Mictofidi
Stomiatoidei
459Parapriacanthus
Diaphus
Luciferina (celenterina) Luciferasi e O2
FATTORI ESSENZIALI
459
λ (nm)
Argyropelecus e Yarrella
Organismo

14
gs © 2001-2009 ver 0.2 Bioluminescenza - 27 -
Teleostei
459ApogonApogonidi
459Luciferina (cypridinaluciferina) Luciferasi e O2
PorichthysBatracoidi
Pemperidi
Mictofidi
Stomiatoidei
459Parapriacanthus
Diaphus
Luciferina (celenterina) Luciferasi e O2
FATTORI ESSENZIALI
459
λ (nm)
Argyropelecus e Yarrella
Organismo
gs © 2001-2009 ver 0.2 Bioluminescenza - 28 -
Teleostei
459ApogonApogonidi
459Luciferina (cypridinaluciferina) Luciferasi e O2
PorichthysBatracoidi
Pemperidi
Mictofidi
Stomiatoidei
459Parapriacanthus
Diaphus
Luciferina (celenterina) Luciferasi e O2
FATTORI ESSENZIALI
459
λ (nm)
Argyropelecus e Yarrella
Organismo

15
gs © 2001-2009 ver 0.2 Bioluminescenza - 29 -
Batteri simbionti• Il batterio marino Vibrio fischeri in
natura esiste allo stato plantonico o come simbionte di pesci e seppie luminescenti
• Colonizza organi specializzati nell’animale ospite (Euprymna scolopes, Anomalops katoptron, Photoblepharon) che usa la luminescenza di V. fischericome esca per le prese o come camuffamento dalla luce lunare,
• La sorgente di luce sono i batteri che allo stato libero non emettono luce.
• Il meccanismo con il quale V. fischeriregola la propria bioluminescenza èlegato alla presenza di un segnale chimico di consenso via N-acil-L-omoserina lattone (AHL).
gs © 2001-2009 ver 0.2 Bioluminescenza - 30 -
Batteri simbionti• Il batterio marino Vibrio fischeri in
natura esiste allo stato plantonico o come simbionte di pesci e seppie luminescenti
• Colonizza organi specializzati nell’animale ospite (Euprymna scolopes, Anomalops katoptron, Photoblepharon) che usa la luminescenza di V. fischericome esca per le prese o come camuffamento dalla luce lunare,
• La sorgente di luce sono i batteri che allo stato libero non emettono luce.
• Il meccanismo con il quale V. fischeriregola la propria bioluminescenza èlegato alla presenza di un segnale chimico di consenso via N-acil-L-omoserina lattone (AHL).
Euprymna scolopes

16
gs © 2001-2009 ver 0.2 Bioluminescenza - 31 -
Batteri simbionti• Il batterio marino Vibrio fischeri in
natura esiste allo stato plantonico o come simbionte di pesci e seppie luminescenti
• Colonizza organi specializzati nell’animale ospite (Euprymna scolopes, Anomalops katoptron, Photoblepharon) che usa la luminescenza di V. fischericome esca per le prese o come camuffamento dalla luce lunare,
• La sorgente di luce sono i batteri che allo stato libero non emettono luce.
• Il meccanismo con il quale V. fischeriregola la propria bioluminescenza èlegato alla presenza di un segnale chimico di consenso via N-acil-L-omoserina lattone (AHL).
Anomalops katoptron
gs © 2001-2009 ver 0.2 Bioluminescenza - 32 -
Batteri simbionti• Il batterio marino Vibrio fischeri in
natura esiste allo stato plantonico o come simbionte di pesci e seppie luminescenti
• Colonizza organi specializzati nell’animale ospite (Euprymna scolopes, Anomalops katoptron, Photoblepharon) che usa la luminescenza di V. fischericome esca per le prese o come camuffamento dalla luce lunare,
• La sorgente di luce sono i batteri che allo stato libero non emettono luce.
• Il meccanismo con il quale V. fischeriregola la propria bioluminescenza èlegato alla presenza di un segnale chimico di consenso via N-acil-L-omoserina lattone (AHL).
Photoblepharon

17
gs © 2001-2009 ver 0.2 Bioluminescenza - 33 -
Photoblepharon
• Possiede un sistema per aprire e chiudere l’organo fotoforo.
gs © 2001-2009 ver 0.2 Bioluminescenza - 34 -
N-acil-L-omoserina lattone (AHL)

18
gs © 2001-2009 ver 0.2 Bioluminescenza - 35 -
Batteri simbionti• Il segnale di consenso è legato alla sintesi e all’accumulo
del segnale chimico di consenso (3-osso-C6-HSL, AHL).• In V. fischeri, il segnale è sintetizzato dalla proteina LuxI e
raccolto dalla proteina LuxR. • Quando V. fischeri è a bassa densità il segnale chimico è
basso.• Quando la densità cresce (nei organi fotofori) il segnale si
accumula ed interagisce con LuxR.• Il complesso LuxR/AHL si lega alla regione del DNA
chiamata “lux box” causando l’attivazione del gene per la sintesi delle proteina che provocano la bioluminescenza,
• Inoltre il complesso LuxR/AHL provoca la biosintesi di AHL (via LuxI) la cui sintesi viene quindi autoindotta
• Allo stato plantonico i batteri sono “bui” mentre nei fotofori siano luminosi.
gs © 2001-2009 ver 0.2 Bioluminescenza - 36 -
Diversi tipi di luminescenza
corrente elettrica in gas ionizzato o semiconduttoreElettroluminescenza
materiale radioattivo incorporato nel fosforo
Radioluminescenza
reazione chimicaChemiluminescenza
reazione enzimaticaBioluminescenza
temperatura e radioattivitàTermoluminescenza
rottura di cristalliTriboluminescenza
onde sonore in liquidiSonoluminescenza
assorbimento di luceFotoluminescenza
CAUSATIPO

19
gs © 2001-2009 ver 0.2 Bioluminescenza - 37 -
Le transizioni elettroniche(Diagramma di Jablonsky)
S0
S1
T1
Assorbimento
Fluorescenza
Fosforescenza
Intersystem crossing
Conversione interna
Energia
Stato fondamentale
Stato di singoletto eccitato
Stato di tripletto
gs © 2001-2009 ver 0.2 Bioluminescenza - 38 -
Disattivazione dello stato eccitato
S0
S1
Energia
Dissipazionetermica
Emissione(Fluorescenza)
Trasferimento di energia
Fotossidazione
Allo stato eccitatodi un’altra molecola
Reazioneredox

20
gs © 2001-2009 ver 0.2 Bioluminescenza - 39 -
Chemiluminescenza• La luminescenza che viene emessa nel corso di una
reazione chimica ha a che fare in genere con un’ossidazione. L’ossidazione può avvenire a causa di acqua ossigenata e dell’ossigeno atmosferico come nel Luminolo
NH
NH
O
O
NH2
O
O
O
O
NH2
N
N
O
O
NH2
O
O
O
O
NH2
2O-O2
*
hν
gs © 2001-2009 ver 0.2 Bioluminescenza - 40 -
Bioluminescenza
• Luminescenza prodotta da reazione catalizzate da enzimi.
• Fotoproteine.

21
gs © 2001-2009 ver 0.2 Bioluminescenza - 41 -
Luciferasi• Le luciferasi appartengono ad una classe di enzimi redox
che hanno affinità diverse per le diverse luciferine:– EC 1.13.12.5
• Renilla-luciferina 2-monoossigenasi; Renilla-type luciferasi; Aequorina; Obelina; Luciferase (Renilla luciferina)
– EC 1.13.12.6• Cipridina-luciferina 2-monoossigenasi; Cipridina-typeluciferasi; Luciferasi (Cipridina luciferina); Cipridina luciferasi
– EC 1.13.12.7• Fotinus-luciferina 4-monoossigenasi (ATP-asi); Luciferasi delle lucciole; Photinus pyralis luciferasi
– EC 1.13.12.8• Watasenia-luciferina 2-monoossigenasi; Watasenia-typeluciferasi
– EC 1.13.12.13• Oploforus-luciferin 2-monoossigenasi; Oploforus luciferasi
– EC 1.14.99.21• Latia-luciferina monoossigenasi (demetilante); Luciferasi(Latia luciferina)
gs © 2001-2009 ver 0.2 Bioluminescenza - 42 -
Gruppi prostetici di fotoproteine• Responsabili
dell'emissione di luce sono, generalmente, proteine che permettono l'ossidazione di un gruppo prostetico.
• I gruppi prostetici sono diversi tra i vari organismi, anche se vengono spesso chiamati con lo stesso nome di luciferina.
N
SS
N
OH
HCOOH
N
SS
N
OH
O
D(-) Luciferina
Ossiluciferina

22
gs © 2001-2009 ver 0.2 Bioluminescenza - 43 -
Gruppi prostetici di fotoproteine• In mare
NN
NH
R'
RO
NH
NH
NHNH CH3
OH
CH3
OO
O
O
O O
CH3
CH2
CH3
NN
NH
O
CH3
NH
CH3
NH
NH2
NH
CH3 CH3
CH3
CH3
O O
H
Cipridina Luciferina
(Vargulina)
Celenterazine
-H-OH-SO4H
-
Luciferinadei dinoflagellati
-OH-SO4H
-
Luciferina
di Latia (Latiidae)
gs © 2001-2009 ver 0.2 Bioluminescenza - 44 -
Gruppi prostetici di fotoproteine• In mare
NN
NH
R'
RO
NH
NH
NHNH CH3
OH
CH3
OO
O
O
O O
CH3
CH2
CH3
NN
NH
O
CH3
NH
CH3
NH
NH2
NH
CH3 CH3
CH3
CH3
O O
H
Cipridina Luciferina
(Vargulina)
Celenterazine
-H-OH-SO4H
-
Luciferinadei dinoflagellati
-OH-SO4H
-
Luciferina
di Latia (Latiidae)

23
gs © 2001-2009 ver 0.2 Bioluminescenza - 45 -
Celenterazine
NN
NH
R'
RO
-OH
-OSO3H
-OH
R’
Oplophorus gracilorostris
Watasenia scintellans
Renilla kolikeri
Origine
-OH
-OSO3H
-H
R
gs © 2001-2009 ver 0.2 Bioluminescenza - 46 -
Bioluminescenza • In alcune specie l’ossidazione èprovocata da una LUCIFERASI ATP dipendente con rilascio di AMP e ossiluciferina.
AMP
LH2
E
AMPL*
EEAMP
L
S
N
S
N
HH
H
COOH
OH
O
PO
P
O
OO
OO
S
N N
H
H
O
OH
O2
hνννν
E
ATP + LH2
Mg++
PP
CO2
AMP + L
D(-)luciferina
Ossiluciferina+ O2 + ATP
+ CO2 + AMP + + hν (562 nm)

24
gs © 2001-2009 ver 0.2 Bioluminescenza - 47 -
Sistema luciferina-luciferasi
N
SS
N
OH
HCOOH N
SS
N
OH
H O
O
P
O
ONO
N
N
OHOHN
NH2
O
N
SS
N
OH
H O
O
P
O
ONO
N
N
OHOHN
NH2
O
O
O
N
SS
N
OH
H
O
O
O
ATP PPi
O2
Mg2+
Luciferina
Luciferasi(EC 1.13.12.7)
Luciferasi
Luciferina-Adenilato
Perossiluciferina-Adenilato
AMP
Perossido cilclico
LuciferasiLuciferasie-
gs © 2001-2009 ver 0.2 Bioluminescenza - 48 -
Sistema luciferina-luciferasi
N
SS
N
OH
O
N
SS
N
OH
H
O
O
O
N
SS
N
OH
O
N
SS
N
OH
O
CO2
Ossiluciferina
Luciferasi
Stabilizzazione dello stato eccitato per risonanza
Perossido cilclico
Luciferasi
libera
*
Luciferasi
*
Luciferasi
Decarbossilazione
hν

25
gs © 2001-2009 ver 0.2 Bioluminescenza - 49 -
Bioluminescenza • Il processo
ossidativo viene catalizzato da LUCIFERASI che hanno una LUCIFERINA come gruppo prostetico e che utilizzano come sorgenti di elettroni una deviazione della catena respiratoria
gs © 2001-2009 ver 0.2 Bioluminescenza - 50 -
Bioluminescenza • In alcune specie l’ossidazione èprovocata da una LUCIFERASI ATP dipendente con rilascio di AMP e ossiluciferina.
AMP
Ossiluciferina

26
gs © 2001-2009 ver 0.2 Bioluminescenza - 51 -
Bioluminescenza • In alcune specie l’ossidazione èprovocata da una LUCIFERASI ATP dipendente con rilascio di AMP e ossiluciferina.
AMP
Ossiluciferina
gs © 2001-2009 ver 0.2 Bioluminescenza - 52 -
Sistema luciferina-luciferasi
• È alimentato da elettroni provenienti dalla normale catena respiratoria batterica, per mezzo della quale una frazione di e- viene trasferita dal substrato all'ossigeno.
N
SS
N
OH
HCOOH N
SS
N
OH
O
D(-) Luciferina Ossiluciferina
O2ATP
AMP + PPi
e-
CO2 hν
FMNH2FMNH
FMN
NADH
NAD+

27
gs © 2001-2009 ver 0.2 Bioluminescenza - 53 -
Celenterazina
• Un altro gruppo prostetico molto importante nel fenomeno della bioluminescenza di animali marini è la celenterazina che emette luce a seguito di un'ossidazione Ca2+dipendente.
• La reazione è catalizzata da un enzima come la obelina (Obelia geniculata) o acqueorina (Aequorea aequorea e A. victoria)
gs © 2001-2009 ver 0.2 Bioluminescenza - 54 -
Celenterazina
NN
NH
OH
O
R
APO
NN
N
OH
O
R
OO CH3APO
NH
O
N
N
OH
R
APO
O2
CO2
hν486 nm
3Ca2+

28
gs © 2001-2009 ver 0.2 Bioluminescenza - 55 -
Bioluminescenza da celenterazina
• In alcuni animali mari la bioluminescenza èdovuta ad una ossidazione Ca2+dipendente della celenterazina.
• La reazione ècatalizzata da enzimi come la obelina (da Obeliageniculata) o acqueorina (da Aequorea aequoreae A. victoria).
gs © 2001-2009 ver 0.2 Bioluminescenza - 56 -
Bioluminescenza da celenterazina
• In alcuni animali mari la bioluminescenza èdovuta ad una ossidazione Ca2+dipendente della celenterazina.
• La reazione ècatalizzata da enzimi come la obelina (da Obeliageniculata) o acqueorina (da Aequorea aequoreae A. victoria).

29
gs © 2001-2009 ver 0.2 Bioluminescenza - 57 -
Bioluminescenza da celenterazina
• In alcuni animali mari la bioluminescenza èdovuta ad una ossidazione Ca2+dipendente della celenterazina.
• La reazione ècatalizzata da enzimi come la obelina (da Obeliageniculata) o acqueorina (da Aequorea aequoreae A. victoria).
gs © 2001-2009 ver 0.2 Bioluminescenza - 58 -
Bioluminescenza da celenterazina
• In alcuni animali mari la bioluminescenza èdovuta ad una ossidazione Ca2+dipendente della celenterazina.
• La reazione ècatalizzata da enzimi come la obelina (da Obeliageniculata) o acqueorina (da Aequorea aequoreae A. victoria).

30
gs © 2001-2009 ver 0.2 Bioluminescenza - 59 -
Colori diversi…
• Per Renilla kolikeri, Aequorea aequoreae A. victoria ed altri organismi, vi è una ulteriore complicazione al fenomeno della bioluminescenza.
• Possiedono, oltre ad un proprio sistema luciferina/luciferasi che genera luce blu, anche una proteina fluorescente che permettere loro di emettere luce nel verde
• La Green Fluorescent Protein (GFP).
gs © 2001-2009 ver 0.2 Bioluminescenza - 60 -
GFP• La medusa A.
victoria presenta una curiosa luminescenza verde dovuta ad una proteina con un peculiare gruppo prostetico.
• Tale gruppo prostetico NON emette luce per fenomeni ossidativi ma “cambia” il colore della bioluminescenza.

31
gs © 2001-2009 ver 0.2 Bioluminescenza - 61 -
GFP• La Green
Fluorescent Proteinha una struttura “beta can” fatta di 11 β-strands
• Contiene al suo interno una cavitàidrofobica con una catena di tre AA (Ser-Tyr-Gly) modificati per formare un fluoroforocaratteristico
gs © 2001-2009 ver 0.2 Bioluminescenza - 62 -
GFP
• Il fluoroforo vero e proprio è formato dall’anello fenolico della Tyr66 connesso con un ponte metilenicoall’imidazolo formato dalla Ser 65 e Gly67
NH OCH3
CH3
ON
N
O
NH
ONHOH
NH2
OOH
O NH2
O

32
gs © 2001-2009 ver 0.2 Bioluminescenza - 63 -
GFP• Il fluoroforo deriva da una modificazione postraduzionale
della proteina.
NNH
O
NH
O
NH
OH
*
OH H
*
O
NN
O
*
*OH
NN
O
NH
O
NH
OH
*
OH
*
NN
O
NH
O
NH
OH
*
OH
*
Ser65
Tyr66 Gly67
Ciclizzazione
Ossidazione
O2
gs © 2001-2009 ver 0.2 Bioluminescenza - 64 -
Fotofisica della GFP
• La capacita della GFP di emettere luce verde deriva dalla proprietà della tirosina di avere diversi pKa di dissociazione dell’OH fenolico se allo stato fondamenta o allo stato eccitato:
OH
*
O
*
OH
*
O
*
+ H+
+ H+
Statofondamentale
Statoeccitato
hν
* *
pKa = 10
pK*a = 5

33
gs © 2001-2009 ver 0.2 Bioluminescenza - 65 -
Fotofisica della GFP• Inoltre, nella GFP la forma deprotonata della Tyr66 è
stabilizzata da interazioni con altri residui:– Legame idrogeno tra OH fenolico e His 148 e Thr203, – Legame idrogeno tra ossigeno carbonilico e Gln94 e Arg96– Legame idrogeno tra OH di Ser65 con Glu222
*
OCH3
N
NH
*
O O
*
*
NH
O
H
NN
O
*O
NH
NH2+
NH
*
H
NH O
H
*H
Gln94
Arg96
Glu222His148
Thr203
FORMA PROTONATA
gs © 2001-2009 ver 0.2 Bioluminescenza - 66 -
Fotofisica della GFP• Inoltre, nella GFP la forma deprotonata della Tyr66 è
stabilizzata da interazioni con altri residui:– Legame idrogeno tra OH fenolico e His 148 e Thr203, – Legame idrogeno tra ossigeno carbonilico e Gln94 e Arg96– Legame idrogeno tra OH di Ser65 con Glu222
*
OCH3 H
N
NH
*
O O
*
*
NH
O
H
NN
O
*O
NH
NH2+
NH
*
H
NH O
H
*H
*
OCH3 H
N
NH
*
O O
*
*
NH
O
H
NN
O
*O
NH
NH2+
NH
*
H
NH O
H
*H
-
Gln94
Arg96
Glu222His148
Thr203
Gln94
Arg96
Glu222His148
Thr203
Tyr66 Tyr66

34
gs © 2001-2009 ver 0.2 Bioluminescenza - 67 -
Fotofisica della GFP
*
O O
*
CH3
OH
O *
NH NH
NH2+
NH
*
NN
O
OO
*
NH
*
OHN
N
*
H
*
O O
N
N
*
*
CH3
OH
O *
NH NH
NH2+
NH
*
NN
O
OO
*
NH
*
O
H
H
*
O O
N
N
*
*
CH3
OH
O *
NH NH
NH2+
NH
*
NN
O
OO
*
NH
*
O
H
H
-
Tyr66
Arg96
Gln94
Glu222
His148
Thr203
Tyr66
Arg96
Gln94
Glu222
His148
Thr203Tyr66
Arg96
Gln94
Glu222
His148
Thr203
H+H+
gs © 2001-2009 ver 0.2 Bioluminescenza - 68 -
Fotofisica della GFP• Inoltre, nella GFP la forma deprotonata della Tyr66 è
stabilizzata da interazioni con altri residui:– Legame idrogeno tra OH fenolico e His 148 e Thr203, – Legame idrogeno tra ossigeno carbonilico e Gln94 e Arg96– Legame idrogeno tra OH di Ser65 con Glu222
Glu222
His148
Thr203
Gln94
Arg96

35
gs © 2001-2009 ver 0.2 Bioluminescenza - 69 -
Fotofisica della GFP
His148Glu222
Gln94
Thr203
Arg96
gs © 2001-2009 ver 0.2 Bioluminescenza - 70 -
Fotofisica della GFP
• La capacità di stabilizzare la forma TyrO-
rende il fluoroforo della GFP eccitabile a 470 nm (blu) dove emette il sistema luciferina luciferasi della A. victoria con emissione verde (509-540nm).

36
gs © 2001-2009 ver 0.2 Bioluminescenza - 71 -
“Il mare appariva come illuminato da sotto la superficie dell’acqua. La luminescenza disegnava sul mare un grande ovale al cui centro sembrava bruciare un falò che andava gradatamente attenuandosi verso le estremità. –Può essere un agglomerato di piccoli animali marini fosforescenti.- osservò un ufficiale…”
(Giulio Verne – 20000 leghe sotto i mari)
gs © 2001-2009 ver 0.2 Bioluminescenza - 72 -
Referenze sul WEB• Siti interessanti per il fenomeno della bioluminescenza:
– The Bioluminescence Web PageLatz Laboratory of Scripps Institution ofOceanography
– Bioluminescence Studies Research and Resources, Department of Biochemistry & Molecular Biology. The University of Georgia
– Harbor Branch Oceanographic
• Interessanti immagini di organismi marini bioluminescenti sono reperibili a: – Zooplankton in the Open Ocean– Harbor Branch Oceanographic– www.biobay.com
• Le coordinate delle strutture proteiche sono reperibili presso la Protein Data Bank
• Dettagliate e più estensive trattazioni sulla struttura della GFP e sulla sua fotofisica sono rintracciabili a:– Structure and Function of Green Fluorescent Protein– Unraveling the mysteries of the Green Fluorescent Protein

37
gs © 2001-2009 ver 0.2 Bioluminescenza - 73 -
Referenze generali sul WEB• Vie metaboliche
– KEGG: http://www.genome.ad.jp/kegg/• Degradazione degli xenobiotici:
http://www.genome.ad.jp/kegg/pathway/map/map01196.html• Struttura delle proteine:
– Protein data bank (Brookhaven): http://www.rcsb.org/pdb/– Hexpasy
• Expert Protein Analysis System: http://us.expasy.org/sprot/• Prosite (protein families and domains): http://www.expasy.org/prosite/• Enzyme (Enzyme nomenclature database):
http://www.expasy.org/enzyme/– Scop (famiglie strutturali): http://scop.berkeley.edu/
• Enzimi: – Nomenclatura - IUBMB: http://www.chem.qmw.ac.uk/iubmb/– Proprietà - Brenda: http://www.brenda.uni-koeln.de/– Expasy (Enzyme nomenclature database): http://www.expasy.org/enzyme/
• Database di biocatalisi e biodegradazione: http://umbbd.ahc.umn.edu/• Citocromo P450: http://www.icgeb.org/~p450srv/• Metallotioneine: http://www.unizh.ch/~mtpage/MT.html• Tossicità degli xenobiotici: Agency for Toxic Substances and Disease Registry
http://www.atsdr.cdc.gov
Crediti e autorizzazioni all’utilizzo
• Questo ed altro materiale può essere reperito a partire da:http://www.ambra.unibo.it/giorgio.sartor/
• Il materiale di questa presentazione è di libero uso per didattica e ricerca e può essere usato senza limitazione, purché venga riconosciuto l’autore usando questa frase:
Materiale ottenuto dal Prof. Giorgio Sartor
Università di Bologna a Ravenna
Giorgio Sartor - [email protected]