Biologia Mole Cola Re Degli Eucarioti I
Transcript of Biologia Mole Cola Re Degli Eucarioti I

Biologia Molecolare degliBiologia Molecolare degli EucariotiEucarioti
Dott.ssa Renata TisiV piano U4
Tel. 02 6448 3522

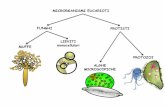
L’organismo modello più semplice per le cellule eucarioti: il lievito
Il li i è i i i ll l• Il lievito è un microrganismo unicellulare appartenenteal regno dei Funghi.Gli i ti (li iti) iù t di ti i i ti• Gli ascomiceti (lieviti) più studiati e impiegatiindustrialmente appartengono al genereSaccharomyces (nella produzione di bevande alcolicheSaccharomyces (nella produzione di bevande alcoliche,lievito da pane, autolisati ed estratti, alcuni enzimi) e algenere Candida (utilizzato come fonte di proteine).g ( p )
• I lieviti sono molto diffusi in natura e facilmenteisolabili in laboratorio per la loro capacità di darecolonie caratteristiche su terreni colturali che possonoessere resi selettivi


I lieviti preferiscono terreni colturali ricchi in zuccheri ehanno spesso esigenze per alcune vitamine e aminoacidi.p g pPer quanto riguarda il fabbisogno di ossigeno, il lorocomportamento varia da specie a specie: i saccaromiceti,ad esempio sono tutti anaerobi facoltativiad esempio, sono tutti anaerobi facoltativi.
temperatura ottimale di crescita è tra i 20 e i 30°CpH ottimale intorno a 4,5‐5,5. Le dimensioni sono moltop , ,variabili e vanno da pochi μ a 20‐30μ di lunghezza. Tutte lecellule sono fornite di una parete
Oggetto di studio e organismo modello è SaccharomycesOggetto di studio e organismo modello è Saccharomycescerevisiae, il cui genoma è stato sequenziato per intero nel 1996.

Il lievito Saccharomyces cerevisiae è una specie eucarioticamonocellulare, facile da manipolare e rapida nella crescita. Inoltre, è uni i i i t GRAS (G ll R i d A S f )microrganismo riconosciuto come GRAS (Generally Recognized As Safe).
E’ un lievito fermentativo: anche in presenza di ossigeno, la quasi totalitàdel glucosio nel terreno viene consumata attraverso la fermentazione ‐>ff tt C bt i d l i l i i è i d tt leffetto Crabtree o repressione da glucosio, e la respirazione è indotta soloa livelli molto bassi dello zucchero.
In particolare, concentrazioni diglucosio superiori a 0,25 g/Loperano una repressione reversibilesulla biosintesi del citocromo A.

Shift diauxicoShift diauxico
Fase esponenzialeFase di lag

In campo biotecnologico ed alimentare vengono richieste grandi quantitàdi proteine alcune delle quali sono difficilmente reperibili in natura: graziedi proteine, alcune delle quali sono difficilmente reperibili in natura: graziealla tecnologia del DNA ricombinante è stato possibile introdurre del DNAeterologo in alcuni organismi, appositamente selezionati, per ottenere lasintesi di prodotti eterologhisintesi di prodotti eterologhi.S. cerevisiae è capace di attuare la maggior parte delle modificazioni post‐traduzionali necessarie per la produzione di proteine di mammiferibiologicamente attive Presenta comunque alcuni svantaggi: infatti la resabiologicamente attive. Presenta comunque alcuni svantaggi: infatti, la resadel prodotto è solo dell’ 1‐5% rispetto al totale e le proteine secrete,inoltre, subiscono spesso una iperglicosilazione, causa di eventualidiminuzioni dell’attività biologica.g
• Es. produzione di rennina(componente attiva del caglio; van der Berg etal 1990)al. 1990)
• produzione di β‐galattosidasi ovina (Rocha et al. 1996)• di albumina serica umana (Fleer et al. 1991a)• di interleuchina 1β umana (Fleer et al. 1991b)• Di glucoamilasi fungina (Bui et al. 1997)• di xilanasi termostabile batterica (Walsh e Berquist 1997)di xilanasi termostabile batterica (Walsh e Berquist, 1997)


Lievito come modelloLievito come modello
di i i i l i i di li i iVengono studiati principalmente tre tipi di lieviti• Saccharomyces cerevisiae: budding yeast.Importante commercialmente nella fermentazione alcolica e nella panificazione.p
• Schizosaccharomyces pombe: fission yeast.Principalmente usato in ricerca originariamentePrincipalmente usato in ricerca, originariamente identificato nella birra africana.
• Candida albicans: fungo patogenico associato a• Candida albicans: fungo patogenico associato apazienti immunocompromessi.

Lievito gemmante e lievito a fissioneLievito gemmante e lievito a fissione
gemma
cellula madre cellula figlia
vacuolo
g
vacuolo
nucleonucleoparete cellulare setto di separazione

Ciclo vitale del lievito gemmanteCiclo vitale del lievito gemmante


Lo stato diploide protegge dalle mutazioniLo stato diploide protegge dalle mutazioni
ll l l d h l d• Le cellule aploidi hanno una singola copia di ogni gene. Mutazioniinattivanti in geni essenziali sono perciò letali.
• Nelle cellule diploidi si può invece avere una situazione di eterozigosi perp p g pcui le mutazioni inattivanti in geni essenziali non sono letali e possonoessere ottenute e propagate.
• La segregazione degli alleli durante la• La segregazione degli alleli durante la
sporulazione mette poi in evidenza il
fenotipo terminale associato allap
letalità.

Analisi delle tetradi

S. cerevisiae e gli studi di funzioneg
cloning by functional complementationcloning by functional complementation

Cloning by functional complementation

• Per clonare geni eterologhi di mammifero in S. cerevisiaeoccorre eliminare gli introni che sono rari in lievito Si preparaoccorre eliminare gli introni, che sono rari in lievito. Si prepara una banca di espressione di cDNA.

Librerie di mutanti in cellule aploidi
Intere collezioni (LIBRERIE) di mutanti possono essere generate tramite classici metodi di mutagenesi:
• agenti chimici
• radiazioniad a o
I mutanti possono poi essere isolati tramite screening di crescita intramite screening di crescita in condizioni restrittive

I mutanti possono essere incrociati per mappare i geni e studiarne le relazioni genetiche
• mediante analisi delle tetradi (prodotto delle meiosi) possiamo determinare se i due geni sono linked e calcolare ladue geni sono linked e calcolare la distanza genetica
• dall’analisi del fenotipo dei doppidall analisi del fenotipo dei doppimutanti si possono stabilire le relazionigenetiche tra i geni mutati (effettoadditivo dei difetti, soppressione difenotipi etc.)

Ex. mutants impaired in growth on galactose as a carbon sourcep g g


Il lievito come modello: il ciclo cellulare
START

La via di Ras e della PKA
Ras2p Ras2p
Cdc25p,Sdc25p
p
GDP
p
GTPIra1,2p
Clonaggio di GEFs da altri organismiorganismi

Le proteine Ras in cellule di mammifero

Studio delle cascate MAP chinasiche: la i iconiugazione

Via di trasduzione del segnaleVia di trasduzione del segnale
Identificazione di mutanti nellaStudi biochimici sulle attività e le
Identificazione di mutanti nella coniugazioneDissezione genetica del pathway
interazioni dei prodotti genici

Specificità del segnale e proteine scaffoldSpecificità del segnale e proteine scaffold
s

AgingAgingBy following the mother‐celly gpedigree microscopically over manygenerations, changes in phenotypeare observed, including cellenlargement and sterility. Thisprocess leads to a loss of divisionpotential after about 20 divisionsyielding a senescent mother cellthat has a blebby, wrinkledappearance

Aging
• Saggi di longevità:
• longevità replicativa: una cellulanewborn viene fatta replicare sunewborn viene fatta replicare susupporto solido, e le figlie vengonorimosse per micromanipolazione; ilnumero di figlie prodotte è unanumero di figlie prodotte è unamisura della RLS (replicative lifespan)
• longevità cronologica: le cellulevengono mantenute in uno stato diquiescenza attraverso carenza dinutrienti;Periodicamente, delle aliquote vengono testate per la capacità di riprendere agerminare tramite deposizione su supporto solido Il tempo necessario per perdere talegerminare tramite deposizione su supporto solido. Il tempo necessario per perdere talecapacità è la chronological life span (CLS).
Questo modello ha fornito informazioni sul coinvolgimento della via di trasduzione delgsegnale di Ras, dei ROS (radicali liberi derivanti dall’ossigeno), della calorie restriction, dellaformazione di ERCs (rDNA extra‐cromosomale) nel fenomeno dell’invecchiamento cellulare.

Sono mostrati gli effetti di due diversi disaccoppiantidiversi disaccoppianti mitocondriali:
DNP dinitrofenolo =>DNP dinitrofenolo => diminuisce i livelli di ROS
CCCP => carbonyl cyanide 3‐chlorophenylidrazone, provoca un accumulo di ROS

Restrizione calorica in lievito
Kaeberlein, Nature 464, 513‐519

TOR controls the growth of proliferating yeast, fly and mammalian cells in response to nutrients
complex 2 (TORC2) contains Tor2 but not Tor1 and controls polarity of the actincytoskeleton via the Rho1/Pkc1/MAPK cell integrity cascade

Nervous yeast: modeling neurotoxic cell deathreview on TIBS Volume 35, Issue 3, March 2010, Pages 135‐144review on TIBS Volume 35, Issue 3, March 2010, Pages 135 144
L’espressione di proteineLespressione di proteineneurotossiche può portare a diverse forme di stress cellulare, quali l’attivazionecellulare, quali l attivazionedella risposta allo shock termico, stress mitocondriale e dell’ER, o anche stress ossidativo (via ROS) ( )e disfunzioni vacuolari e dell’autofagia. Tali stress possono attivare diversipathway di morte cellulare checoinvolgono Yca1p, Aif1p, e ilcitocromo c.

• Gli oligomeri β‐amiloidi extracellulari causano tossicità in lievito. La proteina β‐amiloide intracellulare è stabilesolo se fusa ad un tag di grandi dimensioni, come la GFP, e induce una risposta di heat shock.• l’overespressione di α‐sinucleina in lievito inibisce il traffico vescicolare tra ER e Golgi, che può essere ovviatodall’overespressione della piccola GTPasi, Ypt1p. L’apoptosi è legata a stress ossidativo (via ROS), notevole dannomitocondriale e stress dell’ER a causa del difetto di trafficking. Yca1p contribuisce alla morte cellulare solo durante lafase di crescita esponenziale.• le proteine poliQ formano oligomeri chaperone‐ e prione‐dipendenti e aggregati simili a fibrille, causando danni aimitocondri e all’ ER, provocando stress ossidativo e morte cellulare. Gli aggregati oligomerici possono esseredetossificati dal trasporto in zone di raccolta perinucleari e perivacuolari attraverso la proteina di controllo qualitàdell’ER Cdc48p.• la fosforilazione di tau umana espressa in lievito dipende da Pho85p e Mds1p, gli omologhi in lievito delle chinasi ditau umane. Tau forma filamenti in lievito, ma ha un effetto trascurabile sulla sopravvivenza.• l’espressione di TDP‐43 umana in lievito causa aggregazioni nucleari e citoplasmatiche, rallentamenti della crescitae maggior mortalità. I meccanismi molecolari di tali effetti rimangono elusivi.

Evaluation of growth, survival and cell death upon expression of neurotoxic proteins. The flow‐chart illustrates the multiple experimental
h h l f dapproaches to the analysis of neurotoxicity and neurotoxic cell death and cell stress in yeast.

Biologia Molecolare del lievitogMetodi di trasformazione del lievito:
t f i di PROTOPLASTI• trasformazione di PROTOPLASTI
• trasformazione con LITIO ACETATO
• ELETTROPORAZIONE

Il plasmide 2 micronp
S cerevisiae e molti altri lieviti presentano naturalmente delleS. cerevisiae e molti altri lieviti presentano naturalmente dellemolecole di DNA extra‐cromosomali. Il DNA esogeno può esserequindi mantenuto facilmente in questa forma.
Il plasmide 2μ è una molecola di DNA circolare di 6.3 kb,extracromosomale, che si trova nel nucleo della maggior partedei ceppi di Saccharomyces cerevisiae.
• Non dà apparentemente nessun vantaggio selettivo
• Viene mantenuto in un numero di copie da 50 a 100 pergenoma aploide
’ d l l l• E’ rivestito di nucleosomi come il genoma e viene replicato unavolta per ciclo a partire da un’origine di replicazione autonoma(ARS)(ARS)

L’alto numero di copie del plasmide 2u pone due problemi:
• come si possono accumulare tante copie della molecola se lal l l lsua replicazione viene iniziata una sola volta per ogni ciclo
cellulare?
di t ib it l i t d fi li ll• come vengono distribuite le copie tra madre e figlia alladivisione cellulare?


The 2u circle has two copies of a 599 bp inverted repeat sequence (called "flip" sites) and encodes a site‐directed recombinase called FLP (the "flip" protein) that promotes recombination between these repeats. Recombination between the flip sequences inverts the adjacent regions of the plasmid as shown in the figure below.the plasmid as shown in the figure below.
"flip" sitesp
"flip" sites

Replicazione di 2 micron: rolling circleReplicazione di 2 micron: rolling circle

A model for the role of the cohesin complex in the segregation of the 2 micron plasmid.Mehta S et al. J Cell Biol 2002;158:625-637
Cohesin‐mediated pairing of replicated Cohesin‐mediated pairing pairing of replicated plasmid clusters occurs during the S phase as does the pairing of sister chromatids. The plasmid clusters are tethered to
p gand unpairing are common to the chromosomes and the plasmid clusters as in
A. The plasmids are partitioned howeverclusters are tethered to
sister chromatids, likely by a cohesin‐independent mechanism.
partitioned, however, without physical
attachment to the chromosomes.
© 2002 Rockefeller University Press

I vettori per lievito: origini di replicazionePer i vettori extra‐cromosomali è necessaria una sequenza autonoma di
replicazione che può essere:
• ARS (Autonomous Replication Sequence) isolata dal genoma consente la• ARS (Autonomous Replication Sequence) isolata dal genoma, consente lareplicazione;
• CEN: contiene un centromero cromosomico, si trova in vettori YCp (yeastcentromeric plasmid);
• 2 μ: una parte della sequenza del plasmide naturale 2 micron; in vettoriYEp (yeast episomal plasmid);YEp (yeast episomal plasmid);
I plasmidi origin‐less: YIp (yeast integrating plasmid) non possono replicarsiautonomamente, devono integrarsi nel cromosoma di lievito.


Si tratta sempre di vettori shuttle, cioè in grado di essere mantenuti in lievito magrado di essere mantenuti in lievito ma anche di essere replicati in E. coli, ospite di elezione per la maggior parte delle
d d ll t l i d l DNAprocedure della tecnologia del DNA ricombinante.

Trasformazione integrativaTrasformazione integrativa

P t lt diPer mantenere un alto numero di copie:
Leu2d => gene parzialmenteLeu2d => gene parzialmente difettoso, induce selezione dei cloni con alto numero di copie del plasmide per cui il prodottodel plasmide per cui il prodotto di Leu2d riesce a complementare la funzione e permette la crescita

Marcatori dominanti
Agente tossico Marcatore
( )Amino glucoside 3’‐f f f ( cassetta di espressione derivanteG418 (Geneticina) fosfotransferasi (Gene
KanR)
cassetta di espressione derivante da un trasposone di E. coli
Igromicina BIgromicina B
Fosfotransferasi (Gene hph)
Aminoglicosidi: inibiscono la sintesi proteica causando errori di riconoscimento dei codoni
Zeocina/fleomicina/
bleomicina
Gene Zeo/ble da Streptoalloteichus
hindustanus
Glicopeptidi: intercalano il DNA causandone la frammentazione
hindustanus
Nurseotricina Gene nat

ADE1 e ADE2
I geni ADE1 and ADE2 codificano per la fosforibosilamino‐imidazolo‐I geni ADE1 and ADE2 codificano per la fosforibosilamino imidazolosuccinocarbozamide sintetasi e la fosforibosilamino‐imidazolo‐carbossilasi,rispettivamente, due enzimi della via biosintetica dell’ adenina.
I t ti d 1 d 2 d i t t t d i tI mutanti ade1 e ade2 producono un pigmento rosso apparentemente derivatodalla polimerizzazione dell’intermedio fosforibosilamino‐imidazolo (AIR).
Quindi ceppi ade2 sono rossi, mentre gli ade3 e il doppio mutante ade2 ade3sono bianchi.

ADE3 encodes for a cytoplasmictrifunctional enzyme C1‐tetrahydrofolate synthase

La pigmentazione rossa dei mutanti ade1 e ade2, e la reversione della colorazione daparte della mutazione ade3, sono stati utilizzati per la creazione di diversi test geneticidi stabilità genomica, plasmidica e nello studio dei sistemi di ripartizione deicromosomi.
ade2 ade3 background
YRp withYRp withADE3 andessential gene
in the essential gene
red‐whitesectoredsectoredcolonies

Two steps gene‐replacementTwo steps gene replacement

Gene disruption and single‐step gene replacement
• The YFG1+ gene is disrupted by transforming the strain with a linear fragment containing a URA3
selectable marker flanked by homologous sequences. h h l l d b hThe chromosomal segment is replaced by this URA3
containing fragment after integration by homologous recombination at the two ends.
• The URA3marker introduced in the YFG1 locus, can be excised if URA3 is also flanked by direct repeats of
DNA, preferably not originating from yeast. Homologous recombinants, selection on FOA
medium, lack the URA3marker and retain a single copy of the repeated DNA.
• Single‐step gene replacement of mutant alleles, such g p g pas yfg1‐1, can be carried out by first replacing the YFG1 gene by URA3, transforming the strain with
linear fragment encompassing the yfg1‐1mutation, and selecting transformants on FOA medium, in which
URA3 is replaced by yfg1‐1.

Gene replacementGene replacement


Plasmid shuffling
The chromosomal copy of YFG1 is replaced bythe yfg1‐Δ deletion, but the Yfg+ phenotype ismaintained by the YCp plasmid containingYFG1 and URA3. The strain is transformed witha mutagenized LEU2 plasmid having the YFG1
R i f 1 t tigene. Recessive yfg1‐x mutations aremanifested by selecting for strains on FOAmedium. The strain will not grow on FOAmedium if YFG1 is an essential gene and if themedium if YFG1 is an essential gene and if theyfg1‐xmutation is not functional.

Come si può rivelare che tipo di integrazione è avvenuta?
• Southern blot
• PCR su DNA genomico
Southern blot: richiamo

singolo crossing‐overg g
ricombinazione non omologagene replacement

Verifica delezione genica tramite PCR su DNA genomicoVerifica delezione genica tramite PCR su DNA genomico

Yeast Artificial Chromosome (YAC)Yeast Artificial Chromosome (YAC)

Clonare in uno YAC

Il ceppo ospite è mutato in ade2 e can1 (arginina permeasi: trasporta anche la canavanina, che è tossica)
In presenza del tRNA soppressore SUP4 le mutazioni non senso dei due marcatori sono soppresse
il ceppo diventa ade+ e ritorna sensibile alla canavanina

I promotori di lievito Allison, Fondamenti di biologia molecolare, ed. Zanichelli

I promotori di lievito: studio delle UAS
URS upstream regulatory sequenceUAS upstream activating sequence

Promotori di eucarioti superioriPromotori di eucarioti superioriEnhancers : vi si legano specificiEnhancers : vi si legano specifici fattori di trascrizione, che a loro volta interagiscono con i fattori di t i i ti ldi trascrizione posti sul promotore e con la RNA polimerasi.
• agiscono a distanze molto ampie•agiscono in entrambi gli orientamenti•effetti indipendenti dalla pposizione (a monte o a valle del gene)

Promotori costitutivi
Promotore Caratteristiche
PGK (phospholyceratek )
Efficiente/2‐5% glucosiokinase)
Efficiente/2 5% glucosio
GAPDH (glyceraldehyde‐3‐phosphate dehydrogenase)
Efficiente/2‐5% glucosio
ENO1, ENO2 Efficiente/2‐5% glucosio
TPI (triose phosphateff / l
TPI (triose phosphateisomerase)
Efficiente/2‐5% glucosio
ADH1 (alcoholdehydrogenase I)
moderat. efficiente/2‐5% glucosiodehydrogenase I) glucosio

Promotori inducibiliPromotori inducibili
Promotore Caratteristiche
GAL1 GAL10 (galactokinaseGAL1‐GAL10 (galactokinase‐UDP‐Gal epimerase)
Efficiente/galattosio
PHO5 (acid phosphatase)Moderat. Efficiente/ inibito
d Pi( p p )
da Pi
CUP1 (metallothionein) Moderat. Efficiente/Cu
CYC1 (Cyt. c1) Reprimibile da glucosio
ADH2 (alcohold h d II)
Efficiente/0.1‐0.2% glucosiodehydrogenase II)
/ g

GAL1 Promoter
Promotori inducibili
The most commonly‐used regulated promoter for yeast studies is PGAL1.
There are two regulatory proteins, Gal4p and Gal80p, which effect the transcription ofthe indicated structural genes:the indicated structural genes:
Gal80 binds to Gal4, repressing its transactivation activity.
Gal3p appears to be required for the production of the intracellular inducer fromgalactose.
The UAS of the divergently transcribed GAL1 and GAL10 is contained within a 365‐bpg y pfragment, denoted UASGAL. The sequence responsible for glucose‐dependent repressionis URSGLC.

GAL1 Promoter
The most commonly‐used regulated promoter for yeast studies is PGAL1.
galactoseglucose repression
ginduction

PGAL1 is sufficient for maximal galactose induction and thorough glucose repression. PGAL1can rapidly induce the expression of downstream fused‐genes over 1000‐fold after thecan rapidly induce the expression of downstream fused genes over 1000 fold after theaddition of galactose to cells growing in media with a nonfermentable carbon source.Furthermore, PGAL1 can be turned off by the addition of glucose to the galactose containingmedium.medium.
Proprietà di Gal4p
• due domini indipendenti per legame al DNA (BD) e transattivazione (AD)
• il legame di Gal4 al DNA è inibito dalla presenza di glucosio nel mezzo
Mig1

Experiments to map the DNA‐binding and activation domain of yeast GAL4 proteinactivation domain of yeast GAL4 protein

Esperimento di scambio dei domini BD e AD tra Gal4 e LexA
Il repressore LexA inibisce i genicoinvolti nella la risposta SOS, checodificano per le DNA polimerasi
l d lpreposte al riparo del DNA.In caso di danni al DNA RecA si legaa LexA sui suoi siti di legame sulDNA d l’ t t li iDNA causandone l’autoproteolisi.

Repressione da glucosioInduzione da galattosio

M. Rubio‐Texeira / FEMS Yeast Research 5 (2005) 1115–1128

M. Rubio‐Texeira / FEMS Yeast Research 5 (2005) 1115–1128


Aploinsufficienza: screening genetico basato sul dosaggio genico nei diploidi eterozigoti

(herpes simplex virus)
es.
Nota bene: il dominio attivatore di VP16 rende il tTA un attivatore trascrizionale, nonostante il dominio DNA binding provenga da un repressorenonostante il dominio DNA binding provenga da un repressore


The pTet‐Off Advanced vector constitutively expresses the tetracycline‐controlled transcriptional transactivator, Tet‐Off Advanced . This engineered protein consists of the E. coli TetR protein fused to three i i l "F” t ti ti d i d i dminimal "F”‐type activation domains derived
from the herpes simplex virus VP16 protein (Baron et al., 1997, Triezenberg et al., 1988).
The PTight composite promoter was originally developed as the Ptet‐14 promoter in the laboratory of Dr. H. Bujard and consists of a modified Tet‐Responsive Element (TREmod) containing 7 direct repeats of the tet operator sequence, tetO, which is joined to a minimal CMV promoter (PCMV) ( l h l )(Clontechniques, April 2003).

Tet‐Off: The TRE is located upstream of the minimal immediate early promoter ofcytomegalovirus (PminCMV) which is silent in the absence of activation tTA binds thecytomegalovirus (PminCMV), which is silent in the absence of activation. tTA binds theTRE—and thereby activates transcription of Gene X—in the absence of Tc or Dox.

Cinetica di induzione/repressione
HeLa S3 Tet‐Off trasfettate con plasmide recante coding sequence per Bcl‐2 sotto promotore TRE.Repressione con Tetraciclina alle concentrazioni indicate. Western blot.
Cellule CHO esprimenti tTA sono state trasfettate con un costrutto per l’espressione della luciferasi sotto promotore TRE‐CMV.

Tet‐Off→ switched off in the presence of the Tet/Dox
T OTet‐On→ switched on in the presence of the Tet/Dox

Tet‐On: The reverse Tet repressor (rTetR) was created by four amino acid changes thatreverse the protein’s response to Dox. As a result of these changes, the rTetR domain ofp p g ,rtTA binds the TRE and activates transcription in the presence of Dox.


• Regolazione molto efficace: in presenza di Dox, tTA non si lega al motivo TRE dando una trascrizione basale molto bassaTRE dando una trascrizione basale molto bassa
• alta specificità: il legame del tTA è specifico per il promotore tetO
• Dox e Tet non hanno alcun effetto noto su eucarioti
• Alta e rapida responsività : anche di migliaia di volte in 30 min, ma non sempre riproducibile in ogni sistema cellulare
• Nota: il sistema Tet‐Off risponde a Dox e Tet, il Tet‐On solo a Dox
• I sistemi a regolazione positiva sono in genere più facili da mantenere completamente spenti e da accendere rapidamentecompletamente spenti e da accendere rapidamente
• I sistemi Tet sono utili per la creazione di topi transgenici reversibilmente inducibili

Il sistema Tet‐Off funzionaIl sistema Tet Off funziona perfettamente anche nei mammiferi, sia in cellule in coltura che in organismi in toto, e può essere reso tessuto‐specifico.toto, e può essere reso tessuto specifico.
Watson et al., DNA ricombinante 2°, Zanichelli

Yeast as a toolkitSistemi dei due ibridi
Powerful methods, denoted two‐hybrid systems,have been designed for screening and investigatingi t ti t i B f th f thinteracting proteins. Because of the ease of theassay, exploratory two‐hybrid screens are usuallythe first method of choice when information ofinteracting proteins are desiredinteracting proteins are desired.
(A) Normally, the Gal4 transcription activator bindst DNA t th G l4 bi di it d ti tto DNA at the Gal4p binding sites and activatestranscription of the lacZ reporter gene. (B) A hybridof the Gal4 activation domain with the Yfg2 proteindoes not activate transcription because it does notdoes not activate transcription because it does notlocalize at the Gal4 binding site. (C) A hybrid of theGal4 DNA‐binding site domain with the Yfg1 proteindoes not activate transcription of the reporter genedoes not activate transcription of the reporter genebecause of the lack of the transcriptional activationdomain. (D) Protein‐protein interaction betweenYfg1p and Yfg2p reconstitutes Gal4p function andYfg1p and Yfg2p reconstitutes Gal4p function andactivates transcription of the reporter gene.

The Escherichia coli lacZ gene which encodes β‐galactosidase is the most commonly used reporter
Gene reporter LacZThe Escherichia coli lacZ gene, which encodes β galactosidase, is the most commonly used reporterwith yeast and other systems; its activity can be assayed semiquantitatively on plates by thedifferential staining of colonies using X‐gal (5‐bromo‐4‐chloro‐3‐indolyl‐b‐D‐galactoside) and fullyquantitatively by enzyme assay of liquid cultures on ONPG or CPRG.

Two‐hybrid: protein interactions (not membrane proteins)
sistema LexA

human PKC η C1 domainlega DAG
domain shuffling
lega DAG
rat Raf C1 domainlega smalllega smallG proteins

3AT is a competitive inhibitor of the His3 enzyme and only baits that exhibit good activation of HIS3 reporter transcription will be able to grow in the presence of this compound.

Criticità del sistema del doppio ibrido
• FALSI NEGATIVI : non rileva interazioni deboli o transienti; nonsi può utilizzare con proteine che non possano localizzare nelcomparto nucleare;comparto nucleare;
• FALSI POSITIVI : un ibrido BD con una proteina acida puòfunzionare da transattivatore anche da solo; la trascrizione basaled l t HIS3 è l t t tdel reporter HIS3 non è completamente assente => occorreazzerarla con l’inibitore 3‐AM
L’analisi richiede SEMPRE una validazione con altrometodo (es. coimmunoprecipitazione)

Coimmunoprecipitation

One‐hybrid: BD or AD functions

Three‐hybrid: adaptor proteins/chemicals…

Membrane recruitmentassay: Ras‐rescue cyto‐TRAP assay
La fusione Bait‐hSos può sopprimere la p ppletalità condizionale di un mutante cdc25 ts solo se reclutata in membrana
inducibleexpressionexpression

Yeast‐GFP : localization database